Автор: Лукашов В.В.
Теги: вирусология общая генетика генетика эволюция
ISBN: 978-5-9963-0114-0
Год: 2009
Текст
В. В. Лукашов
МОЛЕКУЛЯРНАЯ
ЭВОЛЮЦИЯ
И ФИЛОГЕНЕТИЧЕСКИЙ
АНАЛИЗ
ИЗДАТЕЛЬСТВО
В. В. Лукашов
МОЛЕКУЛЯРНАЯ
ЭВОЛЮЦИЯ
И ФИЛОГЕНЕТИЧЕСКИЙ
АНАЛИЗ
Учебное пособие
Допущено
Учебно-методическим объединением
по классическому университетскому образованию
в качестве учебного пособия
для студентов, обучающихся по специальности
«биоинженерия» и «биоинформатика»
Москва
БИНОМ. Лаборатория знаний
2009
УДК 578(075.3)
ББК 28.04
Л84
Лукашов В. В.
Л84 Молекулярная эволюция и филогенетический анализ /
В. В. Лукашов. — М. : БИНОМ. Лаборатория знаний,
2009. — 256 с. : ил.
ISBN 978-5-9963-0114-0
В книге обсуждаются принципы эволюционного анализа генетиче-
ской информации. Автор знакомит читателей с теоретическими основами
и практическими подходами к решению задач молекулярной эволюции
и филогенетического анализа. Эта книга является первым отечественным
изданием, освещающим данную область знаний.
Для студентов, аспирантов и научных сотрудников биологических,
медицинских и математических специальностей.
УДК 578(075.3)
ББК 28.04
Научное издание
Лукашов Владимир Владимирович
МОЛЕКУЛЯРНАЯ ЭВОЛЮЦИЯ И ФИЛОГЕНЕТИЧЕСКИЙ
АНАЛИЗ
Ведущий редактор канд. биол. наук Л. А. Аксенова
Художник С. Инфантэ
Технический редактор Е. В. Денюкова
Корректор Л. Н. Макарова
Оригинал-макет подготовлен М. Ю. Копаницкой в пакете 1Я^Х2е
Подписано в печать 11.09.09. Формат 60x90/16.
Усл. печ. л. 16. Тираж 1000 экз. Заказ № 2238
Издательство «БИНОМ. Лаборатория знаний»
125167, Москва, проезд Аэропорта, д. 3
Телефон: (499)157-5272, e-mail: binom@Lbz.ru, http://www.Lbz.ru
Отпечатано в полном соответствии с качеством
предоставленных диапозитивов в ОАО «Издательско-
полиграфическое предприятие «Правда Севера».
163002, г. Архангельск, пр. Новгородский, 32.
Телефакс (8182) 64-14-54, тел.: (8182) 65-37-65, 65-38-78
www.ippps.ru, e-mail: zakazfa’ippps.ru
ISBN 978-5-9963-0114-0
© БИНОМ. Лаборатория знаний,
2009
Посвящаю эту книгу
памяти моего отца
ПРЕДИСЛОВИЕ
Развитие методов полимеразной цепной реакции и автоматиче-
ского определения нуклеотидных последовательностей сделало
возможным получение генетической информации в не пред-
ставимом ранее масштабе. Число нуклеотидных последователь-
ностей в международной базе генетических данных GenBank
превышает 70 миллионов, а их общая длина — 80 миллиардов
нуклеотидов (см. рисунок). Объем этой информации удваивается
в среднем каждые 18 месяцев.
Одновременное развитие компьютерных технологий привело
к тому, что анализ генетической информации, бывший ранее
темой работы относительно небольшого числа специалистов, ста-
новится повседневной задачей многих научных и практических
лабораторий.
В связи с этим, актуально написание книги, освещающей
принципы эволюционного анализа генетической информации.
90
80
80 -
70-
число нуклеотидов, в миллиардах
----число последовательностей,
в миллионах
70
60
2
ч
к
Е-1
о
ф
ч
к
>>
К
60 -
50-
40 -
30 -
20 -
50
40
зо
20
о
к
л
ч
ф
Е-1
ч
и
о
ф
ч
и
о
е
е
s
/
10 -
10
о 4
о
Рост объема информации в генетической базе данных GenBank
6
Предисловие
Эволюционный анализ генетической информации является
составной частью более общего раздела науки — биоинформати-
ки. Спектр задач, которыми занимается биоинформатика, очень
широк: к этой области науки относятся изучение происхож-
дения наследственной информации, анализ регуляции синтеза
макромолекул — нуклеиновых кислот и белков, предсказание
структуры макромолекул, изучение взаимодействий между ними
и многие другие вопросы [Ратнер с соавт., 1985; Гельфанд,
2005].
Одной из ключевых, основополагающих задач биоинформа-
тики является установление законов изменения макромолекул
в ходе их эволюции и, на основании этих данных, — установле-
ние родственных отношений между формами жизни, от которых
эти макромолекулы получены. Этими вопросами занимается
молекулярная филогенетика, возникшая на стыке традиционных
наук об эволюции, передаче наследственной информации и ма-
тематики [Грант, 1980; Иорданский, 2001].
Наша книга является первой отечественной книгой, осве-
щающей современное состояние вопросов, связанных с фило-
генетическим анализом в молекулярной эволюции. При работе
над книгой, автор ставил целью ознакомить читателя с теоре-
тическими основами и практическими подходами к решению
ключевых задач эволюционного и филогенетического анализа.
Эта книга предназначена для студентов, аспирантов и науч-
ных сотрудников биологических, медицинских и математиче-
ских специальностей, занимающихся вопросами эволюционного
и филогенетического анализа, а также медицинских сотрудни-
ков, интересующихся этими вопросами.
В гл. 1 излагаются понятия, используемые в молекулярной
эволюции, включая относящиеся к строению макромолекул,
генетическому коду, видам эволюционных событий и методам
их изучения. При этом подразумевается, что читатель знаком
с принципами организации, воспроизводства и наследования
генетической информации — строением генов и организацией
генома, процессами транскрипции и трансляции, механизмом на-
следования признаков, законами естественного отбора— из кур-
сов биологии, биохимии и генетики. Глава 2 рассказывает
о принципах выравнивания генетических последовательностей.
Эволюционные модели и методы установления эволюционных
дистанций между последовательностями описываются в гл.З.
Глава 4 посвящена принципам филогенетического анализа и ме-
тодам построения филогенетических деревьев. В гл. 5 описыва-
Предисловие 7
ется ряд специальных вопросов, возникающих при изучении
молекулярной эволюции, включая рекомбинационный анализ,
исследование нуклеотидного состава и использования кодонов,
анализ митохондриальной ДНК и молекулярных часов. Специ-
альное внимание уделено использованию генетического анализа
при изучении эпидемиологии инфекционных заболеваний — мо-
лекулярной эпидемиологии. Все эти вопросы рассмотрены с ис-
пользованием многочисленных примеров, на конкретных экспе-
риментальных данных. Наконец, гл. 6 посвящена описанию ком-
пьютерных программ, используемых в эволюционном анализе.
Написание этой книги было бы невозможным без участия мо-
их коллег. Прежде всего благодарю Эдуарда Карамова и Япа Год-
смита (bedankt, Jaap!) за многолетнюю поддержку моих научных
интересов. Серьезную помощь при написании книги оказали ее
рецензенты: Михаил Гельфанд (Институт проблем передачи ин-
формации РАН и Московский государственный университет им.
М. В. Ломоносова) и Александр Гултяев (Университет Лейдена,
Нидерланды). Александр Пастернак (Университет Амстердама,
Нидерланды) помогал в редактировании книги. Владимир Муро-
нец (НИИ физико-химической биологии им. А. Н. Белозерского,
Московский государственный университет им. М. В. Ломоносова)
и Герман Шипулин (ЦНИИ эпидемиологии Роспотребнадзора,
Москва) оказали помощь при публикации книги.
Автор будет признателен за отзывы, замечания, предложения
касательно этой книги, которые следует направлять по адресу:
Lukashov_book@mail. ги.
В заключение предисловия отметим, что
любой математический алгоритм, любая компьютерная программа,
используемая при анализе генетической информации, проанали-
зирует заложенные исследователем данные, используя выбранные
исследователем параметры, — и выдаст результаты анализа. Вопрос
о том, насколько заложенные в программу данные и выбранные
при анализе параметры правильны, соответственно — насколько ре-
зультаты анализа отражают истинные эволюционные события, —
является вопросом, на который должен ответить сам исследователь.
Среди прочих методов — и с помощью компьютерных программ.
Владимир Лукашов,
Амстердам, Нидерланды,
5 апреля 2009 г.
ENGLISH ANNOTATION
Rapid developments in the areas of PCR and automatic sequencing
have made it possible to obtain unprecedented volumes of gene-
tic information. Due to simultaneous progress in computer techno-
logies, evolutionary analysis of genetic information is becoming an
everyday task of many scientific and practical laboratories.
This book is entitled «Molecular evolution and phylogenetic
analysis» and written by Vladimir Lukashov (Academic Medical
Center, University of Amsterdam, The Netherlands and D.I.Iva-
novsky Institute of Virology, Moscow, Russia). The book is aimed at
pre- and post-graduate students as well as researchers and describes
a broad spectrum of theoretical and practical issues arising in the
evolutionary analysis.
Chapter 1 describes the principles, aims, and terminology
used in molecular evolution and phylogenetic analysis. Chapter 2
deals with sequence alignment. Evolutionary models and genetic
distances are described in Chapter 3. Chapter 4 describes phylo-
genetic tree building methods. Chapter 5 discusses a number of
specific issues arising during the evolutionary analysis, including
recombination analysis, nucleotide and amino acid composition,
codon usage, the analysis of mitochondrial DNA and molecular
clock. Specific attention is given to describing the role of
evolutionary analysis in studying the epidemiology of infectious
disease — molecular epidemiology. All these issues are discussed
using actual experimental data. Finally, Chapter 6 describes compu-
ter programmes used in the evolutionary analysis.
ЦЕЛИ, ПРИНЦИПЫ и понятия
МОЛЕКУЛЯРНОЙ эволюции
1.1. Задачи молекулярной эволюции как науки
Молекулярная эволюция как наука включает в себя два направ-
ления исследований. Ее целями являются изучение законов из-
менения наследственной информации в живых системах, вклю-
чая доклеточные и клеточные формы жизни, и изучение истории
развития жизни на Земле, установление родственных отноше-
ний между формами жизни, их генетическим материалом — фи-
логении форм жизни (phylogeny). Первое направление включает
в себя изучение частоты и других характеристик эволюционных
изменений в макромолекулах — нуклеиновых кислотах и белках,
а также механизмов и причин, определяющих эти изменения.
Вторая область исследований молекулярной эволюции, направ-
ленная на реконструкцию эволюционного процесса развития
форм жизни на Земле, установление родственных связей между
формами жизни, создание их эволюционной классификации,
называется молекулярной филогенетикой (phylogenetics). Тра-
диционно изучение пребиотической эволюции, происхождения
жизни, также понимается как задача молекулярной эволюции.
Однако механизмы пребиотической эволюции принципиально
отличаются от законов эволюции живых систем, требуют спе-
циального рассмотрения и, как правило, описываются отдельно.
1.2. Нуклеотидные последовательности
У всех живых систем, включая доклеточные и клеточные
формы жизни, наследственная, или генетическая, информация
содержится в геноме, представленном молекулами нуклеиновых
кислот. У подавляющего большинства форм жизни генетиче-
ская информация передается от поколения к поколению в виде
молекул дезоксирибонуклеиновых кислот (ДНК). Ряд вирусов
является исключением из этого правила и передает генетиче-
скую информацию в виде рибонуклеиновых кислот (РНК) —
РНК-содержащие вирусы.
Молекула нуклеиновой кислоты представляет собой линей-
ную полимерную цепочку, состоящую из мономеров — нуклеоти-
дов. В эволюционном анализе различия в химическом строении
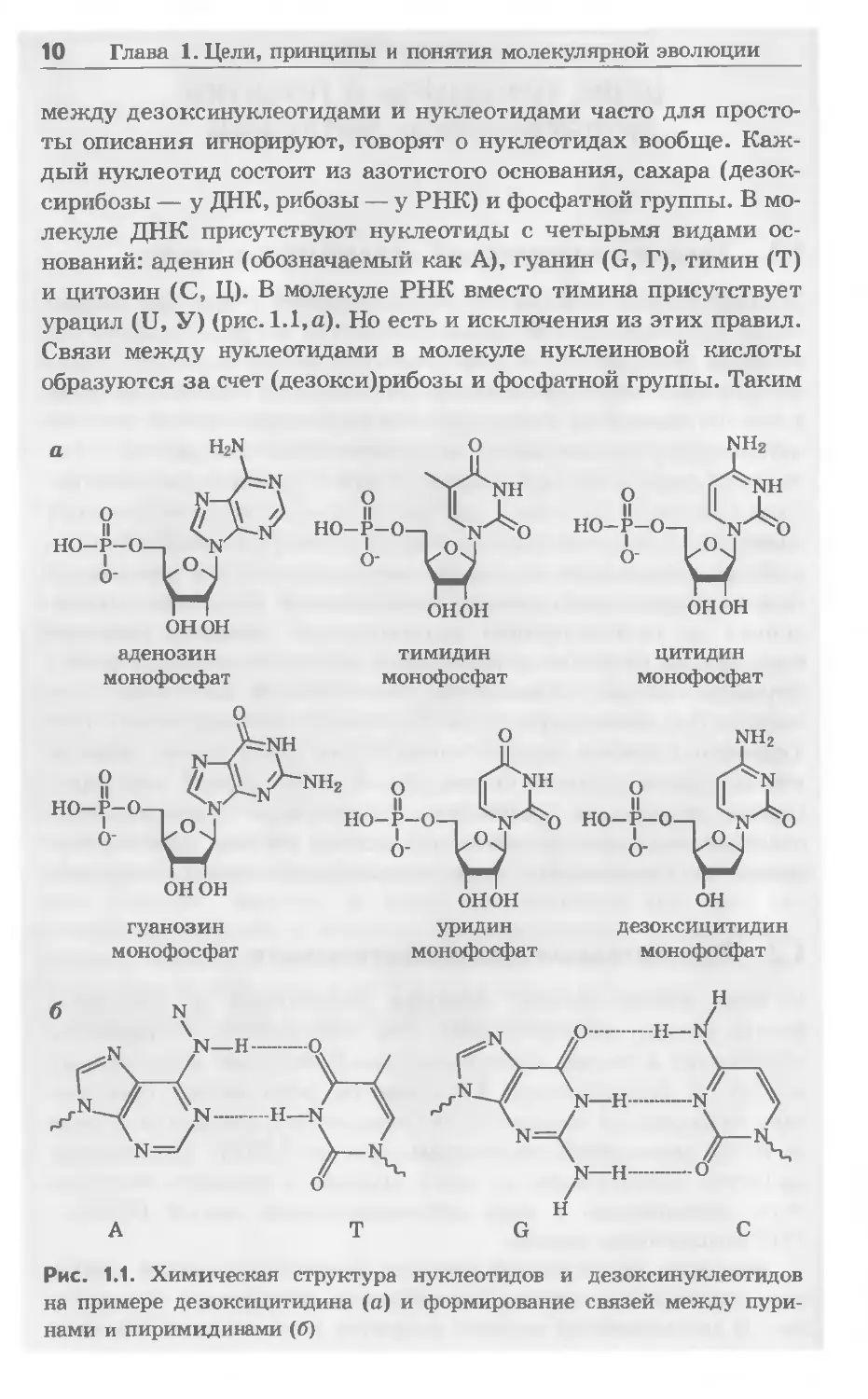
10 Глава 1. Цели, принципы и понятия молекулярной эволюции
между дезоксинуклеотидами и нуклеотидами часто для просто-
ты описания игнорируют, говорят о нуклеотидах вообще. Каж-
дый нуклеотид состоит из азотистого основания, сахара (дезок-
сирибозы — у ДНК, рибозы — у РНК) и фосфатной группы. В мо-
лекуле ДНК присутствуют нуклеотиды с четырьмя видами ос-
нований: аденин (обозначаемый как А), гуанин (G, Г), тимин (Т)
и цитозин (С, Ц). В молекуле РНК вместо тимина присутствует
урацил (U, У) (рис. 1.1, а). Но есть и исключения из этих правил.
Связи между нуклеотидами в молекуле нуклеиновой кислоты
образуются за счет (дезокси)рибозы и фосфатной группы. Таким
он он
аденозин
монофосфат
тимидин
монофосфат
цитидин
монофосфат
уридин
дезоксицитидин
гуанозин
монофосфат
монофосфат
монофосфат
Рис. 1.1. Химическая структура нуклеотидов и дезоксинуклеотидов
на примере дезоксицитидина (а) и формирование связей между пури-
нами и пиримидинами (б)
1.3. Аминокислотные последовательности 11
образом, каждая молекула нуклеиновой кислоты представляет
собой последовательность нуклеотидов, различающихся между
собой основаниями. Нуклеотидную последовательность молеку-
лы РНК часто для простоты записывают как соответствующую
ей молекулу ДНК, т.е. содержащую Т. Такой подход использован
и в этой книге. Соответственно любая молекула нуклеиновой
кислоты записывается как последовательность нуклеотидов
с различными основаниями, например — ATACGAGAAAACGCA.
Эта последовательность нуклеотидов является первичной
структурой молекулы нуклеиновой кислоты. Нуклеотиды А
и G являются пуринами, а С и Т — пиримидинами. В двойной
цепочке ДНК (вторичная структура), пурины А одной цепочки
образуют две водородные связи с пиримидинами Т второй цепоч-
ки (слабая связь), а пурины G — три связи с пиримидинами С
(термодинамически более стабильная сильная связь) (рис. 1.1,6).
В молекуле нуклеиновой кислоты различают кодирующие
и некодирующие участки. Первые кодируют белки, вторые вклю-
чают в себя, в частности, регуляторные элементы.
1.3. Аминокислотные последовательности
Основой структуры белка является полимерная цепочка, состоя-
щая из мономеров-аминокислот — полипептид, аминокислотная
последовательность. С химической точки зрения следует гово-
рить: состоящая из аминокислотных остатков, однако в совре-
менной международной литературе по молекулярной эволюции
для простоты принято использовать упрощенную терминологию.
Каждая аминокислота состоит из R-группы (боковой цепи),
к которой присоединены карбоксильная и аминная группы.
Связь между аминокислотами в полипептидной цепочке осу-
ществляется при взаимодействии аминогруппы одной аминокис-
лоты с карбоксильной группой другой аминокислоты (пептидная
связь). Существует 20 так называемых стандартных амино-
кислот (табл. 1.1). Пользуясь различными критериями, амино-
кислоты можно отнести к разным типам или классам. Так,
в зависимости от химического строения их боковых цепей выде-
ляют алифатические (с дальнейшим делением на моноаминомо-
нокарбоновые, оксимоноаминокарбоновые и т. д.), ароматические
и гетероциклические аминокислоты и иминокислоты. В моле-
кулярной эволюции наиболее существенной является класси-
фикация аминокислот, основанная на их физико-химических
свойствах: полярные или неполярные, нейтральные, кислые или
лапа x. цели, принципы и понятия молекулярной эволюции
основные аминокислоты и т. д. Для обозначения аминокислот
используются трехбуквенные или однобуквенные сокращения
(см. табл. 1.1). Любая молекула белка может быть записана как
последовательность аминокислот, например — Val-Val-Ile-Arg-
Ser-Glu-Asn-Phe-Thr-Asp, или VVIRSENFTD. Далее в нашей
книге использованы однобуквенные сокращения.
Таблица 1.1
Химическая структура, обозначение и свойства аминокислот
Аминокислота Химическая структура Обозначение Свойства
трехбук- венное однобук- венное
1 2 3 4 5
аланин (alanine) О н3« X NHa Ala A неполярная
аргинин (arginine) NH О н Arg R положительно заряженная при pH — 7
аспарагин (asparagine) я Ч я О“Ч Я А Asn N полярная, незаряженная
аспарагиновая кислота (aspartic acid) о ОН NH, Asp D отрицательно заряженная при pH — 7
валин (valine) Vai V неполярная
гистидин (histidine) о </'Ог он NH, His H положительно заряженная при pH = 7
ГЛИЦИН (glycine) О л» NH, Gly G полярная, незаряженная
глутамин (glutamine) О О NHB Gin Q полярная, незаряженная
Продолжение табл. 1.1
1 2 3 4 5
глутаминовая кислота (glutamic acid) О о но^^'^у^он NH, Glu E отрицательно заряженная при pH = 7
изолейцин (isoleucine) СН3 о Н^/ОН NH, He I неполярная
лейцин (leucine) -X™ Leu L неполярная
лизин (lysine) и / HaN^6°H Lys К положительно заряженная при pH=7
метионин (methionine) О HaC"Sx^Y^OH NH, Met M неполярная
пролин (proline) H if О Pro P неполярная
серин (serine) цгон H2N^y*° OH Ser s полярная, незаряженная
тирозин (tyrosine) Tyr Y полярная, незаряженная
треонин (threonine) он о HjC^V^OH nh2 Thr T полярная, незаряженная
триптофан (tryptophan) Trp w неполярная
фенилаланин (phenylalanine) H hn*yoh 0 Phe F неполярная
цистеин (cysteine) о HaC^^Y^OH NH2 Cys C полярная, незаряженная
1.4. Генетический код
Информация о порядке аминокислот в белковой цепочке содер-
жится в кодирующих участках ДНК/РНК. Каждая аминокис-
лота кодирована последовательностью из трех нуклеотидов —
Таблица 1.2
Универсальный генетический код
Прямая таблица 2-я позиция
Т C A G
1-я пози- ция Т ТТТ Phe/P ТТС Phe/P ТТА Leu/L TTG Leu/L TCT Ser/S TCC Ser/S TCA Ser/S TCG Ser/S TAT Tyr/Y TAC Tyr/Y TAA стоп TAG стоп TGT Cys/C TGC Cys/C TGA стоп TGG Trp/W
С СТТ Leu/L СТС Leu/L СТА Leu/L CTG Leu/L CCT Pro/P CCC Pro/P CCA Pro/P CCG Pro/P CAT His/H CAC His/H CAA Gln/Q CAG Gln/Q CGT Arg/R CGC Arg/R CGA Arg/R CGG Arg/R
А ATT Ile/I АТС Ile/I ATA Ile/I ATG Met/M ACT Thr/T ACC Thr/T АСА Thr/T ACG Thr/T AAT Asn/N AAC Asn/N AAA Lys/K AAG Lys/K AGT Ser/S AGC Ser/S AGA Arg/R AGG Arg/R
G GTT Val/V GTC Val/V GTA Val/V GTG Val/V GCT Ala/A GCC Ala/A GCA Ala/A GCG Ala A GAT Asp/D GAC Asp/D GAA Glu/E GAG Glu/E GGT Gly/G GGC Gly/G GGA Gly/G GGG Gly/G
Обратная таблица
Ala/A GCA, GCT, GCG, GCC Leu/L TTA. TTG, СТА, CTT, CTG,CTC
Arg/R AGA, AGG, CGA, CGT, CGG, CGC Lys/K AAA, AAG
Asn/N AAT, AAC Met/M ATG
Asp/D GAT, GAC Phe/F TTT,TTC
Cys/C TGT, TGC Pro/P CCA, CCT, CCG, CCC
Gln/Q CAA, CAG Ser/S AGT, AGC, TCA, TCT, TCG, TCC
Glu/E GAA, GAG Thr/T АСА, ACT, ACG, ACC
Gly/G GGA, GGT, GGG, GGC Trp/W TGG
His/H CAT, CAC Tyr/Y TAT, TAC
Ile/I ATA, ATT, АТС Val/V GTA, GTT, GTG, GTC
старт ATG СТОП TAA, TAG, TGA
триплетом, или кодоном. Соответствие между нуклеотидными
триплетами и кодируемыми ими аминокислотами, а также сиг-
налами начала и окончания синтеза полипептидной цепочки,
трансляции, называют генетическим кодом. В подавляющем
большинстве случаев генетический код в живых системах яв-
ляется одинаковым и называется универсальным, или стан-
дартным, генетическим кодом (табл. 1.2). Однако существуют
и отличия от универсального генетического кода — как правило,
небольшие, в значениях нескольких кодонов. Так, генетический
код митохондрий имеет отличия от универсального кода. При
этом митохондриальный генетический код одинаков у всех по-
звоночных, но отличается от митохондриальных генетических
кодов, например, дрожжей или дрозофил (табл. 1.3).
Поскольку кодон представляет собой последовательность
из трех нуклеотидов и существуют четыре различных нуклео-
тида, то число возможных кодонов составляет 43 = 64. При этом
им необходимо кодировать 20 различных аминокислот и сигнал
окончания трансляции, т.е. 21 сигнал (напомним, что отдельного
сигнала для начала трансляции нет, стартовый кодон кодиру-
ет аминокислоту). Соответственно конкретные аминокислоты
и сигнал окончания трансляции могут кодироваться более чем
одним кодоном. Это явление называют вырожденностью генети-
ческого кода. Из 64 кодонов в универсальном генетическом коде
61 кодирует аминокислоты (смысловые кодоны), а три — сигнал
окончания трансляции (нонсенс-, или стоп-кодоны). Различные
кодоны, кодирующие одну и ту же аминокислоту, называют
Таблица 1.3
Примеры отличий от универсального генетического кода
Кодон Значение в генетическом коде
универсальном митохонд- риальном позвоночных митохонд- риальном дрозофил митохонд- риальном дрожжей
TGA СТОП-КОДОН W W W
СТТ L L L Т
СТС L L L Т
СТА L L L т
CTG L L L т
АТА I М М I
AGA R СТОП-КОДОН S R
AGG R СТОП-КОДОН S R
ю 1лава i. цели, принципы и понятия молекулярной эволюции
синонимичными кодонами. Так, изолейцин кодируется синони-
мичными кодонами АТА, АТТ и АТС. В большинстве случаев,
синонимичные кодоны различаются между собой нуклеотидами
в третьей позиции (см. табл. 1.2).
Отметим, что две аминокислоты, метионин и триптофан,
кодируются только одним кодоном, а три аминокислоты — ар-
гинин, лейцин и серин — кодируются шестью кодонами.
1.5. Мутации
В процессе репликации нуклеиновых кислот могут происходить
ошибки. В результате дочерний геном будет отличаться от ро-
дительского генома. Ошибки, происходящие при репликации
генома, называют мутациями (mutations). У многоклеточных
организмов мутации могут происходить как в половых, так
и в соматических клетках. Соматические мутации не переда-
ются от поколения к поколению, и, таким образом, исключены
из эволюционного процесса.
Мутации могут быть классифицированы в соответствии с чис-
лом затронутых ими нуклеотидов (длиной мутации) и типами
происходящих при этом событий. Мутации могут затрагивать
только один нуклеотид — точечные мутации (point mutations) —
или несколько соседних нуклеотидов. При этом могут проис-
ходить следующие события: замена одного нуклеотида на дру-
гой— нуклеотидная замена (nucleotide substitution), вставка од-
ного или более нуклеотидов (insertion), удаление одного или
нескольких соседних нуклеотидов — делеция (deletion), поворот
участка нуклеиновой кислоты длиной минимум в два нуклео-
тида на 180 — инверсия (inversion). Частным случаем вставки
является удвоение некоего генетического участка — дупликация
(duplication).
Особым случаем мутации является считывание дочерней
молекулы нуклеиновой кислоты не с одной, а с двух и .более
родительских молекул — рекомбинация (recombination).
В кодирующих участках вставки и делеции, если их длина
не кратна трем, приводят к изменению рамки считывания поли-
пептидной цепочки. Это ведет к изменению всей последующей
аминокислотной последовательности и, как правило, потере бел-
ком своей функции, нежизнеспособности такого потомства.
В молекулярной эволюции значительное внимание уделяется
изучению нуклеотидных замен. Анализ рекомбинаций изложен
в разд. 5.1.
1.6. Нуклеотидные замены 17
1.6. Нуклеотидные замены
1.6.1. Транзиции и трансверсии
В кодирующих и некодирующих участках нуклеиновых кис-
лот нуклеотидные замены могут быть разделены на транзиции
(transitions) и трансверсии (transversions). Транзициями называ-
ют замены одного пурина на другой пурин (A—>G, G—»А) или
одного пиримидина на другой пиримидин (С —> Т, Т —> С). Транс-
версиями называют замены между пуринами и пиримидинами
(А—»Т, А—»С, G—»Т, G—»С, Т —А, Т — G, С —A, C->G).
1.6.2. Синонимичные и несинонимичные замены
В кодирующих участках вследствие вырожденности генетиче-
ского кода конкретная нуклеотидная замена может изменять
или не изменять смысл кодона.
Нуклеотидную замену, не изменяющую кодируемую ами-
нокислоту, называют синонимичной заменой (synonymous
substitution, или silent substitution — молчащая). Так, нуклеотид-
ная замена А —»Т в третьей позиции кодона АТА (АТА -+ АТТ)
не изменяет кодируемую аминокислоту (до и после замены —
изолейцин) и, таким образом, является синонимичной.
Нуклеотидную замену, изменяющую кодируемую амино-
кислоту, называют несинонимичной заменой (nonsynonymous
substitution, или non-silent substitution—немолчащая). Так, за-
мена A—»G в третьей позиции того же кодона АТА (АТА—> ATG)
является несинонимичной, поскольку кодируемая аминокислота
изменяется с изолейцина на метионин. Таким образом, про-
исходит замещение (replacement) одной аминокислоты другой
(говорят о замене нуклеотида, но о замещении аминокислоты).
Мутацию, приводящую к изменению триплета с кодиру-
ющего аминокислоту на стоп-кодон, называют нонсенс-мутацией
(nonsense mutation). Так, замена ТАС—»ТАА является нонсенс-
мутацией. Иногда нонсенс-мутации рассматривают как частный
случай несинонимичных мутаций. В таком случае несинони-
мичные замены, приводящие к изменению одной аминокислоты
на другую, но не на стоп-кодон, называют изменяющими смысл
(missense mutations). Как правило, нонсенс-мутации приводят
к потере кодируемым белком своей функции, нежизнеспособно-
сти потомства и соответственно исключаются из эволюционного
процесса.
2 — 2238
juxoo x. цели, принципы и понятия молекулярной эволюции
В любом кодоне каждый из трех нуклеотидов может быть
заменен на любой из трех других нуклеотидов. Соответственно
для каждого кодона число возможных замен составляет 3 х
3 = 9, т. е при одной нуклеотидной замене каждый кодон
может измениться на один из девяти других кодонов. Таким
образом, для всех 64 кодонов число возможных замен состав-
ляет 64 х 9 — 576. Поскольку универсальный генетический код
включает 61 смысловой кодон, то всего в смысловых кодонах
возможны 61 х 9 — 549 нуклеотидных замен. В целом боль-
шинство этих замен будут несинонимичными. Однако, поскольку
синонимичные кодоны, как правило, различаются нуклеотидами
в третьей позиции, то большинство замен в третьей позиции
будут синонимичными. Отметим, что некоторые замены в пер-
вой позиции кодона также являются синонимичными, но ни
одна замена во второй позиции кодона синонимичной не яв-
ляется. Данные по возможным синонимичным и несинонимич-
ным заменам для всех кодонов в каждой позиции приведены
в табл. 1.4.
Таблица 1.1
Возможные типы нуклеотидных замен
в кодирующих последовательностях
Тип замены Число замен %
Всего во всех кодонах 549 100
синонимичные 134 25
несинонимичные 415 75
изменяющие смысл 392 71
нонсенс 23 4
Всего в первой позиции 183 100
синонимичные 8 4
несинонимичные 175 96
изменяющие смысл 166 91
нонсенс 9 5
Всего во второй позиции 183 100
синонимичные 0 0
несинонимичные 183 100
изменяющие смысл 176 96
нонсенс 7 4
Всего в третьей позиции 183 100
синонимичные 126 69
несинонимичные 57 31
изменяющие смысл 50 27
нонсенс 7 4
1.7. Нуклеотидный и аминокислотный состав 19
Говорят и о синонимичных и несинонимичных позициях
кодона. Если все три возможные замены нуклеотида в данной
позиции не изменяют кодируемую аминокислоту, то такая по-
зиция является синонимичной. Если две из трех возможных
замен нуклеотида в данной позиции не изменяют кодируемую
аминокислоту, то такая позиция является синонимичной на две
трети,и т. д.
1.7. Нуклеотидный и аминокислотный состав,
использование кодонов
Процентное содержание различных нуклеотидов и аминокислот
в генетической последовательности называют ее нуклеотид-
ным и аминокислотным составом, или композицией (nucleotide
composition, amino acid composition). Если замена любого нуклео-
тида на любой другой нуклеотид при репликации последователь-
ности, а также последующее закрепление любой нуклеотидной
замены происходят с одинаковой вероятностью, то содержание
любого из четырех нуклеотидов в последовательности будет
приблизительно равно 25%. Однако содержание конкретных
нуклеотидов в геномах может значительно отклоняться от 25%.
В таких случаях говорят о смещениях нуклеотидного состава
(nucleotide bias) или предпочтении того или иного нуклеотида
(nucleotide preference). Скажем, если в некой последовательно-
сти содержание нуклеотидов А, Т, G и С составляет 35, 15, 22
и 28% соответственно, то говорят о преобладании нуклеотида А
за счет нуклеотида Т в составе этой последовательности. Рас-
сматривают также суммарное содержание нуклеотидов G и С —
GC-содержание (GC-content). Поскольку в двойной цепочке ДНК
нуклеотиды G и С образуют между собой три связи (разд. 1.2),
то двухцепочечная молекула ДНК с большим GC-содержанием
термодинамически более стабильна (ее температура плавления
выше).
Как уже отмечалось (разд. 1.4, см. табл. 1.2), вследствие
вырожденности генетического кода, большинство аминокис-
лот может кодироваться более чем одним кодоном. Показа-
но, что в кодирующих последовательностях могут наблюдаться
предпочтения при выборе тех или иных синонимичных кодо-
нов [Grantham et al., 1980]. Скажем, лейцин кодируется шестью
кодонами, однако в наружном мембранном белке II (отрА)
Escherichia coli для кодирования лейцина в 21 из 23 случаев
20 Глава 1. Цели, принципы и понятия молекулярной эволюции
используется лишь один кодон — CTG. При описании распреде-
ления различных синонимичных кодонов в последовательности
говорят об использовании кодонов (codon usage).
Вопросы эволюционного анализа смещения нуклеотидного
состава и использования кодонов обсуждены в разд. 5.2.
1.8. Эволюция нуклеотидной последовательности
Рассмотрим эволюцию молекулы нуклеиновой кислоты. Пусть
в момент времени 0 эта молекула длиной в 30 нуклеотидов имеет
следующую последовательность 0:
111111111122222222223
позиция 123456789012345678901234567890
последовательность 0 ATACGAGAAAACGCAATCCGCGAGAATGCC
При репликации этой молекулы могут происходить или
не происходить ошибки. Если за некий промежуток времени
(измеряемый либо единицами времени, либо числом репликаций
последовательности, т. е. числом поколений) при per ликации
этой молекулы ошибки происходить не будут, то существующая
в момент времени 1 дочерняя последовательность 1 не будет
отличаться от родительской последовательности 0. Соответствен-
но можно говорить, что за промежуток времени 0-1 рассмат-
риваемая последовательность не претерпела эволюционных из-
менений. Для наглядности эти две последовательности можно
записать одну под другой, а нуклеотиды последовательности 1,
совпадающие с таковыми в той же позиции последовательно-
сти 0, обозначить знаками тире:
111111111122222222223
123456789012345678901234567890
0 ATACGAGAAAACGCAATCCGCGAGAATGCC
1 -------------------------------
Предположим, что при дальнейшей репликации этой моле-
кулы будут происходить ошибки. Так, пусть за промежуток
времени 1-2 в рассматриваемой последовательности произошли
две нуклеотидные замены: замена С-»Ав позиции 4 и замена
С —> Т в позиции 21. В таком случае на момент времени 2 порядок
эволюционных изменений в рассматриваемой последовательно-
сти можно записать следующим образом:
«jiwicvin^nuM ыииледовательности Z1
111111111122222222223
123456789012345678901234567890
0 ATACGAGAAAACGCAATCCGCGAGAATGCC
1
2 ---A----------------T---------
Последовательности 0 и 1 не отличаются друг от друга,
являются идентичными (identical). Последовательности же 1
и 2 являются не идентичными, но похожими (similar). Они
имеют различия между собой, иначе говоря — они находятся
на определенной генетической, или эволюционной, дистанции
(genetic distance, evolutionary distance), или расстоянии, друг
от друга.
Пусть за последующий промежуток времени 2-3 в рассмат-
риваемой последовательности произошли еще три замены: в по-
зициях 4 (А—» С), 21 (Т —»А) и 30 (С —»G):
111111111122222222223
123456789012345678901234567890
0 ATACGAGAAAACGCAATCCGCGAGAATGCC
1
2 —-А-----------------Т--------
3 --------------------А--------G
При этом замена нуклеотида в позиции 30 произошла впер-
вые (первичная замена), в то время как в позициях 4 и 21
нуклеотидные замены ранее уже происходили — в этих позици-
ях за промежуток времени 2-3 произошли вторичные замены.
Таким образом, за весь рассмотренный промежуток времени 1-3
в позициях 4 и 21 произошли множественные нуклеотидные
замены (multiple hits at one site, множественные удары в одной
позиции): в позиции 4 произошли множественные замены С—»
А —»С, в позиции 21 — множественные замены С —»Т —»А.
Как видно, вторичная замена в позиции 4 привела к тому,
что нуклеотид, находящийся в этой позиции, снова совпадает
с таковым у предка, последовательности 0. Такие вторичные
замены называют обратными заменами (back substitutions), или
реверсиями (reversions).
Пусть в дальнейшем за промежуток времени 3-4 в рассмат-
риваемой последовательности произошла делеция нуклеотидов
13-15 и замена нуклеотида в позиции 29 (С—>А), а затем,
LL
1лава i. цели, принципы и понятия молекулярной эволюции
за промежуток времени 4-5 — замена нуклеотида в позиции 18
(С —♦ Т). Обозначая делении точками (иногда используют и про-
тивоположные обозначения: совпадающие нуклеотиды — точки,
делении — тире), все эти эволюционные изменения, т. е. эволю-
ционную историю рассматриваемых последовательностей, можно
записать следующим образом:
111111111122222222223
123456789012345678901234567890
0 ATACGAGAAAACGCAATCCGCGAGAATGCC
1
2 ---А-------------Т-------
3 -----------------А-------G
4 ----------...----а-----AG
5 ----------. .Т—А-------AG
Позиции, в которых в эволюционной истории происходило
множество замен, называют изменчивыми, или вариабельными
(variable), позициями. Так, в рассматриваемом случае позиции 4
и 21 — изменчивые. Позиции, в которых замены не происхо-
дили, или их было мало, называют соответственно абсолют-
но или относительно консервативными (conserved). Абсолютно
консервативные позиции называют также инвариантными. Так,
позиция 1 — абсолютно консервативная, а позиции 18, 29 и 30 —
относительно консервативные. Говорят и об изменчивых и кон-
сервативных участках последовательности. Так, в рассматривае-
мом случае участок 1-12 или 1-20 можно назвать относительно
консервативным, а участок 21-30 — относительно изменчивым.
В рассматриваемом случае мы исходили из того, что каждая
родительская последовательность дает только одну дочернюю
последовательность. Естественно, в общем случае это не так.
Каждая родительская последовательность может быть репли-
цирована несколько раз, и происходящие при каждом цикле
ее репликации ошибки могут быть различны. В таком слу-
чае дочерние последовательности одной родительской последо-
вательности будут различаться между собой. Более того, даже
если дочерние последовательности одинаковы, ошибки при их
дальнейшей репликации будут, в общем случае, различны.
Рассмотрим такой случай (рис. 1.2). Пусть в момент времени 0
молекула нуклеиновой кислоты имеет последовательность 0.
Предположим, что за каждый промежуток времени одна роди-
поколение
последовательность 0 123456789012345678
▼
3.1.1.1 (1)
---A-G-----------Т
3.1.1.2(5)
---A-G-----------С
3.1.2.1(2)
—G-------СС-. . .AGT
3.1.2.2(6)
--G----------. . ,__т
▼
3.2.1.1 (3)
__т-------------д_
3.2.1.2 (7)
__т-------------Д-
▼
3.2.2.1 (4)
-----т----------G
3.2.2.2 (8)
-----т----------G
Рис. 1.2. Эволюция нуклеотидной последовательности. Параллельные мутации в позиции 18 (А—>Т) подчеркнуты
тельская последовательность дает две дочерние последователь-
ности. Пусть за промежуток времени 0-1 последовательность 0
даст две не отличающиеся друг от друга идентичные дочерние
последовательности 1.1 и 1.2. Очевидно, что при последующей
репликации мутации, возникающие в последовательностях 1.1
и 1.2, будут, в общем случае, различными: мутации при ре-
пликации последовательности 1.1 не зависят от того, какие
мутации произошли при репликации последовательности 1.2.
Если между потомками этих последовательностей (форм жизни,
от которых они получены) не будет происходить обмена генети-
ческой информацией (отсутствие полового процесса, рекомбина-
ций и т. д., не важно, в силу каких именно причин), то даль-
нейшая эволюция последовательности 1.1 не будет зависеть
от эволюции последовательности 1.2. В таком случае говорят,
что при эволюции последовательности 0 в поколении (момент
времени) 1 произошла дивергенция между дочерними линиями
(divergence), разделение на две независимые родственные эволю-
ционные линии (evolutionary lineages), возможное начало видо-
образования. Говорят также об ответвлении (branching out) эво-
люционных линий, их почковании, разветвлении эволюционного
дерева. Отметим, что в последующем в дочерних последователь-
ностях этих линий могут происходить и одинаковые мутации:
так, в рассматриваемом примере в этих эволюционных лини-
ях независимо друг от друга произошли одинаковые мутации
в позиции 18 (А—>Т, рис. 1.2). Одинаковые мутации, которые
произошли в родственных эволюционных линиях независимо
друг от друга, называют параллельными мутациями (parallel
mutations).
Таким образом, через промежуток времени 0-3 потомство
последовательности 0 будет представлять собой восемь раз-
личающихся между собой последовательностей — генетически
гетерогенную (heterogeneous) группу последовательностей. Про-
цесс, ведущий к образованию такой гетерогенной группы, на-
зывают диверсификацией (diversification). Все рассматриваемые
на рис. 1.2 последовательности происходят от единого общего
предка — последовательности 0. Последовательности, имеющие
общее эволюционное происхождение, называют гомологичными
(homologous) (разд. 1.10). Группу таких гомологичных последова-
тельностей называют монофилетической (monophyletic) группой,
имеющей общее эволюционное происхождение, общего предка.
Поскольку задачей молекулярной филогенетики является уста-
новление эволюционной истории генов и форм жизни после их
дивергенции от общего предка, то эта область молекулярной
эволюции по определению анализирует гомологичные последо-
вательности. Выяснение вопроса, являются ли некие последова-
тельности гомологичными, а не просто похожими друг на друга
(например, в результате конвергентной эволюции, разд. 1.10),
является важной задачей эволюционного анализа.
Одним из ключевых понятий молекулярной эволюции являет-
ся понятие последнего общего предка группы последовательно-
стей (в дословном переводе с английского — наиболее недавнего
общего предка, most recent common ancestor, MRCA) и времени
его существования. Последний общий предок всех форм жизни
на Земле обозначается термином LUCA (Zast universal common
ancestor). Для всех рассматриваемых последовательностей по-
коления 3 последний общий предок существовал в поколении
(момент времени) 1, а не в поколении 0, поскольку диверсифика-
ция группы последовательностей поколения 3 произошла после
момента времени 1.
При рассмотрении представленного на рис. 1.2 примера эво-
люции нуклеотидной последовательности мы исходили из то-
го, что для поколения 3 известны все предки, родительские
последовательности, порядок диверсификации последовательно-
стей в группе, все мутации, происходившие в их эволюционной
истории. Естественно, что на практике — ни вымершие (extinct)
предки неких существующих сегодня последовательностей, ни
порядок их диверсификации, ни происходившие при эволюции
последовательностей мутации неизвестны. Собственно, задачей
молекулярной эволюции и является установление сценария со-
бытий, имевших место в эволюционной истории анализируемой
группы последовательностей, форм жизни. При этом базовый
принцип молекулярной эволюции как науки основан на положе-
нии о том, что наиболее вероятный, наиболее близкий к истин-
ному сценарий эволюции группы последовательностей — это тот
сценарий, который требует минимального числа эволюционных
событий. Безусловно, это может быть и не так, и истинным —
может быть и другой сценарий, с большим числом событий,
но в данном случае действует научный принцип, известный
как бритва, или лезвие, Оккама: без необходимости не следу-
ет утверждать многое (лат. — Pluralitas non est ponenda sine
necessitate). To, что можно объяснить посредством меньшего,
не следует выражать посредством большего. В науке под брит-
вой Оккама понимают общий принцип, утверждающий, что если
существует несколько логически непротиворечивых определе-
X JXCAUCX
. ЦЕЛИ, принципы и понятия молекулярной эволюции
ний или объяснений какого-либо явления, то верным следует
считать наиболее простое из них.
Итак, задачей молекулярной эволюции является установ-
ление эволюционной истории группы гомологичных последо-
вательностей, генов, организмов, живых систем вообще — на-
пример, эволюционной истории существующих сегодня восьми
последовательностей поколения 3, о которых имеется генети-
ческая информация (см. рис. 1.2). Запишем эту информацию,
учитывая, что предки этих последовательностей нам неизвест-
ны, и неизвестно, в каких участках каких именно современных
последовательностей имели место делеции или вставки:
1 ATCAGGGAGAATAAAGCT
2 ATGCGAGAGCCTAGT
3 ATTCGAGAGAATAAAGAA
4 ATCCGTGAGAATAAAGCG
5 ATCAGGGAGAATAAAGCC
6 ATGCGAGAGAATGCT
7 ATTCGAGAGAATAAAGAA
8 ATCCGTGAGAATAAAGCG
Первым этапом филогенетического анализа группы после-
довательностей является идентификация вставок и делеций,
имевших место в их эволюционной истории — выравнивание
последовательностей (гл. 2). Методы эволюционного анализа по-
следовательностей изложены в гл. 3-5.
1.9. Консенсусные последовательности
Для анализируемой группы последовательностей можно рас-
смотреть так называемую консенсусную последовательность,
или консенсус (consensus) — искусственную последовательность,
в каждой позиции которой находится нуклеотид, наиболее часто
встречающийся у анализируемых последовательностей. Как пра-
вило, при расчете консенсусной последовательности в нее запи-
сывают нуклеотид, присутствующий как минимум в 50% анали-
зируемых последовательностей (консенсус-50%). При этом отсут-
ствие нуклеотида в конкретной позиции (одной из) анализиру-
емых последовательностей можно принимать или не принимать
во внимание. Если в 50% последовательностей в данной позиции
присутствует один нуклеотид, а в других 50% последовательно-
стей — другой нуклеотид, то в консенсусную последовательность
принято записывать нуклеотид из первой последовательности
группы. При расчете консенсусной последовательности можно
использовать и менее, и более строгие пороговые критерии.
Так, в консенсус можно включать нуклеотиды, просто наиболее
часто встречающиеся в конкретной позиции, даже если они
присутствуют менее чем в 50% последовательностей, или наобо-
рот — минимум в 90% последовательностей, или даже в 100%
последовательностей. Если гетерогенность конкретной позиции
не отвечает заданным критериям — скажем, выбран порог в 50%,
но ни один из нуклеотидов не присутствует минимум в 50%
последовательностей — то такую позицию принято обозначать
знаком вопроса, или N, или X. Все эти правила действительны
и для аминокислотных последовательностей.
Кроме того, в консенсусной нуклеотидной последовательно-
сти можно отражать разнообразие нуклеотидов в конкретной
позиции (гетерогенность позиции, heterogeneity). Для этого ис-
пользуют коды Международного союза теоретической и при-
кладной химии ШРАС (International Union of Pure and Applied
Chemistry). Так, если в некой конкретной позиции в одних
последовательностях находится А, а у других — Т, то А/Т ге-
терогенность этой позиции в консенсусной последовательности
можно отразить как W. Коды ШРАС для обозначения нуклеоти-
дов приведены в табл. 1.5.
Для рассмотренного выше примера (пусть нам известны толь-
ко последовательности поколения 3 на рис. 1.2) консенсусные
последовательности можно записать следующим образом:
консенсус-100%
консенсус-50%
консенсус-IUPАС
1
2
3
4
5
6
7
8
111111111
123456789012345678
AT??G?GAG??T???G??
ATCCGAGAGAATAAAGC?
ATBMGDGAGMMTRVWGMN
ATCAGGGAGAATAAAGCT
ATGCGAGAGCCTAGT
ATTCGAGAGAATAAAGAA
ATCCGTGAGAATAAAGCG
ATCAGGGAGAATAAAGCC
ATGCGAGAGAATGCT
ATTCGAGAGAATAAAGAA
ATCCGTGAGAATAAAGCG
Таблица 1.5
Коды IUPAC для обозначения нуклеотидов
Код Обозначает Комплементарный нуклеотид
А А T(U)
С С G
G G С
T(U) Т (U) А
м А или С К
R А или G Y
W А или Т (U) W
S С или G S
Y С или Т (U) R
К G или Т (U) М
V А или С или G В
н А или С или Т (U) D
D А или G или Т (U) Н
В С или G или Т (U) V
X или N А или С или G или Т (U) X или N
Для наглядности индивидуальные последовательности можно
записать относительно консенсуса (скажем, 50%-го), используя
тире для обозначения одинаковых нуклеотидов:
консенсус-507,
1
2
3
4
5
6
7
8
111111111
123456789012345678
ATCCGAGAGAATAAAGC?
---A-G----------Т
--G-----CC--GT
—Т-------------ДА
-----Т----------G
------------------A-G-С
—G----------GCT
__Т------------да
----Т-----------G
Поскольку последовательности поколения 3 имеют разную
длину, то в их эволюционной истории происходили вставки
и/или делеции нуклеотидов. Однако, поскольку эволюционная
1.10. Гомологичные и сходные признаки, конвергенция 29
история поколения 3 нам (как мы договорились) неизвестна,
то неизвестно, где именно произошли вставки-делеции. Эти
вопросы устанавливаются в процессе выравнивания последова-
тельностей (гл. 2).
1.10. Гомологичные и сходные признаки,
конвергенция
В разд. 1.8 подчеркивалось, что молекулярная филогенетика
анализирует гомологичные последовательности (имеющие общее
эволюционное происхождение). Наличие у нескольких организ-
мов похожих признаков не обязательно является свидетельством
общего эволюционного происхождения этих признаков и соот-
ветственно самих организмов. Классическим примером сходного,
или подобного, признака, имеющего независимое эволюционное
происхождение, является наличие крыльев у насекомых, птиц
и летучих мышей. Наличие таких сходных признаков называют
гомоплазией (homoplasy), а сами признаки — не гомологичными,
но аналогичными (analogous). Причиной гомоплазии являет-
ся конвергентная эволюция (convergent evolution, сходящаяся),
конвергенция — эволюционный процесс, при котором у нерод-
ственных организмов в процессе адаптации к одним и тем же
природным условиям независимо образуется сходный признак.
Наличие того или иного нуклеотида (аминокислоты) в опре-
деленной позиции последовательности также является призна-
Таблица 1.6
Дивергентная, параллельная и конвергентная эволюция
Эволюция
дивергентная параллельная конвергентная
Родительские последовательности 1 2 1 2 1 2
Нуклеотиды в позиции i у роди- тельских последо- вательностей А Л 1 1 к А Л Т А
Нуклеотиды в той же позиции У дочерних после- довательностей А Т С Т Т A G A G
JV
ijitLBti i. цели, принципы и понятия молекулярной эволюции
ком. Вышеизложенные рассуждения важны для понимания того,
что схожесть между некими генетическими последовательностя-
ми, наличие у них одинаковых нуклеотидов (аминокислот) в со-
ответствующих позициях не является достаточным основанием
для заключения о гомологичности этих позиций, последователь-
ностей вообще. Наличие одного и того же нуклеотида (амино-
кислоты) может быть и результатом конвергентной эволюции.
Примеры дивергенции, конвергенции и параллельной эволюции
приведены в табл. 1.6. Отметим, что на молекулярном уровне
конвергенция белковых молекул не сводится к схожести их
аминокислотных последовательностей: функциональное сходство
белков может достигаться и схожестью их архитектуры.
1.11. Естественный отбор и неодарвинизм
В рассматриваемых в разд. 1.8 примерах мы исходили из то-
го, что любые мутации в родительских геномах не изменяют
приспособленности (fitness, adaptation) дочерних геномов к окру-
жающей среде — характеристик, сводящихся в конечном счете
к плодовитости родителей и их потомства. Соответственно мы
подразумевали, что все мутации имеют одинаковые, 100%-ные,
вероятности быть переданными дочерним геномам и закрепить-
ся в популяции потомков. Однако сформулированная Дарвином
теория естественного отбора исходит из того, что вероятность
закрепления (отбора) некоего наследственного признака в попу-
ляции зависит от качеств этого признака — от того, насколько
он способствует преимущественному выживанию и преимуще-
ственному производству плодовитого потомства особью, этим
признаком обладающей [Darwin, 1871]. Теория естественного
отбора, законы наследственности и динамика генов в популяции
подробно изучаются в курсах общей биологии и генетики. Если у
особи в популяции произошла мутация, и через некое время эта
мутация присутствует у всех особей этой популяции, то говорят
о закреплении, фиксации (fixation) данной мутации в популяции.
Если через некое время эта мутация отсутствует у всех особей
этой популяции, то говорят о потере (loss) или вымирании
(extinction) данной мутации в популяции.
В современной науке эволюционные идеи Дарвина объеди-
нены с данными других наук, прежде всего — генетики, в син-
тетическую теорию эволюции, называемую неодарвинизмом.
В молекулярной эволюции неодарвинизм понимает мутацию как
единственный источник изменчивости, а факторы естественного
1.12. Закрепление мутации в популяции 31
отбора — как ведущие движущие силы эволюции (evolutionary
driving forces), определяющую судьбу мутаций — их преиму-
щественное закрепление или потерю. Неодарвинизм подразу-
мевает, что конкретная мутация может изменить — улучшить
или ухудшить — или не изменить приспособленность организма
к окружающей среде. Мутации, улучшающие приспособленность
организма, подвергаются действию положительного естествен-
ного отбора — эволюционным силам, факторам, направленным
на отбор такой мутации, ее преимущественное закрепление в по-
пуляции. Мутации, ухудшающие приспособленность организма,
подвергаются действию отрицательного естественного отбора —
эволюционным силам, факторам, направленным на преимуще-
ственное удаление такой мутации из популяции. Мутации, не из-
меняющие приспособленности организма к окружающей среде,
называют селективно нейтральными (neutral). Вероятность их
закрепления или удаления из популяции определяется не эво-
люционными силами, а исключительно вероятностными, стоха-
стическими процессами. Процесс изменения частоты мутации
в популяции под действием стохастических процессов называют
случайным генетическим дрейфом (random genetic drift).
Для кодирующих нуклеотидных последовательностей основ-
ными факторами естественного отбора являются эволюционные
факторы, действующие на уровне белка. Соответственно дей-
ствию этих факторов подвергаются несинонимичные, изменя-
ющие белок, но не синонимичные замены. В целом синони-
мичные замены принято считать селективно нейтральными,
или близкими к нейтральным. Однако существуют и факторы
естественного отбора, действующие преимущественно и даже
исключительно на уровне синонимичных замен (разд. 5.2).
1.12. Закрепление мутации в популяции
Математически приспособленность некой исходной последова-
тельности (дикого типа, wild type) к окружающей среде можно
обозначить за 1, а изменение ее приспособленности после некой
мутации, селективное преимущество или селективный недоста-
ток у дочерней, мутантной последовательности — за я. В таком
случае приспособленность дочерней последовательности к окру-
жающей среде будет равна 1 + я, где s имеет положительное или
отрицательное значения для мутаций, улучшающих или ухудша-
ющих приспособленность последовательности, соответственно.
Вероятность закрепления (отбора) этой мутации в популяции, Р,
at.
1лава 1. цели, принципы и понятия молекулярной эволюции
будет зависеть от з. Теория естественного отбора подразумевает
преимущественное, но не обязательное закрепление либо уда-
ление мутации, соответственно улучшающей («полезная» мута-
ция) или ухудшающей («вредная» мутация) приспособленность.
Таким образом, даже для мутации, ухудшающей приспособ-
ленность последовательности, существует ненулевая вероятность
быть закрепленной в популяции.
При обсуждении вероятности отбора мутации необходимо
ввести понятие эффективного размера, или численности, попу-
ляции (effective population size), Ne. В популяционной биологии
размер популяции N определяется как общее число особей
(последовательностей) в популяции. Однако (далеко) не все
они принимают участие в репродукции, соответственно — в эво-
люционных процессах. В первом приближении эффективный
размер популяции можно понимать как ту часть популяции,
которая принимает участие в размножении, соответственно —
в эволюционных процессах. Более строго понятие эффективного
размера популяции определяется как число размножающихся
особей, достаточное для описания процессов распределения ча-
стот мутаций во всей популяции при случайном генетическом
дрейфе [Wright, 1931; Wright, 1938].
Показано, что вероятность закрепления мутации в популяции
диплоидных организмов определяется формулой
1 _ „-4Nrsq
Р = ----------
1 _ e-ANts ’
где q — начальная частота мутации в популяции [Kimura, 1962].
Если в популяции размером .V происходит одна мутация,
то ее начальная частота для диплоидных организмов будет равна
(1/2)А’.
Поскольку е~х « (1 — .г) при малых т, то при приближении
s к 0 (т. е. для мутаций, близких к нейтральным) Р ~ q. Таким
образом, вероятность нейтральной мутации быть зафиксирован-
ной в популяции равна ее начальной частоте. Соответственно,
чем меньше размер популяции, тем более вероятно закрепле-
ние нейтральной мутации (эффект бутылочного горлышка, см.
ниже).
Если эффективный размер популяции равен размеру популя-
ции, уравнение для расчета вероятности закрепления мутации
в популяции диплоидных организмов можно переписать как
р= l~e~2S
1.12. Закрепление мутации в популяции 33
При небольших положительных значениях з (разумно счи-
тать, что каждая отдельная мутация будет лишь незначительно
увеличивать приспособленность особи) и больших значениях N,
Р ~ 2s. Таким образом, вероятность фиксации мутации с з = 0.01
составит 2%.
Рассмотрим численный пример. Предположим, что в по-
пуляции, состоящей из тысячи особей, произошли мутации
с s = 0.0,01 и —0,001. Если упрощенно предполагать, что Ne = N,
то вероятности закрепления этих мутаций будут составлять 0,05,
2 и 0,004% соответственно. Таким образом, даже улучшающие
приспособленность организма к окружающей среде мутации
с s = 0,01 будут в 98% случаев, тем не менее, потеряны, удалены
из популяции в силу случайных событий. Даже при наличии
факторов естественного отбора, случайные события играют
значительную роль в эволюции живых систем.
Отметим, что роль случайных событий возрастает при умень-
шении Nf. Возрастание роли случайных событий при резком
уменьшении численности популяции называют эффектом гене-
тической воронки, или бутылочного горлышка (bottleneck effect).
В предельном случае, когда от некой гетерогенной популяции
(для простоты будем рассматривать гаплоидные бесполые ор-
ганизмы) остается только одна особь — например, в результате
катастрофы, — то все имеющиеся у этой особи мутации, включая
и ухудшающие приспособленность, будут со 100%-ной вероят-
ностью закреплены в дочерней популяции. В таких случаях
говорят об эффекте основателя (founder effect) [Mayr, 1954;
Mayr, 1963]. Подобные эффекты проявляются и в случае про-
никновения ограниченного числа особей в новую восприимчивую
среду обитания, что часто происходит, например, при распро-
странении патогенов. В таких случаях победитель — имеется в
виду первый проникший в новую среду обитания — получает
все, т.е. закрепляет в дочерней популяции все имеющиеся у него
мутации. Эффект основателя наблюдается и при межвидовом пе-
реносе вирусов — скажем, парвовируса кошек собакам [Lukashov
and Goudsmit, 2001], — и при внутривидовом распространении
вируса в новую, ранее этим вирусом не пораженную, популя-
цию — скажем, при распространении вируса иммунодефицита
человека первого типа, ВИЧ-1, на территории бывшего СССР
(подробно рассмотрено в разд. 5.5.3).
Кроме собственно вероятности закрепления мутации в попу-
ляции, важно рассмотреть и время закрепления мутации, t —
число поколений, необходимое для закрепления мутации. При
3 —2238
J4
ишвд i. цели, принципы и понятия молекулярной эволюции
этом следует говорить о среднем условном времени закрепления
мутации, учитывая тот факт, что большинство мутаций будет
потеряно.
Показано, что если начальная частота мутации в популяции
диплоидных организмов составляет (1/2)Аг, то для нейтраль-
ных мутаций, которые в конечном счете будут закреплены
в популяции, среднее условное время закрепления t составит
4N поколений [Kimura and Ohta, 1969].
Для мутаций с селективным преимуществом s, которые будут
закреплены в популяции, среднее условное время закрепления
составит
2
t = - ln(2./V) поколений,
s
Таким образом, для популяции диплоидных организмов чис-
ленностью в миллион особей со временем репродукции в два
года, для закрепления нейтральной мутации потребуется в сред-
нем 8 миллионов лет. Для сравнения, закрепленные в конечном
счете в этой популяции мутации с селективным преимуществом
в 0,01 будут в среднем закреплены за 5800 лет.
Показано, что для мутации с селективным недостатком s
среднее условное время закрепления равно среднему условному
времени закрепления мутации с селективным преимуществом
s [Maruyama and Kimura, 1974]. Таким образом, закрепление
мутации с селективным недостатком в 0,01 также произойдет
в среднем за 5800 лет. Этот, на первый взгляд, парадоксальный
математический результат, тем не менее, легко понять с точки
зрения естественного отбора. Движущие силы естественного от-
бора направлены на преимущественное удаление из популяции
мутации с селективным недостатком. Однако судьба такой мута-
ции определяется и стохастическими, случайными процессами.
Если стохастические процессы в течение достаточно короткого
времени не приведут случайным образом к закреплению такой
мутации, то она будет удалена из популяции постоянно действу-
ющими факторами отрицательного отбора. Соответственно такая
мутация будет либо закреплена в популяции относительно быст-
ро, либо, в противном случае, потеряна. Таким образом, улуч-
шающие и ухудшающие приспособленность организма мутации
с одинаковым по модулю значением л будут в среднем условно
закреплены за одинаковое время. Однако процент улучшающих
приспособленность мутаций, закрепленных в популяции, будет
значительно большим.
1.14. Нейтральная теория молекулярной эволюции 35
1.13. Концепция молекулярных часов
Поскольку ошибки, происходящие при репликации генетическо-
го материала, могут происходить при каждом цикле реплика-
ции, то, чем больше проходит этих циклов, тем больше может
произойти ошибок, мутаций. Кроме того, закрепление мутации
в популяции занимает определенное время. Таким образом, чем
больше времени проходит после момента дивергенции после-
довательностей, форм жизни, тем, в общем случае, больше
эволюционных различий будет наблюдаться между ними.
Впервые этот феномен был отмечен при анализе аминокис-
лотных последовательностей гемоглобина и цитохрома С у раз-
личных организмов [Zuckerkandl and Pauling, 1962; Margoliash,
1963]. В дальнейшем Цукеркандль и Полинг сформулировали
концепцию существования молекулярных часов (molecular clock)
в эволюции живых систем [Zuckerkandl and Pauling, 1965].
Эта концепция утверждает, что для конкретной генетической
последовательности скорость эволюции постоянна во времени
и одинакова у всех дочерних последовательностей, т. е. во
всех нисходящих эволюционных линиях. Иначе говоря, если
известно, что дивергенция двух различающихся между собой
на один нуклеотид последовательностей произошла 10 лет тому
назад, то дивергенция между этими двумя последовательно-
стями и третьей последовательностью, имеющей два отличия
от них, произошла 20 лет тому назад (доверительный интервал
для этой даты может быть рассчитан с помощью соответству-
ющего математического аппарата).
Математические подходы, основанные на концепции моле-
кулярных часов, широко используются при изучении филоге-
нетических отношений между формами жизни и датировании
эволюционных событий. Вопросы, связанные с анализом моле-
кулярных часов, подробно разбираются в разд. 5.3.
1.14. Нейтральная теория молекулярной эволюции
В конце 1960-х гг. Кимура [Kimura, 1968] и независимо
от него — Кинг и Джукс [King and Jukes, 1969] сформулирова-
ли гипотезу, получившую впоследствии название нейтральной
теории молекулярной эволюции (neutral theory of molecular
evolution) [Kimura, 1983]. Книга Кимуры переведена на русский
язык [Кимура, 1985]. В отличие от (традиционно понимаемого)
дарвинизма, нейтральная теория молекулярной эволюции посту-
36 Глава 1. Цели, принципы и понятия молекулярной эволюции
лирует, что на молекулярном уровне подавляющее большинство
эволюционных изменений (практически — все изменения) опре-
деляются не действием факторов положительного отбора, а слу-
чайным генетическим дрейфом селективно нейтральных (или
близких к таковым) мутаций. Согласно нейтральной теории,
судьба мутаций в популяции определяется исключительно сто-
хастическими процессами. Генетическая гетерогенность любой
популяции, таким образом, в любой момент времени представ-
ляет собой переходное состояние, некий разрез динамического
процесса, в котором каждая конкретная мутация находится
на пути либо к ее закреплению в популяции, либо к ее потере.
Соответственно, с точки зрения нейтральной теории молеку-
лярной эволюции, любая конкретная наблюдаемая генетическая
гетерогенность популяции по своей сути преходяща.
Теория нейтральной эволюции в значительной степени ба-
зируется на экспериментальных данных о молекулярных часах
в эволюции живых систем (хотя, по мнению автора этой книги,
существование молекулярных часов можно понять и принять
и с точки зрения дарвинизма).
Теория нейтральной эволюции вызвала (и продолжает вы-
зывать) значительную критику со стороны (традиционных) дар-
винистов, но одновременно явилась катализатором становления
и развития многих идей и методов, лежащих в основе совре-
менного эволюционного анализа. Если эволюция (в значительной
степени) определяется стохастическими процессами, то для опи-
сания, анализа эволюционного процесса должен и может быть
применен имеющийся математический аппарат, описывающий
случайные процессы, теория вероятности.
До сегодняшнего времени нейтральную теорию молекулярной
эволюции принято противопоставлять неодарвинизму. Пример
дискуссии между этими направлениями обсужден в разд. 5.2.1.
Однако, по мнению автора этой книги, эти два направления
эволюционного учения можно и следует понимать не как проти-
востоящие, взаимоисключающие, но как дополняющие, обогаща-
ющие друг друга. Обе теории исходят из того, что большинство
новых мутаций ухудшают приспособленность организма, быст-
ро теряются в популяции и, таким образом, не наблюдаются
при эволюционном анализе. Далее, неодарвинизм не постулирует
обязательного отбора мутаций, улучшающих приспособленность
организма, но говорит о преимущественном их закреплении
в популяции. Как обсуждалось в разд. 1.12, улучшающие при-
способленность организма к окружающей среде мутации могут
1.15. Эволюционная систематика 37
быть, тем не менее (и даже — в подавляющем большинстве слу-
чаев), потеряны. Таким образом, их судьба с точки зрения и нео-
дарвинизма, и теории нейтральной эволюции, в значительной
степени определяется стохастическими процессами. С другой
стороны, нейтральная теория не отрицает наличия мутаций,
улучшающих приспособленность организма, не утверждает, что
абсолютно все закрепленные мутации являются нейтральными,
но постулирует, что таких мутаций — (подавляющее) большин-
ство.
1.15. Эволюционная систематика
Как отмечалось в разд. 1.1, одной из ключевых задач молеку-
лярной эволюции как науки является создание классификации
форм жизни — систематики, таксономии.
Безусловно, понимание необходимости и стремление решить
эту задачу существовали задолго до начала молекулярной эры
в биологии. Еще Линнеем были сформулированы принципы
систематики растений, живых (и неживых — минералы) систем
вообще [Linnaei, 1735]. Не останавливаясь подробно на анализе
предложений Линнея, описанных в имеющейся учебной литера-
туре, отметим ключевое для нашей книги положение: при раз-
работке классификации форм жизни целью Линнея было исклю-
чительно облегчение идентификации растений и животных как
представителей либо нового, либо уже известного вида, рода,
семейства и т. д. После изучения морфологических признаков
некоего организма, он мог быть легко классифицирован путем
сравнения выявленных признаков с описанными для известных
видов. Принципы систематики Линнея, ее последующее разви-
тие базировались исключительно на установлении схожести
наблюдаемых морфологических признаков форм жизни, но
не на установлении их общего эволюционного происхождения.
Одним из классических примеров здесь является использование
наличия у животных копыт как важного классифицирующего
признака, в связи с чем выделялась группа копытных животных
(надотряд Ungulata), объединявшая в том числе парнокопытных
коров (отряд Artiodactyla) и непарнокопытных лошадей (отряд
Perissodactyla). Наличие копыт у некоего нового вида животных
позволяло классифицировать этот вид как представителя группы
копытных с последующей более детальной классификацией
на основании других морфологических признаков (включая
и число копыт).
^ICIDQ
. цели, ПРИНЦИПЫ И ПОНЯТИЯ МОЛвКуЛЯрНОЙ ЭВОЛЮЦИИ
Предложенные Линнеем подходы к систематике не базирова-
лись, и не могли базироваться, на принципе общего эволюцион-
ного происхождения форм жизни. Доминировавшая во времена
Линнея догма о неизменности видов, полностью им разделяемая,
делала невозможным саму постановку вопроса об эволюционных
отношениях между формами жизни, их филогении.
По мере развития палеонтологии, показавшей изменение
форм жизни, и эволюционного учения вообще, линнеевские
принципы систематики смогли достаточно просто (бесконфликт-
но и быстро, в отличие от ряда других направлений биологии)
учесть идеи об изменчивости видов. Принципы Линнея стали
применяться и для описания ископаемых останков, окамене-
лостей (fossils), систематики вымерших организмов как пред-
ставителей той или иной группы существующих организмов
(или новой группы) на основании анализа морфологических
признаков. Однако сам подход к систематике остался при этом
прежним.
Только в середине XX века Хеннигом были сформулированы
принципиально новые идеи о том, что в основе классификации
форм жизни должны лежать родственные, эволюционные отно-
шения между ними: классификация должна быть филогенети-
ческой [Hennig, 1950J. Для этого подхода Хенниг предложил
термин «кладистика» (cladistics), в отличие от традиционно-
го, фенетического, подхода (phenetics). Так, копыта у парно-
и непарнокопытных животных имеют различное эволюционное
происхождение, соответственно — наличие копыт не дает ника-
ких оснований для выделения группы копытных животных. Эво-
люционно парнокопытные гораздо более родственны, скажем,
китам (отряд Cetacea), чем лошадям. Более подробно различия
между кладистикой и фенетикой обсуждены в разд. 4.7.
Несмотря на то что кладистические идеи достаточно быстро
получили широкое признание (хотя по целому ряду вопросов
споры между сторонниками фенетического и кладистического
подходов продолжаются до сих пор), их практическое примене-
ние в биологии до начала молекулярной эры было ограничено.
Понимание того, что генетические последовательности могут
быть использованы для изучения филогении форм жизни, по-
явилось сразу же после открытия структуры ДНК [Crick, 1958].
Однако только развитие методов быстрого получения и анализа
большого объема генетической информации за последние два
десятилетия предоставило широкие возможности для эволюци-
онного анализа генетического материала. Подчеркнем, что мор-
1.16. Проведение эволюционного анализа 39
фологические признаки также могут быть проанализированы
с помощью кладистического подхода: различие между фене-
тикой и кладистикой определяется не типом анализируемых
данных (морфологические, генетические и др.), но подходом к их
анализу.
1.16. Проведение эволюционного анализа
В заключение этой главы кратко просуммируем ее ключевые по-
ложения и опишем логику и порядок проведения эволюционного
анализа.
Генетическая информация в живых системах представлена
молекулами нуклеиновых кислот, последовательностями нук-
леотидов. При репликации этих молекул могут происходить
ошибки, мутации, в том числе — замены, вставки и делеции
нуклеотидов. В результате дочерние геномы будут отличаться
от родительских. Разные мутации происходят с разной часто-
той, вероятностью. Эти мутации могут приводить к изменению
приспособленности живой системы и подвергаться действию
факторов естественного отбора, или быть эволюционно ней-
тральными. Кроме факторов естественного отбора, случайные,
стохастические процессы (в значительной степени) определяют
судьбу мутаций — их эволюционное закрепление или потерю.
Каждая родительская последовательность может давать
несколько дочерних последовательностей, и мутации в этих
дочерних последовательностях, вообще говоря, будут различны-
ми. Дальнейшая независимая эволюция этих дочерних после-
довательностей лежит в основе видообразования (дивергенция
между эволюционными линиями).
Все эти эволюционные события происходят во времени. Чем
больше времени прошло с момента разделения независимых
эволюционных линий, тем, в общем случае, больше генети-
ческих различий будет наблюдаться между ними (концепция
молекулярных часов).
В результате всех этих событий имеющаяся у живых систем
генетическая информация будет различной, их нуклеотидные
последовательности будут отличаться друг от друга.
Реконструкцией родственных отношений между формами
Жизни на основании анализа их генетических последователь-
ностей, установлением законов эволюционных изменений на-
следственной информации занимается молекулярная филогене-
тика — ключевой раздел молекулярной эволюции. При филогене-
i *izi mvjivxvj'Jizijjhvjh дВиЛЮЦЙИ
тическом анализе, гомологичные (имеющие общее эволюционное
происхождение) генетические последовательности анализируе-
мых форм жизни сравнивают между собой с помощью различ-
ных методов.
Поскольку при эволюции последовательностей происходят
как замены, так и вставки-делеции нуклеотидов, то первым
этапом эволюционного анализа в общем случае является вы-
явление этих вставок-делеций. Этот этап анализа, называемый
выравниванием последовательностей (гл. 2), необходим для того,
чтобы установить гомологичные позиции анализируемых после- |
довательностей.
После процедуры выравнивания последовательностей можно
оценить число и качество различий между ними, рассчитать
эволюционные дистанции (гл.З). Анализируя генетические раз-
личия между последовательностями с помощью различных ме-
тодов, можно реконструировать их эволюционную историю, род-
ственные отношения между ними (собственно филогенетический
анализ, гл. 4).
При проведении филогенетического анализа возникает ряд
специальных вопросов, описываемых в гл. 5.
Эволюционный анализ генетической информации проводится
при помощи многочисленных компьютерных программ с исполь-
зованием различных баз генетических данных, описываемых
в гл. 6.
В качестве самостоятельного раздела науки, со своими соб-
ственными задачами и методами исследований молекулярная фи-
логенетика является ключевой составляющей более общего на-
правления науки — биоинформатики, включающей в себя в том
числе и сравнительную геномику, науку о взаимоотношениях
между структурами и функциями макромолекул. Филогенетика
и сравнительная геномика разделяют один и тот же объект
исследования, макромолекулы во многом взаимосвязаны и опи-
раются на результаты друг друга. В конце нашей книги приведен
список книг для дополнительного чтения, включая и литературу
по сравнительной геномике.
।лава l
ВЫРАВНИВАНИЕ
ГЕНЕТИЧЕСКИХ
ПОСЛЕДОВАТЕЛЬНОСТЕЙ
2.1. Цели выравнивания последовательностей
Как описывалось в разд. 1.5, в эволюции генетических последо-
вательностей происходят как замены, так и вставки и делеции.
Первым этапом филогенетического анализа является иденти-
фикация вставок и делеций, имевших место в эволюционной
истории анализируемой группы последовательностей. Эту про-
цедуру называют выравниванием (to align, alignment) после-
довательностей. Выравнивание последовательностей направлено
на выявление гомологичных (имеющих общее эволюционное
происхождение) позиций анализируемых последовательностей,
установление наиболее вероятного, т. е. требующего наименьше-
го числа эволюционных событий, сценария эволюции анализи-
руемой группы.
2.2. Принципы
выравнивания последовательностей
Рассмотрим гомологичные нуклеотидные последовательности 1
и 2:
111111111122
123456789012345678901
1 ATACCTGCGATA GCTTCTGAТ
| | | | | | | | | | **********
2 ATACCTGCGAAffCrrCTffAr.
Как видно, первые десять нуклеотидов у этих последователь-
ностей одинаковы (здесь и далее отмечены вертикальными лини-
ями), а последующие нуклеотиды — неодинаковы (здесь и далее
отмечены звездочками). Кроме того, последовательность 1 длин-
нее последовательности 2 на один нуклеотид. Можно предпо-
лагать, что после дивергенции этих последовательностей от их
(неизвестного) общего предка произошли как минимум десять
нуклеотидных замен и делеция последнего нуклеотида в после-
довательности 2, т. е. минимум одиннадцать мутаций. Однако
1ливи z. выравнивание генетических последовательностей
отметим, что нуклеотиды 11-20 последовательности 2 присут-
ствуют в том же порядке и в последовательности 1, но находятся
в позициях 12-21 (выделены курсивом). Таким образом, более
вероятным (подразумевающим наименьшее число эволюцион-
ных событий) сценарием эволюции этих последовательностей
было не одиннадцать, а одно эволюционное событие: делеция
нуклеотида Т в позиции 11 последовательности 2. В таком слу-
чае последовательности 1 и 2 могут быть выровнены следующим
образом:
111111111122
123456789012345678901
1 ATACCTGCGATAGCTTCTGAT
IIIIIIIIII IIIIIIIIII
2 ATACCTGCGA.AGCTTCTGAT
Отметим, что в общем случае равновероятным будет и сцена-
рий, когда в последовательности 1 после позиции 10 произошла
вставка нуклеотида Т. Как правило, при анализе генетических
последовательностей неизвестно, имела ли место вставка в этой
позиции в одной последовательности или же делеция — в со-
ответствующей позиции другой последовательности. Поэтому
говорят о наличии пробела (gap) в этой позиции последова-
тельностей. В англоязычной литературе используется и термин
индел (indel, от insertion-de/etion).
Подчеркнем еще раз уже упомянутое в разд. 1.16 и 2.1 поло-
жение, что процедуру выравнивания следует понимать как уста-
новление гомологичных участков генетических последователь-
ностей: после проведения выравнивания предполагается, что
каждая позиция последовательности 1 является гомологичной
соответствующей позиции последовательности 2.
Все эти рассуждения справедливы как для нуклеотидных,
так и для аминокислотных последовательностей.
Как отмечалось выше, базовым принципом выравнивания по-
следовательностей является установление наиболее вероятного
сценария их эволюции, т. е. такого сценария, который требовал
бы минимального числа эволюционных событий: минимально-
го числа замен при минимальном же числе пробелов. Таким
образом, целью выравнивания является максимизация числа
одинаковых нуклеотидов или аминокислот в соответствующих
позициях последовательностей при минимизации числа необхо-
димых для этого пробелов.
2.2. Принципы выравнивания последовательностей 43
Рассмотрим другие нуклеотидные последовательности 1 и 2:
111111
123456789012545
1 AGATCCGACTCTACG
11*11♦ **I ******
2 AGGTCGTTCAGACGT
(А)
Как видно, эти две последовательности имеют одинаковую
длину. Таким образом, можно предполагать, что в эволюции
этих последовательностей не происходили делеции или встав-
ки. Если это так, то эти последовательности можно считать
выровненными (случай А). Поскольку пять из пятнадцати нук-
леотидов у последовательностей одинаковы, то при выравни-
вании последовательностей таким способом следует считать,
что в эволюции этих последовательностей произошли минимум
десять нуклеотидных замен. Однако можно предполагать, что,
несмотря на одинаковую длину последовательностей, в их эво-
люционной истории происходили делеции или вставки. Если это
так, то рассматриваемые последовательности можно выровнять
различными способами:
1 AGATCCGACTCTACG.
||*||***|** ||| (Б)
2 AGGTCGTTCAG.ACGT
1 AGATCCGA.CTCTACG.
||*|| |* |* *||| (B)
2 AGGTC.GTTCA.GACGT
1 AGATCCGACTCT.ACG.
11*11 **ll* III (Г)
2 AGGTC..GTTCAGACGT
1 AGATCCGACTCT.ACG.
11*11 1* II* III (Д)
2 AGGTC.GT.TCAGACGT
1 AGATCCGACTCT.ACG.
11*11 1 *11* III (E)
2 AGGTC.G.TTCAGACGT
44
1 лава z. выравнивание генетических последовательностей
Как видно, в случае Б у последовательностей совпадают
восемь нуклеотидов, и присутствуют два пробела длиной в один
нуклеотид каждый. В случае В совпадают девять нуклеоти-
дов, и присутствуют четыре пробела длиной в один нуклеотид
каждый. Такое же число совпадающих нуклеотидов — ив слу-
чае Г. Однако в случае Г присутствуют три пробела, один
из которых — длиной в два нуклеотида. В случаях Д и Е —
число совпадающих нуклеотидов возрастает до десяти, число
пробелов — до четырех.
Из рассматриваемого примера понятно, что в общем случае,
чем больше пробелов вводится при выравнивании последова-
тельностей, тем меньше различий (замен) эти последователь-
ности имеют после выравнивания. В рассматриваемом примере
при увеличении числа пробелов от нуля до четырех число
замен уменьшается от десяти (случай А) до трех (случаи
Д и Е). Однако целью выравнивания является установление
эволюционного сценария, подразумевающего наименьшее чис-
ло всех эволюционных событий: не просто наименьшее число
замен, но наименьшее число замен при наименьшем же чис-
ле пробелов. Таким образом, понятно, что в решении задачи
минимизации числа эволюционных событий подзадачи мини-
мизации числа замен (т. е. максимизации числа одинаковых
нуклеотидов или аминокислот) и минимизации числа/длины
пробелов находятся в естественном, неизбежном противоречии
друг с другом. Решение этой задачи требует количественного
сопоставления вероятностей различных эволюционных событий:
какой из приведенных выше случаев А-Е наиболее вероятен?
Является ли более вероятным сценарий А, согласно которому
в эволюционной истории анализируемых последовательностей
произошли десять замен — или, скажем, сценарий Б, согласно
которому имели место шесть замен и две вставки или деле-
нии?
Естественным подходом к решению этой задачи являет-
ся введение системы положительных и отрицательных оценок
(scores), присваиваемых для проведения выравнивания различ-
ным эволюционным событиям. Более вероятным событиям —
скажем, сохранению нуклеотида (аминокислоты) присваивают-
ся большие (положительные) значения, менее вероятным —
меньшие или даже отрицательные (штрафы, penalty) значения.
Так, штрафы присваиваются за несовпадение нуклеотида или
аминокислоты (mismatch penalty), начало пробела (gap opening
penalty) и продолжение пробела (gap extension penalty). Причи-
2.2. Принципы выравнивания последовательностей 45
цы введения различных штрафов за начало и продолжения про-
бела объяснены ниже. Критерием оптимальности выравнивания
последовательностей является сумма всех наложенных оценок:
чем она больше, тем оптимальнее (ближе к реконструируемому
эволюционному сценарию) выравнивание.
Предположим, что в рассматриваемых случаях А-Е мы ис-
пользуем систему оценок, в которой сохранению нуклеотида
присвоено значение 1, а все три вида штрафов одинаковы
и равны —1. Тогда сумма оценок по всем позициям составит —5
для случая А (пять совпадающих и десять несовпадающих
нуклеотидов, 5—10), 0 для случая Б (восемь совпадающих, шесть
несовпадающих нуклеотидов, два начала пробела, 8 — 6 — 2),
1 для случаев В (девять совпадающих, четыре несовпадающих
нуклеотида, четыре начала пробела, 9 — 4 — 4) и Г (девять
совпадающих, четыре несовпадающих нуклеотида, три начала
пробела, одно продолжение пробела, 9 —4 —3 — 1)иЗ для случаев
Д и Е (десять совпадающих, три несовпадающих нуклеотида,
четыре начала пробела, 10 — 3 — 4). Таким образом, при выбран-
ной системе штрафов оптимальными способами выравнивания
последовательностей следует считать случаи Д и Е.
Однако известно, что в эволюции генетических последова-
тельностей замены нуклеотидов происходят гораздо чаще, чем
делеции и вставки. Учитывая это, логично присваивать больший
штраф за начало пробела, чем за несовпадение нуклеотидов.
При увеличении штрафа за начало пробела, скажем до —3,
при остальных штрафах (за несовпадающий нуклеотид и про-
должение пробела) в —1, общий счет составит —5 для случаев
А (5-10), Г (9-4-3x3 - 1), Д (10-3-4x3) и Е (10-3-4x3),
-7 для случая В (9 — 4 — 4x3),а оптимальным будет считаться
выравнивание в случае Б, для которого общий счет составит —4
(8 - 6 - 2 х 3).
Как правило, в практической работе при выравнивании по-
следовательностей штраф за несовпадение нуклеотида устанав-
ливают в —1, начало пробела штрафуют гораздо более серьезно
(скажем, штраф от —3 до —20 и даже серьезнее), а штраф
за продолжение пробела устанавливают около —1. Причина вве-
дения отдельных штрафов за начало пробела (большого штрафа)
и его продолжения (много меньшего штрафа) связана с тем, что
вставка или делеция нескольких последовательных нуклеотидов
может быть результатом и одного эволюционного события. По-
скольку пробел длиной, скажем, в десять нуклеотидов может
являться результатом и одного эволюционного события, то его
«с i ичсских последовательностей
нельзя штрафовать так же сильно, как и десять пробелов длиной
в один нуклеотид каждый, поскольку эти десять пробелов —
результат десяти эволюционных событий.
Системы положительных и отрицательных оценок могут
быть и гораздо более сложными. Например, известно, что раз-
личные нуклеотидные замены и аминокислотные замещения
имеют неодинаковые вероятности: так, при нуклеотидных за-
менах транзиции (разд. 1.6.1) более вероятны, чем трансвер-
сии, а, скажем, гидрофобные аминокислоты чаще замещаются
гидрофобными, но не гидрофильными (разд. 1.3). Естественно,
эти неодинаковые вероятности можно учитывать и при уста-
новлении системы штрафов для выравнивания последовательно-
стей, устанавливая, скажем, штраф за транзиции меньше, чем
за трансверсии. Особое распространение неодинаковые штра-
фы получили при выравнивании аминокислотных последова-
тельностей, для которых предложены таблицы, отражающие
вероятности сохранения каждой аминокислоты и ее замещения
каждой другой аминокислотой. Эти таблицы (матрицы) подробно
описаны в разд. 3.4.
2.3. Алгоритмы выравнивания
двух последовательностей
2.3.1. Принцип матрицы точек
Как показывают рассмотренные в разд. 2.2 примеры, выравнива-
ние близкородственных (не сильно отличающихся друг от друга)
последовательностей является достаточно простой задачей и мо-
жет быть произведено при их визуальном анализе. Для более
сложных случаев следует использовать специальные компьютер-
ные программы (разд. 6.4).
Основным компьютерным алгоритмом для выравнивания
двух генетических последовательностей является матричный
подход, в простейшем случае — метод матрицы точек (dot-matrix
method). При таком подходе две выравниваемые последователь-
ности записываются как строка и столбец таблицы, и все оди-
наковые нуклеотиды (или аминокислоты) обозначаются точками
в соответствующих ячейках таблицы (рис. 2.1). Если две после-
довательности идентичны, то точки будут находиться в каждой
ячейке по диагонали таблицы (рис. 2.1,а). Если две последо-
вательности имеют замены, но не имеют пробелов, то точки
будут находиться в большинстве ячеек по диагонали таблицы
2.3. Алгоритмы выравнивания двух последовательностей 47
последовательности
до выравнивания:
HKRWRWMKG
| | | | |****
HKRWRMKGA
и после выравнивания:
HKRWRWMKG.
Hill III
HKRWR.MKGA
рис. 2.1. Использование метода матрицы точек для выравнивания
последовательностей
4в Глава 2. Выравнивание генетических последовательностей
(рис. 2.1,6). Если же в анализируемых последовательностях име-
ется пробел, то диагональ смещается по вертикали или горизон-
тали (рис. 2.1, в). Таким образом, если в выравниваемых после-
довательностях есть или только замены, или только пробелы,
то подход с использованием матрицы точек облегчает визуальное
выравнивание последовательностей: следует найти (смещенную)
диагональную линию, на которой находится наибольшее число
точек.
Естественно, что в общем случае выравниваемые последова-
тельности имеют как замены, так и пробелы. В таком случае за-
дача выравнивания сводится к поиску оптимального пути через
матрицу, т.е. пути, обладающего наибольшим общим счетом: по-
ложительные оценки за совпадение нуклеотидов (аминокислот)
минус наложенные штрафы.
Понятно, что в приведенном на рис. 2.1 примере с короткими
последовательностями возможно проанализировать все суще-
ствующие пути прохождения матрицы и выбрать из них путь
с наибольшим счетом. Однако при увеличении длин последо-
вательностей число путей прохождения матрицы (т. е. число
возможных выравниваний) резко возрастает: для двух после-
довательностей длиной в п нуклеотидов (аминокислот) число
возможных выравниваний N составляет приблизительно
22п
т. е. для двух последовательностей длиной, скажем, в 300 нук-
леотидов (аминокислот) возможно приблизительно 10179 различ-
ных вариантов выравнивания. Естественно, что расчет оценок
для каждого из возможных путей прохождения матрицы для та-
ких последовательностей практически невозможен. Однако зада-
чу нахождения оптимального пути через матрицу можно решить
с помощью подходов, основанных на принципе динамического
программирования (dynamic programming) [Eddy, 2004с].
В двух следующих разделах мы рассмотрим сначала кон-
кретные примеры алгоритмов динамического программирования
для выравнивания генетических последовательностей, а затем —
общие принципы динамического программирования. Эти два
раздела можно читать и в обратном порядке: сначала понять
общие принципы, а затем — частные примеры конкретных алго-
ритмов.
2.3. Алгоритмы выравнивания двух последовательностей 49
2.3.2. Алгоритмы Нидлмена—Вунша и Смита—Уотермена,
глобальное и локальное выравнивание
Исторически первым и широко используемым до сих пор
алгоритмом нахождения оптимального пути через матрицу,
основанном на принципе динамического программирования,
является алгоритм Нидлмена—Вунша (Neddleman—Wunsch
algorithm) [Needleman and Wunsch, 1970]. Изначально предло-
женный для выравнивания аминокислотных последовательно-
стей, этот алгоритм применим и для выравнивания нуклеотид-
ных последовательностей. Рассмотрим этот алгоритм на примере
выравнивания двух аминокислотных последовательностей 1 и 2:
1 LKRCACRWGSCHM
2 KHRCICWSGCGM
Как более детально изложено в разд. 2.3.3, оптимальный путь
через матрицу — это та последовательность переходов из ячейки
в ячейку, которая имеет наибольшую суммарную оценку. На
первом этапе выравниваемые последовательности записываются
как первая строка и первый столбец таблицы-матрицы, и ячей-
кам с одинаковыми аминокислотами присваивается значение 1,
а ячейкам с разными аминокислотами — 0 (рис. 2.2, а). После
этого определяется максимально возможная сумма чисел во всех
ячейках при всех возможных шагах через матрицу сверху-вниз
слева-направо (в данном случае максимально возможная сумма
чисел — 8). Начиная с самой верхней левой ячейки для каждой
последующей ячейки матрицы определяется максимальная воз-
можная оценка, как если бы выровненные последовательности
заканчивались в этой позиции. Для этого к начальному числу
ячейки (0 или 1) прибавляется максимальное число из строки-
столбца сверху-слева от этой ячейки (рис. 2.2,6). Оптимальное
выравнивание достигается передвижением от первой верхней
левой ячейки со значением 1 к ячейкам с большими значени-
ями (рис. 2.2, в). В данном случае оптимальное выравнивание
возможно двумя способами:
1 LK.RCACRW.GSCHM 1 LK.RCACRWGS.СНМ
I 11*1 I I 1*1 или I 11*1 | I |*|
2 .KHRCIC.WSG.CGM 2 .KHRCIC.W.SGCGM
4 - 223S
jv i лава г. выравнивание генетических последовательностей
Рис. 2.2. Принцип алгоритма Нидлмена—Вунша для выравнивания последо-
вательностей. (а) Две выравниваемые последовательности записываются как
первые строка и столбец матрицы, и ячейкам с одинаковыми в обеих последова-
тельностях аминокислотами присваивается значение I, с неодинаковыми — 0.
(б) Первый (прямой) проход матрицы. Начиная с верхней левой ячейки к
значению каждой ячейки прибавляется максимальное число из строки-столбца
сверху-слева — так, для отмеченной знаком вопроса ячейки к ее собственному
значению (0) прибавляется максимально число из предыдущих строки-столбца,
выделенных серым цветом (3), и значение этой ячейки становится равным 3.
(в) После заполнения таким образом всех ячеек матрицы, определен максималь-
но возможный счет (в данном случае — 8). Двигаясь в обратном направлении (от
ячейки со значением 8), определяется оптимальный путь прохождения матрицы.
В рассматриваемом случае два пути — два варианта перехода от значения 5
к значению 7 — являются одинаково оптимальными
2.3. Алгоритмы выравнивания двух последовательностей 51
В каждом из этих двух альтернативных вариантов вырав-
нивания у последовательностей совпадают восемь аминокислот,
что достигается пятью пробелами длиной в одну аминокислоту
каждый.
Описанный пример применения алгоритма Нидлмена—
Вунша является упрощенным (собственно, для простоты объ-
яснения авторы алгоритма при его публикации также исполь-
зовали упрощенный пример) — скажем, не введены штрафы
за пробелы (точнее, они равны 0), для любых несовпадений
аминокислот использована одинаковая, нулевая, оценка. Однако
введение штрафов и/или различных, более сложных систем
оценок не меняет сути алгоритма.
Как очевидно из обсуждаемого примера, алгоритм
Нидлмена—Вунша направлен на выравнивание последователь-
ности 1 и 2 по всей их длине. Такие подходы называют
направленными на глобальное выравнивание (global alignment).
Принципом таких подходов является максимизация числа
одинаковых нуклеотидов (аминокислот) у двух последовательно-
стей. Математически этот алгоритм направлен на максимизацию
схожести (similarity), S, двух последовательностей следующим
образом:
S = max (z — WkZkj.
где т — число совпадающих нуклеотидов (аминокислот), Wk —
штраф за пробел длиной в к нуклеотидов (аминокислот), Zk —
число пробелов длиной в к нуклеотидов (аминокислот).
Для ряда практических задач выравнивание последователь-
ности по всей их длине не является необходимым или даже
желательным. Так, алгоритм Нидлмена—Вунша не направлен
на идентификацию коротких участков последовательностей, об-
ладающих высокой схожестью. Для подобных задач разработан
алгоритм Смита—Уотермена (Smith-Waterman algorithm) [Smith
and Waterman, 1981]. Этот алгоритм подразумевает, что оп-
тимальный путь через матрицу может начинаться и заканчи-
ваться в любой ячейке, а не обязательно только в ячейках
первой и последней строки и первого и последнего столбца.
Смит и Уотермен модифицировали алгоритм Нидлмена—Вунша,
присваивая не 0, а отрицательное значение, т. е. штраф, ячейкам
с неодинаковыми нуклеотидами (аминокислотами). Математиче-
ски этот алгоритм направлен на минимизацию числа неодина-
ковых нуклеотидов (аминокислот), генетической дистанции D
52 Глава 2. Выравнивание генетических последовательностей
между последовательностями:
D = inin (у + wkzk).
где у — число неодинаковых нуклеотидов (аминокислот), w'k —
штраф за пробел длиной в к нуклеотидов (аминокислот), анало-
гичный U’k.
Алгоритм Смита—Уотермена направлен не на глобальное,
а на локальное выравнивание (local alignment). Такие алго-
ритмы широко используются, например, для поиска в базах
генетических данных последовательностей, имеющих сходство
с анализируемой последовательностью. В частности, принцип
работы программы BLAST (разд. 6.3) основан на алгоритмах
локального выравнивания.
Продемонстрировать принципиальное различие в алгоритмах
Нидлмена—Вунша и Смита—Уотермена можно следующим при-
мером. Во многих случаях требуется выровнять не полностью
перекрывающиеся последовательности. Скажем, одна из двух
выравниваемых последовательностей содержит конец гена 1
и начало гена 2, а вторая — полный ген 2 (его начало и конец).
В таком случае использование алгоритма Нидлмена—Вунша
может привести к неправильному, не имеющему биологического
смысла выравниванию:
конец._гена_1_начало_гена_2
***** I I I I I1*1***** | | | | | | |
начало_гена_2_конец._гена_2
Очевидно, что в данном случае выровнены участки последова-
тельностей, не являющиеся гомологичными. Использование же
алгоритма Смита—Уотермена приведет к выявлению гомологич-
ных участков анализируемых последовательностей:
конец_гена_1_начало_гена_2.............
I I I I I I I I I I I I I
.............начало_гена_2_конец_гена_2
Естественно, что в практической работе не всегда известно,
являются ли выравниваемые последовательности полностью или
частично перекрывающимися. Рассмотрим две последовательно-
сти:
1 GAAACACCCTCACAATTGTA
2 ATGGCAGGCTTTACACAT
2.3. Алгоритмы выравнивания двух последовательностей 53
Использование алгоритма Нидлмена—Вунша может приве-
сти к их выравниванию следующим образом:
1 GAAACACCCTC.ACAATTGTA
****||**||* |||**|
2 ATGGCAGGCTTTACACAT...
Использование алгоритма Смита—Уотермена для тех же по-
следовательностей может привести к следующему результату:
1 TTGACACACTCCCAATTGTA
11*11111 I
2 ACCCCAGGCTTTACACA.T.........
Как видно, число совпадающих нуклеотидов в обоих слу-
чаях одинаково — восемь. Однако использование алгоритма
Нидлмена—Вунша ведет к девяти неодинаковым нуклеотидам,
а применение направленного на минимизацию числа неодинако-
вых нуклеотидов алгоритма Смита—Уотермена — только к од-
ному. При этом использование алгоритма Смита—Уотермена
приводит к большему числу пробелов — однако подавляющее
большинство этих пробелов (все, кроме одного) находится
в неперекрывающихся (возможно, не гомологичных) участках
последовательностей. Таким образом, использование алгоритма
Смита—Уотермена привело к идентификации в обеих последо-
вательностях достаточно длинных участков, имеющих высокое
сходство между собой — возможно, гомологичных друг другу.
При описании различий между этими двумя алгоритмами,
подходами глобального и локального выравнивания вообще,
можно использовать физическое понятие фазового перехода.
В зависимости от выбранных исследователем начальных усло-
вий, системы штрафов, компьютерная программа выравнивания
будет исходить либо из того, что выравниваемые последователь-
ности являются полностью перекрывающимися, и стремиться
выровнять последовательности по всей их длине, либо из того,
что эти последовательности перекрываются не полностью, и
стремиться найти перекрывающиеся участки [Waterman, 1983;
Waterman, 1984; Smith et al., 1985].
Ответ на естественный вопрос о том, какое именно из ма-
тематически оптимальных выравниваний является биологиче-
ски правильным, отражающим истинные события в эволюции
спшичгашл последовательностей
выравниваемых последовательностей, не является задачей алго-
ритмов выравнивания. Алгоритмы Нидлмена—Вунша и Смита—
Уотермена направлены на поиск математически оптимального
выравнивания при заданных исследователем параметрах. Ответ
на вопрос о том, какое именно из математически оптималь-
ных выравниваний является биологически правильным, явля-
ется задачей самого исследователя, с учетом других данных
(о структуре гена, порядке функциональных доменов и т. д.).
В простых случаях (близкородственные полностью перекры-
вающиеся последовательности) алгоритмы Нидлмена—Вунша,
и Смита—Уотермена будут давать одинаковые результаты.
До сих пор мы рассматривали случаи, когда за любое несов-
падение аминокислот (нуклеотидов) в алгоритме Нидлмена—
Вунша присваивается оценка 0, а в алгоритме Смита—
Уотермена — одинаковый штраф. Однако, исходя из эксперимен-
тальных наблюдений и рассуждений о том, что вероятности
различных замещений (замен) неодинаковы, можно присваиватв
и различный штраф за различное несовпадение аминокислот
(нуклеотидов). Скажем, известно, что аминокислоты определен-
ного класса чаще замещаются аминокислотами того же класса
(скажем, гидрофобные аминокислоты — гидрофобными же). Со-
ответственно замещениям в пределах одного и того же класса
можно присваивать меньший штраф. Таким образом, для любой
аминокислоты можно оценить вероятность ее замещения любой
другой аминокислотой и присваивать конкретному замещению
больший или меньший штраф в зависимости от вероятности
этого замещения. Эти вероятности можно представить в виде
таблицы, матрицы штрафов, отражающих вероятности замеще-
ний (и сохранения) аминокислот. Исходя из различных рас
суждений и экспериментальных данных, предложен ряд таких
матриц. Наиболее часто используемые при выравнивании по-
следовательностей матрицы — РАМ и BLOSUM. Подробно этот
вопрос рассмотрен в разд. 3.4.
2.3.3. Общие принципы динамического программирования
при выравнивании последовательностей
Рассмотрим общие принципы динамического программирования
на примере выравнивания двух генетических последовательно-
стей х и у длиной соответственно впит нуклеотидов или
аминокислот (рис. 2.3).
z.a. Алгоритмы выравнивания двух последовательностей 55
Рис. 2.3. Принципы динамического программирования при выравни-
вании генетических последовательностей, (а) В любую ячейку матри-
цы, кроме ячеек первой строки и первого столбца, можно перейти
с помощью одного из трех возможных элементарных шагов, и из
любой ячейки, кроме ячеек последней строки и последнего столбца,
можно сделать три различных шага: в ячейку снизу, в ячейку справа,
и в ячейку снизу-справа по диагонали (шаги показаны стрелками).
В любую ячейку первой строки и первого столбца можно перейти
только из предыдущей ячейки той же строки или того же столбца,
соответственно, (б) Любой путь через матрицу (два возможных пути
показаны сплошными и пунктирными стрелками) пересекает j-ю стро-
ку и г-й столбец и, при этом — только однажды. Установление всех
ячеек (их г, j-координат), через которые проходит путь с наибольшим
суммарным счетом, и есть установление оптимального пути через
матрицу
56 Глава 2. Выравнивание генетических последовательностей
Как уже отмечалось (разд. 2.3.1), число всех возможных
путей через матрицу настолько велико, что сравнение всех этих
путей практически невозможно. Однако любой путь через мат-
рицу— это последовательность элементарных шагов, переходов
из одной ячейки в другую, соседнюю (рис. 2.3, а). Из некой
ячейки, кроме ячеек последнего столбца и последней строки,
можно сделать три различных шага: в ячейку снизу, в ячейку
справа и в ячейку снизу-справа по диагонали. Соответственно
в любую ячейку, кроме ячеек первого столбца и первой строки,
можно перейти с помощью одного из трех возможных элемен-
тарных шагов, из одной из трех соседних ячеек: сверху, слева,
или сверху-слева по диагонали. Отметим также, что в каждую
(кроме первой) ячейку первого столбца и первой строки можно
перейти с помощью только одного шага: из предыдущей ячейки
того же столбца или той же строки, соответственно. Переход
в соседнюю ячейку снизу означает пробел в последовательно-
сти, записанной как строка матрицы, шаг в соседнюю ячейку
справа — пробел в последовательности, записанной как столбец
матрицы. Наконец, шаг в ячейку снизу-справа по диагонали
означает выравнивание соответствующих позиций последова-
тельностей. При этом отметим, что нуклеотиды (аминокислоты)
в этих выровненных позициях необязательно совпадают у обеих
последовательностей: естественно, и несовпадающие нуклеоти-
ды (аминокислоты) могут быть выровнены.
Таким образом, суммарное число элементарных шагов в мат-
рице — несравнимо меньше числа возможных путей через мат-
рицу. Очевидно, что оптимальный путь через матрицу — это та
последовательность элементарных шагов, которая имеет наи-
большую суммарную оценку, определяемую выбранной иссле-
дователем системой штрафов. Установление последовательности
этих элементарных шагов — то же самое, что и установление
всех ячеек, через которые этот оптимальный путь проходит.
Кроме того, отметим важную и очевидную деталь: любой
путь через матрицу, в том числе и оптимальный, обязательно пе-
ресекает все столбцы и все строки матрицы, при этом пересекает
только один раз, пройдя через любые /-й столбец и j-ю строку,
путь через матрицу уже к ним не возвращается (рис. 2.3,6).
Таким образом, задача установления всех ячеек матрицы, через
которые проходит оптимальный путь, сводится к элементарным
подзадачам: для каждого г'-го столбца, следует установить ту
ячейку (или те ячейки, их j-e координаты), которая лежит
на оптимальном пути. Естественно, следует установить и как
2.3. Алгоритмы выравнивания двух последовательностей 57
именно эта ячейка должна быть пройдена: по диагонали или
с шагом вниз или направо, т. е. с пробелом.
Каждая ячейка матрицы с координатами (i.j) имеет свою
собственную изначальную (до выравнивания) оценку: положи-
тельную за совпадение г'-й и j-й позиций (совпадение нук-
леотидов или аминокислот в позиции г последовательности х
и позиции j последовательности »/) или штраф за несовпадение.
Можно использовать и простую, и сложную систему штрафов —
скажем, различные штрафы за различные несовпадения: выбор
системы штрафов, безусловно, является ключевым элементом
практической работы, но никоим образом не влияет на суть
самого принципа динамического программирования. Кроме того,
каждый шаг, каждый переход в эту ячейку с координатами (i.j)
имеет свою оценку: скажем, нулевую за переход по диагонали,
т.е. из ячейки сверху-слева с координатами (г — 1. j — 1), штраф —
за переход сверху или слева (т. е. пробел), из ячеек с координа-
тами (г, j — 1) и (i — 1, j), соответственно.
Таким образом, для любого прохода ячейки можно ввести
(промежуточную) суммарную оценку, являющуюся суммой соб-
ственной оценки ячейки и оценки перехода в нее.
Итак, через какую именно ячейку некоего г-го столбца будет
проходить оптимальный путь через матрицу? Очевидно, что
для такой ячейки г-го столбца должны соблюдаться два условия.
Во-первых, оптимальный путь до этого г-го столбца (сумма всех
оценок), т. е. до столбца i 1, должен заканчиваться в такой
ячейке столбца г — 1, откуда возможен элементарный шаг в эту
ячейку г-го столбца. Поскольку в любую ячейку (i.j) можно
перейти одним из трех элементарных шагов, то переход в ячейку
с координатой (г. j) возможен из одной из трех ячеек: с координа-
тами (г — 1, j— 1), (г— 1. j) и (i.j — 1). Таким образом, оптимальный
путь до г-ro столбца и j-й строки должен заканчиваться в одной
из этих трех ячеек. Во-вторых, шаг именно в эту ячейку (i.j)
из предыдущего либо этого столбца и предыдущей либо этой
строки должен быть оптимальным (максимальная оценка).
Понятно, что оптимальный путь до г-го столбца и j-й стро-
ки — это такой путь, который имеет наибольшую суммарную
°Ценку. Таким образом, для любой ячейки (i.j) можно рас-
смотреть уже окончательную суммарную оценку, являющуюся
суммой собственной оценки ячейки, оценок переходов в нее
и оценок всех ячеек, из которых можно перейти в ячейку (i.j),
т-е- ячеек (i—1. j —1), (г — 1, j), (i, j — 1). Из трех возможных сумм,
вМбирается наибольшая и присваивается этой ячейке (г, j).
58 Глава 2. Выравнивание генетических последовательностей
Таким образом, для расчета оценки ячейки (г, j) нужно знать
ее собственную изначальную оценку (известна), оценки перехо-
дов в нее (известны, установлены системой штрафов) и оценки
ячеек (г — 1, j — 1), (г — 1, j) и (г, j — 1), которые неизвестны. Как же
рассчитываются эти оценки?
Напомним, что три вида перехода возможны для всех ячеек
матрицы, кроме ячеек первого столбца (l,j) и первой строки
(г, 1). В первую ячейку матрицы, т. е. ячейку первого столбца
первой строки с координатами (1,1), переходы невозможны.
Соответственно окончательная суммарная оценка этой ячейки
будет ее собственной оценкой, положительной за совпадение
или штрафом за несовпадение нуклеотида (аминокислоты). Во
вторую ячейку первого столбца, ячейку (1,2), возможен только
один переход — из первой ячейки (1,1) (рис. 2.3,а). Посколь-
ку собственная оценка второй ячейки известна, как известны
и оценка первой ячейки и оценка шага, суммарная оценка второй
ячейки может быть установлена (нет необходимости выбирать
одну из трех оценок — возможен только один переход, только
одна оценка). Аналогично могут быть заполнены ячейки всего
первого столбца и всей первой строки матрицы.
После этого рассмотрим ячейку второго столбца второй стро-
ки матрицы, т. е. ячейку с координатами (2.2). В нее возможны
уже три перехода: из ячеек (2,1), (1,2) и (1,1). Поскольку
оценки всех этих трех ячеек установлены, собственное значе-
ние ячейки (2,2) известно, известна оценка каждого из трех
переходов в нее — все три возможные суммы можно установить,
выбрать из них наибольшую и присвоить ее ячейке (2,2). Так
последовательно устанавливаются оценки всех ячеек матрицы.
Оценка в самой нижней правой ячейке будет равна максимально
возможной оценке при выравнивании последовательностей х
и у. Само выравнивание ведется в обратном направлении —
снизу-вверх справа-нале во, поскольку все предыдущие опти-
мальные шаги запоминаются алгоритмом динамического про-
граммирования.
2.3.4. Методы слов
Кроме описанных выше методов динамического программиро-
вания, для выравнивания последовательностей используются
и методы слов (word methods), основанные на иерархическом
подходе. Такие методы не направлены на поиск оптимального
выравнивания двух последовательностей по всей их длине, но
2.4. Множественное выравнивание 59
существенно более эффективны при поисках последовательно-
стей, схожих с анализируемой, в больших базах генетических
данных.
В основе таких подходов лежит разбивание анализируемой
последовательности на короткие неперекрывающиеся участки
(слова) и последующее сравнение этих слов с последователь-
ностями в базе данных. Такие подходы позволяют предвари-
тельно установить участки последовательностей, которые могут
быть выровнены, и применить более медленные и аккуратные
алгоритмы выравнивания только для этих участков, что позво-
ляет избежать ненужных расчетов. Подобные принципы лежат
в основе программы BLAST (разд. 6.3), используемой для поиска
схожих последовательностей в больших базах данных. Методы
слов называют также А>кратными методами, где к — длина сло-
ва, т. е. число нуклеотидов (аминокислот) в словах, на которые
разбиваются анализируемые последовательности. Естественно,
чем меньше к, тем более трудоемок процесс выравнивания, но
и аккуратен его результат.
2.4. Множественное выравнивание
Как правило, на практике необходимо одновременно выровнять
не две, а (значительно) больше последовательностей. В таких
случаях говорят о выравнивании множества последовательно-
стей, или множественном выравнивании (multiple alignment).
Концептуально основанные на динамическом программировании
алгоритмы Нидлмена—Вунша и Смита—Уотермена могут быть
прямо применены для выравнивания более чем двух последо-
вательностей. В таком случае рассматриваемая матрица из дву-
мерной становится n-мерной, где п — число выравниваемых по-
следовательностей. Однако такой подход имеет существенные
практические ограничения. Это связано с возрастанием необ-
ходимого при таком подходе числа математических операций,
Равного тп, где т — длина последовательностей. Таким образом,
Для выравнивания пяти коротких последовательностей длиной
в 100 нуклеотидов (т. е. суммарное число нуклеотидов будет рав-
но 500) необходимо произвести 1005 = 1О10 операций. Такое же
число операций необходимо для выравнивания двух последова-
ТеЛьностей длиной в 100000 каждая (т. е. при суммарном числе
кУКлеотидов, равном 200000). Соответственно при таком подходе
Ддя выполнения множественного выравнивания, скажем, ста
С°следовательностей длиной в 10000 нуклеотидов каждая требу-
60 Глава 2. Выравнивание генетических последовательностей
ется 1О400 операций, что становится практически невыполнимой
задачей. Очевидно, что множественное выравнивание требует
специальных подходов.
Одним из наиболее широко используемых подходов к вы-
равниванию нескольких последовательностей является алго-
ритм прогрессивного множественного выравнивания (progressive
multiple alignment), основанный на проведении выравнивания
в три этапа. На первом этапе все последовательности попарно
выравниваются между собой с использованием того или ино-
го алгоритма выравнивания, и среди них выявляются группы
схожих между собой последовательностей. На втором этапе про-
изводится выравнивание последовательностей в каждой такой
группе, после чего — выравнивание групп между собой. Более
подробно этот алгоритм обсуждается в разд. 6.4 при описании
программы для множественного выравнивания Clustal.
Выравнивание последовательностей является одной из наибо-
лее трудоемких (в смысле требующегося компьютерного време-
ни) и спорных задач, возникающих при анализе генетических
последовательностей. Естественным вопросом при проведении
любого выравнивания является вопрос о том, насколько мате-
матически оптимальное выравнивание является оптимальным
биологически, эволюционно, т. е. устанавливает гомологичные
позиции и участки последовательностей. Понятно, что биоло-
гически значимое выравнивание последовательностей, скажем,
некоего гена должно выявлять и учитывать его структуру —
положения промотора, экзонов и интронов, — для того чтобы
обеспечить эволюционную оптимальность выравнивания.
Серьезные теоретические вопросы и практические проблемы
возникают при выравнивании последовательностей, имеющих
дупликации (скажем, одна из выравниваемых последовательно-
стей имеет две копии некоего гена, а другая — три), инверсии,
вставки чужеродного генетического материала (скажем, ретро-
элементов) и т. д.
Подходы к учету и решению этих и ряда других вопросов,
возникающих при выравнивании последовательностей, намече-
ны в ряде компьютерных программ (разд. 6.4), однако на сего-
дняшнее время эти проблемы далеки от своего решения.
Отметим также, что при выравнивании кодирующих нуклео-
тидных последовательностей, в общем случае удобнее и эффек-
тивнее транслировать их в аминокислотные последовательности
и провести выравнивание на аминокислотном уровне.
Глава 3
ГЕНЕТИЧЕСКИЕ ДИСТАНЦИИ
И ЭВОЛЮЦИОННЫЕ МОДЕЛИ
3.1. Наблюдаемые, истинные и расчетные дистанции
После выравнивания нуклеотидных или аминокислотных по-
следовательностей первым этапом их эволюционного анализа
в большинстве случаев является определение генетических, или
эволюционных, дистанций между ними. Эволюционная дистан-
ция между двумя последовательностями измеряется их раз-
личиями, числом и качеством нуклеотидных замен или ами-
нокислотных замещений между ними. Поскольку каждая за-
мена требует определенного времени, то с течением времени
генетическая дистанция между любыми двумя гомологичными
(т. е. происходящими от общего предка) последовательностями,
вообще говоря, увеличивается. Если предполагать, что эволю-
ция рассматриваемой группы последовательностей происходила
согласно модели молекулярных часов (разд. 1.13), т. е. скорость
эволюции (число замен за единицу времени) была постоянна
во времени и одинакова для всех последовательностей группы,
то эволюционные дистанции между последовательностями могут
быть прямо интерпретированы как филогенетические отноше-
ния между ними (формами жизни, от которых эти последова-
тельности получены). Иначе говоря, чем меньше эволюционные
дистанции между двумя последовательностями, тем менее давно
они имели общего предка и, соответственно, тем они более
родственны друг другу. Так, в рассматриваемом ниже примере
последовательность 1 имеет меньше отличий от последователь-
ностей 2 и 3 (одно отличие), чем от последовательности 4 (четыре
отличия):
111111111122222222223
123456789012345678901234567890
1 ATACGAGAAAACGCAATCCGCGAGAATGCC
2 ------G------------------------
3 т----------------------
4 -----С-------------А-------AG
Таким образом, генетические дистанции между последова-
Тельностями 1, 2 и 3 — меньше, чем между ними и после-
62 Глава 3. Генетические дистанции и эволюционные модели
довательностью 4. Соответственно при соблюдении принципа
молекулярных часов в рассматриваемой группе последователь-
ностей можно сделать вывод о более близком родственном,
филогенетическом отношении между последовательностями 1-3
(формами жизни, от которых они получены) по сравнению
с их отношениями с последовательностью (формой жизни) 4.
Кроме того, если известна дата дивергенции каких-либо после-
довательностей (скажем, последовательностей 1 и 2), то после
установления генетической дистанции между этими последова-
тельностями можно рассчитать среднюю скорость их эволюции
и установить даты дивергенции последовательности 4 от общего
предка группы 1-4.
В простейшем случае для установления эволюционных ди-
станций между двумя последовательностями могут использо-
ваться наблюдаемые различия, т. е. нуклеотиды или амино-
кислоты, неодинаковые у анализируемых последовательностей.
Такие различия могут быть выражены либо как абсолютное
число неодинаковых нуклеотидов или аминокислот, n.,i, либо
как их пропорция в пересчете на длину последовательностей
п, выражаемую числом сравниваемых нуклеотидов или амино-
кислот. Наблюдаемая пропорция нуклеотидов или аминокислот,
неодинаковых у двух последовательностей, и выраженная либо в
частях от единицы, либо в процентах, называется р-дистанцией
(p-distance). Так, в рассматриваемом примере последовательно-
сти 1 и 2 имеют один неодинаковый нуклеотид, а последователь-
ности 1 и 4 — четыре неодинаковых нуклеотида. В пересчете
на длину последовательностей, составляющую 30 нуклеотидов,
р-дистанция между последовательностями 1 и 2 составляет 1 /30,
т. е. 0,033, или 3,33%, а дистанция между последовательностями
1 и 4 — 4/30, т. е. 0,133, или 13,3%. Таким образом, р-дистанция
между двумя последовательностями устанавливается как
а ее дисперсия (variance) равна
п
Отметим, что эволюционная дистанция может пониматься
и как средняя вероятность, средняя частота замены каждого
нуклеотида. Так, можно сказать, что после дивергенции после-
довательностей 1 и 2 от их общего предка средняя наблюдаемая
частота замены каждого нуклеотида составляла 0,033. В среднем
3.1. Наблюдаемые, истинные и расчетные дистанции 63
в каждой из тридцати позиций нуклеотидные замены произо-
шли с частотой 0,033. Можно говорить и о средней эволюцион-
ной дистанции двух последовательностей от их общего предка.
Эта дистанция (не дистанция между последовательностями 1
й 2, но их средняя дистанция от общего предка) составля-
ет 0.033/2 = 0.017 (одна замена между последовательностями
означает, что после дивергенции от общего предка в одной
из последовательностей произошла замена, и она изменилась
на 0,033, в другой последовательности изменений не произошло,
в среднем частота замен после дивергенции составила 0,017).
Кроме понятия различий между последовательностями ис-
пользуется и противоположное понятие — схожести генетиче-
ских последовательностей: две последовательности, различа-
ющиеся между собой на 4%, являются на 96% одинаковыми.
Подчеркнем, что использование понятия «гомология» для опи-
сания схожести последовательностей, употребление выражения
«две последовательности гомологичны на 96%» неправомерно.
Выражение «две последовательности гомологичны» означает
происхождение этих последовательностей от единого общего
предка, и понятие «гомология» количественным не является.
Две последовательности либо гомологичны, либо нет.
Использование наблюдаемых p-дистанций в эволюционном
анализе допустимо в тех случаях, когда число неодинаковых
нуклеотидов или аминокислот в последовательностях невелико,
скажем, менее 1-5%. Однако по ряду причин использование
наблюдаемых дистанций не является оптимальным для уста-
новления истинных эволюционных дистанций. Перечислим ос-
новные ограничения и недостатки использования р-дистанций.
Во-первых, наблюдаемая дистанция не может превышать 1
(100%) в случае, когда все нуклеотиды или аминокислоты у двух
последовательностей различны. Собственно говоря, при анализе
Двух даже эволюционно не связанных между собой нуклео-
тидных последовательностей средняя p-дистанция между ними
не будет превышать 0,75, если частота встречаемости всех
четырех нуклеотидов в обеих последовательностях составляет
25% (таким образом, в 25% случаев нуклеотиды в обеих по-
следовательностях будут случайно совпадать). Во-вторых, од-
н° наблюдаемое различие между двумя последовательностями
Не означает, что это различие являлось результатом только
°Дной замены: могли иметь место множественные замены в этой
п°зиции. Кроме того, отсутствие отличий в каких-либо позициях
йе означает, что в этих позициях не происходили замены: могли
64 Глава 3. Генетические дистанции и эволюционные модели
иметь место обратные замены (частный случай множественных
замен в позиции) или замены в обеих последовательностях. При-
меры таких эволюционных событий, обратных, параллельных
и конвергентных мутаций обсуждены в разд. 1.8 и 1.10. Таким
образом, наблюдаемая дистанция недооценивает истинную ди-
станцию — в реальной эволюционной истории истинное число
замен было, в общем случае, больше наблюдаемого. В-третьих,
известно, что различные типы замен имеют разную вероятность.
Так, например, вероятность транзиций в общем случае — выше
вероятности трансверсий. Таким образом, транзиции в целом
занимают меньше времени, что необходимо учитывать при уста-
новлении эволюционных дистанций. Так, в рассматриваемом
примере последовательность 1 имеет одно отличие и от по-
следовательности 2, и от последовательности 3. Однако разли-
чие между последовательностями 1 и 2 является транзицией,
а различие между последовательностями 1 и 3 — трансверсией.
Поскольку транзиции в целом более вероятны, более часты, чем
трансверсии, т. е. занимают меньше времени, логично считать,
что эволюционная дистанция между последовательностями 1
и 2 меньше, чем между последовательностями 1 и 3, а сами
последовательности 1 и 2 — более родственны друг другу (име-
ют более недавнего общего предка), чем последовательности 1
и 3. В-четвертых, логично считать, и экспериментально пока-
зано, что вероятности (скорость) замен в различных позициях
последовательностей, вообще говоря, могут различаться. Так,
в рассматриваемом примере последовательности имеют много
замен в позиции 6 и мало — в позиции 21. Таким образом, можно
предположить, что замены в позиции 6 более вероятны, требуют
меньше времени, что необходимо учитывать при анализе эво-
люционных дистанций между последовательностями. В-пятых,
важно учитывать нуклеотидный (аминокислотный) состав по-
следовательностей. Перечисление причин, по которым число
наблюдаемых между последовательностями различий неадекват-
но отражает истинные эволюционные дистанции между ними,
может быть продолжено.
Таким образом, наблюдаемая p-дистанция, в общем слу-
чае, не равна (меньше) истинной эволюционной дистанции D,
т. е. неадекватно отражает время эволюции независимых линий,
последовательностей. Поскольку в подавляющем большинстве
случаев истинная эволюционная дистанция неизвестна, для ее
оценки используются расчетные дистанции, основанные на раз-
личных представлениях о частоте различных нуклеотидных
3.2. Эволюционные модели 65
замен (аминокислотных замещений). Математически эти пред-
ставления описываются в различных эволюционных моделях
(evolutionary model). Большинство этих моделей рассматривают
эволюцию нуклеотидных последовательностей.
При переходе к описанию эволюционных моделей, отметим
существенный аспект. Наличие некой нуклеотидной замены
является результатом мутации и ее последующего отбора, за-
крепления. Таким образом, наблюдаемая нуклеотидная замена
является результатом комплексного процесса. Так, большая на-
блюдаемая частота замен, скажем G на А, может быть резуль-
татом как более частых ошибок при репликации, при которых
G меняется на А, так и положительного отбора именно таких
мутаций. При описании моделей эволюции этот комплексный
процесс учитывается как единое целое, без раздельного ана-
лиза его составляющих. В филогенетическом анализе прин-
ципиальным является вопрос о точности установления эволю-
ционной дистанции, точной оценке частот различных типов
замен. Данные о причинах различий частот замен обсуждаются
в разд. 5.2.1.
3.2. Эволюционные модели и дистанции
между нуклеотидными последовательностями
3.2.1. Модель Джукса—Кантора
Исторически первой моделью для расчета эволюционных ди-
станций между последовательностями является предложенная
в 1969 г. модель Джукса—Кантора (Jukes—Cantor model),
JC69 [Jukes and Cantor, 1969]. Модель исходит из того, что
вероятность (частота) замены любого нуклеотида на любой дру-
гой нуклеотид за единицу времени одинакова и составляет а.
Таким образом, модель Джукса—Кантора является однопарамет-
рической. Например, если в рассматриваемой позиции после-
довательности находится нуклеотид А, то за единицу времени
этот нуклеотид может быть заменен на нуклеотид Т, или С,
или G с одинаковой частотой о. Таким образом, частота замены
А на любой другой нуклеотид за единицу времени составляет
За.
Чтобы понять логику, используемую при разработке эволю-
ционных моделей вообще, не ограничимся приведением форму-
Лы для расчета нуклеотидных дистанций по модели Джукса—
Кантора, а рассмотрим эту модель подробно.
5 223Я
бб Глава 3. Генетические дистанции и эволюционные модели
Пусть в момент времени 0 рассматриваемую позицию в по-
следовательности занимает нуклеотид А. Таким образом, веро-
ятность нахождения нуклеотида А в этой позиции в момент вре-
мени 0 равна 1. Поскольку, согласно модели Джукса—Кантора,
вероятность замены этого нуклеотида А на один из трех других
нуклеотидов — Т, С или G — за единицу времени составляет а,
то через единицу времени, в момент времени 1, этот нуклео-
тид А будет заменен на другой нуклеотид с вероятностью За.
Соответственно вероятность нахождения нуклеотида А в этой
позиции в момент времени 1 составит
•Рд(1) = 1 - За.
Таким образом, за первую единицу времени возможны два
сценария эволюции в рассматриваемой позиции: нуклеотид А
мог либо остаться неизменным с вероятностью 1 — За, либо быть
заменен на другой нуклеотид с вероятностью За.
Однако при расчете вероятности нахождения нуклеотида А
в рассматриваемой позиции после второй единицы времени,
в момент времени 2, следует учитывать уже три возможных
сценария. Два первых сценария — те же: если нуклеотид А не
изменился за первую единицу времени, то он, во-первых, мог
остаться неизменным и за вторую единицу времени, а во-вторых,
мог быть заменен. Но, кроме того, следует учитывать и третий
сценарий: если за первую единицу времени нуклеотид А был
заменен на другой нуклеотид, то за вторую единицу времени
в рассматриваемой позиции могла произойти еще одна заме-
на, обратная, и этот измененный за первую единицу времени
нуклеотид мог быть заменен обратно на А. Модель Джукса—
Кантора учитывает этот третий сценарий множественных замен
в одной позиции, и вероятность нахождения нуклеотида А в этой
позиции в момент времени 2 составляет
Рд(2) = (1 — За)Рд(1) + а(1 — Р4(1)).
Вообще, вероятность нахождения нуклеотида А в рассмат-
риваемой позиции в любой момент времени t + 1 определяется
вероятностью нахождения нуклеотида А в рассматриваемой по-
зиции в момент времени t:
f*A(t+i) = (1 - 3a)PA(t) + a(l — Рдр)).
Это уравнение можно переписать и для расчета изменения
вероятности нахождения нуклеотида А (т. е. замены этого нук-
3.2. Эволюционные модели 67
леотида) в рассматриваемой позиции за единицу времени:
АРA(t) — Рд(1+1) — РA(t) = —+ о(1 — Р 4(tj) = — + а.
Понятно, что вероятность нахождения нуклеотида А можно
рассматривать с точки зрения не только дискретного, но и непре-
рывного процесса:
dPА(‘) л о
-^ = -4аРлю + а,
а
р ____ l-1-iP I 4at
PAW - + I Ра(0) - 4 )е
Это уравнение справедливо для любых начальных условий.
Если начальным условием выбрать нахождение нуклеотида А
в рассматриваемой позиции, т. е.
Ра(0) = 1,
то вероятность нахождения нуклеотида А в этой позиции через
время t (т. е. вероятность сохранения нуклеотида, отсутствия
замены) составит
Р — 1 4- ^e~4at
Paw - 4 + 4е •
Если же начальным условием выбрать отсутствие нуклеоти-
да А в рассматриваемой позиции, т. е. Рд(о) = 0» то вероятность
нахождения нуклеотида А в этой позиции через время t составит
Таким образом, модель Джукса—Кантора позволяет устано-
вить вероятность нахождения нуклеотида А в рассматриваемой
позиции последовательности через время t при условии как
нахождения, так и отсутствия нуклеотида А в этой позиции
в момент времени 0. Например, если в момент времени 0
в данной позиции находится нуклеотид Т, то вероятность его
замены на А за время t составит
1 1 c-4at
^TAW - 4 - 4Р
Поскольку модель Джукса—Кантора является однопарамет-
Рической и предполагает, что вероятность замены любого нук-
Леотида на любой другой нуклеотид одинакова, то
^TAW — РCAW ~ РSA{t) — PtCW
68 Глава 3. Генетические дистанции и эволюционные модели
и т. д. Естественно, в таком случае и вероятность сохранения
любого нуклеотида одинакова, т. е.
^AA(t) = Ртт(€) = РCC(t) = PGG(t)-
Соответственно можно рассматривать общую вероятность того,
что за время t начальный нуклеотид i не изменится или изме-
нится на нуклеотид j, соответственно и Ptjftp Очевидно,
что
Р — 1 + p~iat
P"W - 4 + 4е ’
а
Р , , — - — -P~4at
PijW 4 4е
Таким образом, если в момент времени 0 в рассматриваемой
позиции находился нуклеотид г, то с течением времени от нуля
к бесконечности вероятность нахождения нуклеотида г в этой по-
зиции нелинейно снижается от 1 к 0,25. Если же начальный нук-
леотид— не г, то с течением времени вероятность нахождения
нуклеотида i в этой позиции нелинейно повышается от 0 к 0,25.
Таким образом, с течением времени вероятность нахождения
нуклеотида г в рассматриваемой позиции приближается к 0,25
независимо от того, какой нуклеотид находился в этой позиции
изначально. Это справедливо для любого из четырех нуклеоти-
дов. Соответственно, согласно модели Джукса—Кантора, с тече-
нием времени вероятность нахождения любого из четырех нук-
леотидов А, Т, С и G в каждой позиции рассматриваемой после-
довательности стремится к 0,25, а содержание каждого из нук-
леотидов во всей последовательности — к 25%. Таким образом,
модель предсказывает стремление к достижению равновесия
в содержании всех четырех нуклеотидов в последовательности,
или стационарного состояния, что, безусловно, в общем случае
эволюции последовательностей неверно. При эволюции многих
живых систем наблюдается тенденция к предпочтительному
накоплению того или иного нуклеотида в их геномах (скажем,
нуклеотида А), а не тенденция к достижению равновесного
состояния (подробно этот вопрос обсужден в разд. 5.2).
До сих пор мы рассматривали вероятности замен в одной
последовательности с течением времени, сравнивали дочернюю
последовательность с родительской. Однако в практической ра-
боте, как правило, сравнивают между собой дочерние после-
довательности в отсутствие генетических данных об их общем
3.2. Эволюционные модели 69
предке при неизвестной родительской последовательности. При-
веденные выше рассуждения справедливы для каждой из этих
сравниваемых дочерних последовательностей.
Рассмотрим две гомологичные последовательности х и у.
Предположим, что в момент времени 0 эти последовательности
имели последнего общего предка, т. е. были идентичными, после
чего их эволюция шла независимо друг от друга. Понятно, что,
в соответствии с моделью Джукса—Кантора, вышеприведенные
формулы применимы к эволюции каждой из этих последова-
тельностей. Таким образом, если в момент времени 0 общий
предок этих последовательностей имел нуклеотид А в некой
позиции, то для каждой из двух последовательностей х и у, рас-
сматриваемых отдельно, вероятность нахождения нуклеотида А
в этой позиции через время t составит Р4д(/). Таким образом,
вероятность того, что через время t последовательности х и у
будут иметь нуклеотид А в этой позиции, составит РАА^-
Как правило, последовательность общего предка х и у неиз-
вестна. Неизвестно, находился ли в рассматриваемой позиции
общего предка последовательностей х и у нуклеотид А, или
нет. Что известно — это то, что в настоящее время последова-
тельности х и у имеют или не имеют одинаковый нуклеотид А
в рассматриваемой позиции. Последовательности х и у будут
иметь одинаковый нуклеотид А в этой позиции, если общий
предок х к у имел А в этой позиции, и в этой позиции либо
не происходило замен, либо произошли замены и обратные
замены на А, или если общий предок х и у имел другой
нуклеотид в этой позиции, но в обеих последовательностях х
и у произошли одинаковые замены этого нуклеотида на А.
Понятно, что вероятность того, что в обеих последовательностях
произошли одинаковые замены на А, составляет
р2 . р2 I р2
^TA(t) + 'CM(t) + rGA(t)'
Естественно, что аналогичные рассуждения правомерны
Для всех позиций анализируемых последовательностей. Как
отмечалось в разд. 3.1, в эволюционном анализе наблюдаемые
Различия между последовательностями можно представить
и в пересчете на длину последовательностей как пропорцию
одинаковых нуклеотидов. Например, если две сравниваемые
последовательности длиной в 10 нуклеотидов каждая отлича-
ется между собой на один нуклеотид, то можно сказать, что
Для каждой из этих десяти позиций вероятность иметь замену
составляет в среднем 10%, или 0,1. Таким образом, ожидаемая
70 Глава 3. Генетические дистанции и эволюционные модели
вероятность того, что последовательности а: и у будут иметь
одинаковый нуклеотид в некой позиции через время t, может
быть представлена и как пропорция одинаковых нуклеотидов
в последовательностях х и у, 7(t):
At) = PAA(t) + PAT(t) + PAC(t) + PAG(ty
Подставляя значения для каждой из этих вероятностей,
определяемые вышеприведенными формулами для расчета Рщц
и Py(t)» имеем
т ч . 3 -8ot
At) - 4 + 4е
Таким образом, определяет пропорцию одинаковых нук-
леотидов у двух гомологичных последовательностей через вре-
мя t. Понятно, что модель Джукса—Кантора определяет на-
блюдаемую пропорцию различий между последовательностями,
т. е. p-дистанцию, как
р= 1-7(0 = j(l-e-8Qf),
откуда
/ 4 \
8at = — In I 1 — -р j.
Теперь перейдем к собственно эволюционным дистанциям,
т. е. числу замен в пересчете на одну позицию, произошедших
за время независимой эволюции обеих последовательностей по-
сле их дивергенции от общего предка. Модель Джукса—Кантора
исходит из того, что число замен в каждой из последова-
тельностей а: и у за время t составляет 3at. Соответственно
при сравнении последовательностей х и у расчетное число замен,
или дистанция Джукса—Кантора между последовательностями,
составит
Djc6S = 2(3of)-
Подставляя в это выражение значение at, определяемое
предыдущей формулой, имеем формулу для расчета дистанции
Джукса—Кантора между двумя последовательностями:
3 / 4 \
DjCrs = — 41п( 1 ~ зр)'
3.2. Эволюционные модели 71
Дисперсия дистанции Джукса—Кантора определяется форму-
лой
wn
V (Djc6S) -
|р)2п’
где п — длина сравниваемых последовательностей.
Как видно, для расчета дистанции Джукса—Кантора
между двумя последовательностями необходимо знать лишь
^.дистанцию между ними. Собственно говоря, модель Джукса—
Кантора «исправляет» /9-дистанцию, учитывая множественные
замены в одной позиции, поэтому иначе она называется
поправкой или коррекцией Джукса—Кантора (Jukes—Cantor
correction). Эта коррекция возрастает нелинейно по мере
роста p-дистанции. Необходимо также отметить, что ди-
станция Джукса—Кантора может быть рассчитана только
при p-дистанции меньше 0,75, поскольку при р > 0,75 аргумент
логарифма принимает отрицательное значение.
Отметим, что в отличие от p-дистанции, значение которой
по определению не может превышать 1 (или 100%, когда две
сравниваемые последовательности различаются во всех пози-
циях), значение дистанции Джукса—Кантора, а также всех
описываемых далее дистанций, основывающихся на более слож-
ных эволюционных моделях, могут превышать 1, и для них
неприменимо понятие процента.
3.2.2. Модель Кимуры
Модель Кимуры (Kimura model), Кео, исходит из эксперимен-
тальных данных о том, что частота транзиций, в общем случае,
отличается от (выше) частоты трансверсий, что следует учиты-
вать при расчете эволюционных дистанций между последова-
тельностями [Kimura, 1980]. Принимая во внимание различную
частоту транзиций и трансверсий, модель Кимуры предполагает,
что транзиция любого нуклеотида в рассматриваемой позиции
За единицу времени происходит с частотой а, а частота каждой
Из Двух возможных трансверсий — (3 (напомним, что для каж-
дого нуклеотида возможна одна транзиция и две трансверсии,
Разд. 1.6.1). Соответственно модель Кимуры является двупара-
^етрической.
Таким образом, если в момент времени 0 вероятность на-
хождения нуклеотида А в рассматриваемой позиции составляет
то вероятность нахождения нуклеотида А в этой позиции
72 Глава 3. Генетические дистанции и эволюционные модели
в момент времени t + 1 составит
^A(n-i) = (1 — а — 2d)F4(t) + 0Рт(г) + @Pc(t) + aPCW-
Аналогично рассчитываются и вероятности нахождения трех
других нуклеотидов:
Ppt+i) = (! _ а - 20)PT(t) + /ЗРдц) + /3PCW + aPC(t),
Pc\t+i) = (1 - а - 2ft)PC(t) + РРдщ + Жз(1) + aPrft),
PG(t+i) = (1 — а — 20)Pc(f) + /3Pr(t) + 0Pc(t) + aPA(t).
При этом предполагается, что на протяжении эволюции рас-
сматриваемой последовательности частота встречаемости каж-
дого из четырех нуклеотидов одинакова, т. е. составляет 25%
для каждого из них. Поскольку для любого нуклеотида возмож-
на одна транзиция с частотой о и две трансверсии с частотой /3
для каждой из них, то вероятность сохранения каждого из че-
тырех нуклеотидов за время t одинакова, т. е.
PAA(t) = PrT(t) — Pcctt) = PGG(t)-
Обозначая вероятность сохранения нуклеотида как Р„(1)> Для
непрерывного процесса имеем
p..t. = 1 + . le-2(a+0)t
Пг(‘) 4 4 2
Кроме того, для каждого из четырех нуклеотидов частота
транзиций одинакова. Обозначив ее как Ptsip, имеем
р , , - 1 + 1 -2(a+0)t
^s(t)-4+4e 2е
Одинаковы и вероятности каждой из двух возможных транс-
версий для каждого нуклеотида, Ftr(tj:
= J -
При этом
Рщр + fts(t) + 2Ftv^t) = 1.
Таким образом, используемые в модели Кимуры рассуждения
аналогичны тем, которые использованы в модели Джукса—
Кантора, с учетом двух типов замен. Аналогичен и переход от ве-
роятности сохранения или замены нуклеотида в рассматривае-
мой позиции при эволюции одной последовательности к установ-
лению эволюционных дистанций между двумя гомологичными
последовательностями. Опуская математические выкладки этого
3.2. Эволюционные модели 73
перехода, получаем формулу для расчета дистанции Кимуры
между двумя последовательностями:
De80 = ln(l - 2Р - Q) - | ln(l - 2Q).
где Р и Q — наблюдаемые пропорции транзиций и трансверсий,
т,е. отношение наблюдаемых транзиций и трансверсий к наблю-
даемому общему числу замен между двумя последовательностя-
ми соответственно.
Дисперсия дистанции Кимуры определяется формулой
,z/n .CfF + CjO-tCxF + CaQ)2
где
г = 1 г = 1 с = 1
1 1-2F-Q’ 2 1 — 2Q 3 2(С]+С2)’
Подобно модели Джукса—Кантора, использующей наблюда-
емую дистанцию для расчета наиболее вероятной дистанции,
модель Кимуры может быть использована и для расчета ве-
роятного числа транзиций, s, и трансверсий, г, исходя из их
наблюдаемого числа:
s = 1П(1 _ 2Р - Q) + 1 ln(l - 2Q),
г = —1 ln(l — 2Q).
3.2.3. Модель Таджимы—Неи
Таджима и Неи предложили модель TN84 (Tajima-Nei model),
Учитывающую неодинаковую частоту встречаемости каждого
их четырех нуклеотидов при расчете генетических дистанций
Между двумя последовательностями [Tajima and Nei, 1984].
Модель Таджимы—Неи исходит из того, что замены любого
нуклеотида на А происходят с частотой а, любого нуклеотида
на Т — с частотой /3, любого нуклеотида на С — с частотой э,
н любого нуклеотида на G — с частотой 6. В таком случае форму-
Ла Для расчета эволюционных дистанций между нуклеотидными
Последовательностями, согласно модели Таджимы—Неи, может
6ыть записана как
DtNisi = “ЫН fl - tY
\ О)
74 Глава 3. Генетические дистанции и эволюционные модели
где
Здесь дг и gj — частоты встречаемости нуклеотидов г и j
[i.j = A,T,C,G), a Xij—относительная частота встречаемости!
двух нуклеотидов i и j.
3.2.4. Другие эволюционные модели
Как видно из предыдущих разд. 3.2.1-3.2.3, различные моде-
ли, рассматривающие эволюцию нуклеотидных последователь-
ностей, учитывают множественные замены в одной позиции,
а различия между моделями определяются тем, что они исходят
из различных частот, вероятностей разных замен. Так, в модели
Кимуры учитывается различная частота транзиций и транс-
версий, а в модели Таджимы—Неи — частоты замен на любой
из четырех нуклеотидов отличаются друг от друга, но не зависят
от того, какой из трех других нуклеотидов находился в этой
позиции ранее. Предложены и более сложные модели для расче-
та генетических дистанций. Так, Тамура предложил модель Т92
(Tamura model), учитывающую влияние суммарного содержания
нуклеотидов G и С (GC-содержание, разд. 1.7 и 5.2.1), обозначен-
ное как 0, на частоту транзиций и трансверсий [Tamura, 1992]:
DT92 = -20(1-0) Infl- Р_ _qV1(1-20(1-0)) ln(l-2Q).
\ ZkJI -1 Vv I / Zu
В более сложных моделях учитывается и возможное влияние I
содержания каждого из четырех нуклеотидов на частоту тран-
зиций и трансверсий — модель Хазегавы с соавт. (Hasegawa et al.
model) [Hasegawa et al., 1985], и возможность неодинако-
вой частоты транзиций для пуринов и пиримидинов — модель
Тамуры—Неи (Tamura—Nei model) [Tamura and Net, 1993].
Более того, можно рассмотреть и общую модель, в которой!
частоты замен любого нуклеотида в рассматриваемой пози-
ции на любой другой нуклеотид отличаются друг от друга,
т. е. 12-параметрическую модель, в которой каждый из четырех
нуклеотидов может быть заменен на три других нуклеотида,!
и все эти замены проходят с различными частотами (4 х 3 = 12
частот-параметров). Для всех обсужденных моделей, предпо-
лагаемые ими частоты замен каждого нуклеотида на каждый
3.2. Эволюционные модели 75
Таблица 3.1
Частоты замен нуклеотидов в различных эволюционных моделях
Нуклеотид
изначально после замены
А T c G
1. Модель Джукса—1 Еантора
А — a a a
Т a — a a
" С a a — a
G a a a —
2. Модель Кимуры
А — 0 0 a
Т /3 — a 0
С 0 a — 0
G a 0 0 —
3. Модель Таджимы—Неи
А — 0 7 6
Т a — 7 6
С a 0 — 6
G a 0 7 —
4. Модель Тамуры
А — (i-e)/3 0/3 <00
Т — 0a Q0
С (l-O)a — 0a
G (1- 0)a (1-0)3 Q0 —
а и (3 — частоты соответственно транзиций и трансверсий, а О — GC-содержание
5. Модель Хазегавы и др.
А — 9T0 gc0 gca
Т длР — gca 9G0
С 9A0 gra — gc0
G 9A<* 9T0 9C0 —
Q и [3 — частоты соответственно транзиций и трансверсий, gt — Содержание нуклеотида г (i = A,T,C,G)
6. Модель Тамуры—Неи
А — 9T0 9C0 geon
т 9A0 — gca2 9G0
9A0 9tO12 — 9G0
G 9AQ1 9T0 9C0 —
Qi и о2 — частоты транзиций между соответственно пуринами и пи- римидинами, (3 — частота трансверсий, gt — содержание нуклеотида (7)
76 Глава 3. Генетические дистанции и эволюционные модели
другой нуклеотид, как и частоты сохранения нуклеотида, при-
ведены в табл. 3.1. Принципы, которыми следуют руководство-
ваться при выборе модели для анализа нуклеотидных последо-
вательностей в конкретных практических случаях, обсуждены
в разд. 3.2.6.
3.2.5. Гамма-дистанции
Рассмотренные нами до сих пор модели исходят из того, что
частоты замен являются одинаковыми для всех позиций рас-
сматриваемых последовательностей. Естественно, это предполо-
жение может, вообще говоря, и не соблюдаться. Рассмотрим
пример с последовательностями 1-5:
111111
123456789012345
1 ATACGAGAAAACGCA
2 G-------------
3 т-------------
4 с-------------
5 ---------------С
Как видно, последовательности 1-5 отличаются друг от друга
на один нуклеотид — либо в позиции 6, либо в позиции 15. При
этом в обеих этих позициях последовательности 1 находится
нуклеотид А. Рассмотренные нами ранее модели исходят из то-
го, что частоты замен нуклеотида на другой нуклеотид могут
и различаться в зависимости от заменяемого и заменяющего
нуклеотида, но эти частоты одинаковы для любой позиции.1
Однако, рассматривая данный пример, можно предположить, что
при эволюции последовательностей 1-5 замены в позиции 6
происходили чаще, требовали меньше времени, чем замены
в позиции 15. Соответственно можно предположить, что эволю-
ционная дистанция между последовательностями 1 и 5 больше
дистанции между последовательностью 1 и последовательностя-
ми 2-4, несмотря на то, что последовательность 1 отличается
от последовательностей 2-4 и 5 на один нуклеотид.
Таким образом, при расчете эволюционных дистанций можно
учитывать не только разную частоту разных типов замен, но
и разную вероятность замен в разных позициях. Так, можно
ожидать, что частота замен в участках последовательности’
кодирующих антигенные детерминанты, будет, вообще говоре
3.2. Эволюционные модели 77
Bbinie (замены будут занимать меньше времени), чем в участ-
ках- не подвергающихся иммунному давлению. Это предполо-
жение подтверждено и экспериментально. В кодирующих по-
следовательностях замены в первой и второй позиции кодона
в большинстве случаев будут несинонимичными, в то время как
замены в третьей позиции кодона будут в большинстве случаев
синонимичными. Экспериментальные данные показывают, что
в большинстве случаев замены в третьей позиции кодона проис-
ходят с частотой, в разы и десятки раз превышающий частоту
замен в первой и второй позиции кодона. Причины различий в
частотах синонимичных и несинонимичных заменах обсуждают-
ся в разд. 3.3 и 5.2. Можно привести и другие примеры неравной
частоты замен для различных позиций последовательности.
Различие частот замен в различных позициях последователь-
ностей можно учесть в любой из обсужденных нами моделей.
Для того, чтобы продемонстрировать, как это различие частот
замен влияет на расчет эволюционных дистанций, рассмотрим
простейший случай для модели Джукса—Кантора. Предполо-
жим, что в рассматриваемой нами паре последовательностей
есть два класса позиций, частоты замен любого нуклеотида
на любой другой нуклеотид в которых различны и составляют
О] и «2. а пропорции позиций этих классов в последовательности
составляют соответственно Д и /2- В таком случае, по модели
Джукса—Кантора, средняя пропорция одинаковых нуклеотидов
в этих двух последовательностях через время t будет составлять
I = flh + flh,
где Д и I2 — пропорции одинаковых нуклеотидов первого и вто-
рого класса, соответственно рассчитываемые путем подстановки
°i и о2 вместо а в формулы модели Джукса—Кантора, приве-
денные в разд. 3.2.1.
Аналогично может быть переписана и формула для расчета
генетических дистанций между последовательностями.
Очевидно, что графики таких функции будут отличаться
От графиков функций, в которой частота замен во всех позициях
Одинакова. Для демонстрации этого предположим, что в анали-
зируемых последовательностях в 5% позиций не происходило
замен в силу, например, функциональной значимости этих пози-
ций (т. е МуТацИИ в этих позициях происходили, но приводили
К потере белком своих функций и удалению этих мутаций
популяции). Таким образом, для пропорции нуклеотидных
п°зиций Д = 0.05 частота замен а будет равна 0. Как показано
78 Глава 3. Генетические дистанции и эволюционные модели
при обсуждении модели Джукса—Кантора, при стремлении t
к бесконечности в случае равной частоты замен во всех позици-1
ях I стремится к 0,25. Очевидно, что в рассматриваемом примере
с о — 0 для 5% позиций, пропорция одинаковых нуклеотидов
будет стремиться к 0,25 только в остальных 95% позиций, а нук-
леотиды в этих 5% позиций с течением времени по определению
будут оставаться одинаковыми. Таким образом, для всех пози- j
ций суммарно пропорция одинаковых нуклеотидов I будет стре-
миться не к 0,25, а к 0.05 х 1 + 0.95 х 0.25 = 0.2875. Понятно, что
в реальных случаях вариации в частоте замен в различных по-
зицях могут описываться и более сложно. В общем случае речь
идет не о нескольких классах позиций с различной частотой
замен, но о неком вероятностном распределении частот замен
в разных позициях. Предполагается, и для ряда последователь-
ностей показано, что частота замен в различных позициях ва-
рьирует согласно Г(гамма)-распределению (Г-distribution) [Uzzell
and Corbin, 1971; Jin and Nei, 1990; Tamura and Nei, 1993;
Wakeley, 1993]. Изучение этого распределения является пред-'
метом высшей математики. В рамках нашей книги следует
сказать, что форма Г-распределения определяется параметром а
(не путать с ранее введенным для определения частоты замен
параметром о). При а < 1, Г-распределение принимает форму
экспоненциального распределения. Переводя это в вариации
частот замен, можно говорить о случае, когда для значительной
пропорции позиций частоты замен близки к 0. При увеличе-
нии а, Г-распределение может быть приблизительно описано
нормальным распределением. Таким образом, можно говорить
о случае, когда для значительной пропорции позиций частота |
замен близка к средней, но есть некоторая пропорция позиций, I
частоты замен в которых значительно больше (изменчивые,
вариабельные позиции) и значительно меньше (консервативные
позиции) средней частоты замен.
Если в рассматриваемой группе последовательностей ча-
стоты замен в различных позициях варьируют согласно
Г-распределению, то параметр а может быть учтен при рас-
чете генетических дистанций между последовательностями со-
гласно описанным выше моделям. В таких случаях говорят |
о Г-дистанции для модели Джукса—Кантора, рассчитываемой
по формуле
3.2. Эволюционные модели 79
о Г-ДистанЦии Для модели Кимуры, рассчитываемой по формуле
D = | ((1 - 2Р - Q)-^a + |(1 - 2Q)-V“ - 0.
и т. д-
Установление значения параметра а для анализируемой груп-
пы последовательностей является отдельной задачей. Более то-
го, вопрос о том, варьирует ли частота замен во всех слу-
чаях, согласно Г-распределению, остается в значительной сте-
пени открытым. Ряд распространенных компьютерных про-
грамм для филогенетического анализа (например, программа
MEGA, разд. 6.5) предоставляет исследователю возможность рас-
считывать Г-дистанции и использовать эти дистанции в фи-
логенетическом анализе, но не включают в себя алгоритмов
для расчета а. Вместо этого можно либо рассчитать значе-
ние а с помощью специальных программ — например, програм-
мы GZ-GAMMA (разд. 6.6), - либо использовать значение а, рас-
считанное ранее для родственной группы последовательностей.
Можно и просто использовать значение а, исходя из общих
сведений о вариации частот замен в различных позициях ана-
лизируемой группы последовательностей. Для простоты можно
использовать одно из трех значений о: 0,5, 1 или 2, в зависи-
мости от того, предполагается ли исследователем, что различия
в частотах замен в различных позициях являются большими,
умеренными или небольшими соответственно.
3.2.6. Сравнение различных моделей
Несмотря на обилие эволюционных моделей, не существует
модели, которая была бы оптимальна для любого случая, оди-
наково хороша при анализе любой группы последовательностей.
Вообще говоря, более сложные модели описывают эксперимен-
тальные данные лучше. Однако это не означает, что на практике
всегда следует отдавать предпочтение более сложным моделям.
Прежде всего в ряде случаев более простые и более слож-
Нь’е модели дают практически одинаковые результаты. Так,
пРи небольшой наблюдаемой дистанции между последователь-
н°стями (скажем, при р < 0.05), модель Джукса—Кантора дает
фактически одинаковые с моделью Кимуры и Г-дистанциями
Результаты, независимо от различия частот транзиций и транс-
ВеРсий и вариации в частоте замен между позициями. В то же
вРемя более сложные модели требуют расчета большего числа
°аРаметров, каждый из которых рассчитывается со стандартным
80 Глава 3. Генетические дистанции и эволюционные модели
отклонением, что ведет к возрастанию итогового стандартного и
отклонения при расчете дистанции. Малый размер эксперимен-
тальной выборки (относительно короткие последовательности)
гораздо сильнее влияет на более сложные модели (ошибка вы-
борочного исследования). Очевидно, что дополнительные пред,
положения могут вызывать и увеличение ошибки при расче-
тах.
Сравнению различных методов расчета генетических дистан-
ций и разработке статистических подходов к выбору эволюцион-
ной модели с помощью компьютерных симуляций был посвящен
ряд работ [Goldman, 1993; Zharkikh, 1994; Posada and Crandall,
2001b]. При таком подходе компьютерная программа генерирует
некую последовательность и случайным образом производит
в ней замены в соответствии с заданным исследователем алго-
ритмом: определенным отношением числа транзиций и транс-I
версий, разной вероятностью замен для различных нуклеоти- II
дов и т.д. Поскольку при такой симуляции известно число замен,
произведенных компьютерной программой, известна истинная I
эволюционная дистанция, то существует возможность сравнить,
насколько точно различные эволюционные модели ее устанавли-
вают.
Обобщая эти работы, отметим, что на практике при выборе!
эволюционной модели можно руководствоваться следующими!
принципами:
— если различные модели дают приблизительно одинаковые
результаты, следует использовать более простую модель,
имеющую меньшую дисперсию;
— при дистанциях до 0,05 можно использовать модель
Джукса—Кантора;
— при возрастании дистанции до 0,3, можно использовать
модель Джукса—Кантора при небольшом отношении чис- ]
ла транзиций и трансверсий (скажем, менее 2). Если
отношение транзиций и трансверсий больше 2, и число
сравниваемых нуклеотидов велико, то следует использо-
вать модель Кимуры;
— если дистанции выше, а содержание четырех нуклеотидов
значительно отличается от 25%, при небольшом отноше-
нии числа транзиций и трансверсий следует использовать
модель Таджимы—Неи, при большом — модели ТамурьИ
или Тамуры—Неи;
— при дистанциях выше 0,3 и вариациях в частоте замей
в различных позициях, следует использовать Г-дистанциШ ।
3.3. Синонимичные и несинонимичные дистанции 81
— хорошим правилом является использовать несколько раз-
личных моделей, и в случае, если их результаты значи-
тельно отличаются друг от друга — установить причину
этого.
При возрастании дистанции выше 1, т. е. когда в каждой
позиции в среднем произошла более чем одна замена, ни одна
из моделей не будет давать надежных результатов. В таких
случаях говорят о влиянии эффекта насыщения (saturation)
нуклеотидных замен. Собственно, влияние этого эффекта начи-
нает сказываться уже при меньших нуклеотидных дистанциях
между последовательностями. Это будет связано и с трудностя-
ми (ненадежностью) выравнивания таких последовательностей,
и с большим стандартным отклонением при расчете дистанции.
В таких случаях для установления эволюционных отношений
между формами жизни следует использовать менее изменчивые
участки генома, или использовать анализ либо аминокислотных
последовательностей, либо несинонимичных позиций.
Разработаны и математические подходы, позволяющие оце-
нить пригодность той или иной эволюционной модели для ана-
лиза конкретной группы последовательностей и установить
параметры этой модели. Такие подходы основаны, в част-
ности, на статистическом сравнении функций правдоподобия
(разд. 4.3.3 и 5.3.3) различных эволюционных моделей, и вклю-
чены, например, в компьютерные программы PAML и PAUP*
(разд. 6.5).
3.3. Синонимичные и несинонимичные дистанции
и их отношение
Рассмотренные нами в предыдущих разделах эволюционные
модели применимы для анализа как кодирующих, так и некоди-
РУющих нуклеотидных последовательностей. Для кодирующих
последовательностей особый интерес представляет раздельный
анализ синонимичных и несинонимичных позиций, синонимич-
ных и несинонимичных замен (разд. 1.6.2).
При репликации кодирующего участка генома ошибки в сино-
нимичных и несинонимичных позициях равновероятны. Однако,
п°скольку синонимичные замены не ведут к замещению амино-
кислот, т. е. не изменяют структуру белка, то они не подвер-
гаются влиянию факторов естественного отбора, действующих
На Уровне функции белка. Таким образом, синонимичные за-
МеНы, как правило, подвергаются гораздо меньшему давлению
*’"2238
82 Глава 3. Генетические дистанции и эволюционные модели
отрицательного отбора, в результате чего их число выше числа
несинонимичных замен. Однако в ряде случаев, как, например,
для антигенных детерминант, число несинонимичных замен
превышает число синонимичных. Это связано с положительным
отбором аминокислотных замещений, ведущим к изменению
антигенной детерминанты и ускользанию от иммунного ответа.
Отношение числа несинонимичных и синонимичных замен,
несинонимичных и синонимичных дистанций, Dn/Ds, является
важной характеристикой движущих факторов эволюции анали-
зируемого участка генома. Если это отношение (статистически
достоверно) ниже единицы, т. е. число несинонимичных замен
меньше числа синонимичных замен, то говорят, что в анализи-
руемом участке генома доминирует отрицательный отбор. При
отношении Dn/Da > 1 говорят, что доминирует положительный
отбор. Часто используется и обратная величина, Ds/Dn.
Отметим, что и при Dn/Ds < 1 не исключено, что в отдельных
участках, отдельных позициях анализируемых последовательно-
стей доминирует положительный отбор. Такой анализ для каж-
дой позиции может быть проведен с помощью, например, гро-
граммы PAML (разд. 6.5).
Поскольку большинство замен в первой и второй позициях
кодона — замены несинонимичные, а большинство замен в тре-
тьей позиции кодона — синонимичные (разд. 1.6.2, табл. 1.4),
то самым простым способом оценки не синонимичных и си-
нонимичных дистанций между последовательностями является
раздельный подсчет замен в первой-второй и третьей позициях!
соответственно. Естественно, что такая оценка будет прибли-1
зительной. Для более точной оценки, в простейшем случае,
при анализе синонимичных и несинонимичных дистанций могут
использоваться наблюдаемые дистанции, ps и рп, т. е. отношение
числа синонимичных и несинонимичных замен, и n,i, к числу]
синонимичных и несинонимичных позиций, s и п соответствен-
но. Этот метод был предложен Неи и Годжобори (Nei—Gojobori
method), NGgc [Nei and Gojobori, 1986]. Отметим, что число
синонимичных и несинонимичных позиций в последовательно-
сти должно быть предварительно рассчитано, оно может быть]
и дробным. Так, например, в кодоне АТТ (изолейцин) первая
и вторая позиции являются несинонимичными, поскольку любая
замена в этих позициях приводит к изменению аминокислоты,
а третья позиция является несинонимичной на одну треть:
из трех возможных в этой позиции замен, замены Т на А или
С не будут изменять аминокислоту, а замена Т на G приведет
3.3. Синонимичные и несинонимичные дистанции 83
К замещению аминокислоты. Сумма s и п будет равна числу
нуклеотидов в рассматриваемой последовательности, при этом
п, как правило, много больше s.
Понятно, что если анализируемые последовательности
в неком кодоне имеют одно отличие, скажем, АТА и АТТ,
то можно достаточно уверенно считать, что наиболее вероятно
в эволюции этих последовательностей имела место одна замена,
и она была синонимичной. Если же таких различий в одном
кодоне больше, скажем, анализируемые последовательности
имеют в некой позиции кодоны АТА и AGG, то и здесь
можно с высокой степенью уверенности говорить о числе
замен: наиболее вероятно, их было две. Однако были ли они
синонимичными или несинонимичными? Поскольку замен две,
то возможны два сценария: либо сначала произошла замена во
второй позиции, а потом в третьей, либо наоборот. В первом
случае имеем замены
АТА —> AGA —» AGG. т. е. изолейцин —» аргинин —» аргинин,
одна несинонимичная и одна синонимичная замена. Во втором
случае имеем
АТА —» ATG —» AGG. т. е. изолейцин —> метионин —> аргинин,
две несинонимичные замены. Понятно, что в общем случае нет
критериев для определения того, какой из двух возможных
сценариев имел место. Поскольку в данном случае равноверо-
ятными были два сценария — либо одна несинонимичная и одна
синонимичная, либо две несинонимичные замены, то логично
считать, что в среднем в этом кодоне произошло 1,5 несинони-
мичных и 0,5 синонимичных замен.
Если же среди возможных сценариев замен в кодоне есть
такой, который подразумевает в качестве промежуточного этапа
появление стоп-кодона в этой позиции, то такой сценарий исклю-
чается из анализа. Подразумевается, что такой сценарий приво-
дил бы к потере кодируемым белком его функции, и потому —
Невозможен. Рассмотрим пример, в котором в анализируемой по-
зиции двух последовательностей находятся кодоны ТАТ и САА.
И в этом случае можно предполагать две замены в этом кодоне,
н его первой и третьей позициях, и два сценария эволюции: либо
сначала произошла замена в первой позиции, а потом в третьей,
•«ибо наоборот. В первом случае имеем
ТАТ —> CAT —> САА. т. е. тирозин —> гистидин —> глицин,
84 Глава 3. Генетические дистанции и эволюционные модели
две несинонимичные замены, во втором случае имеем
ТАТ —» ТАА —> САА. т. е. тирозин —> стоп-кодон —> глицин.
Поскольку второй сценарий приводил бы к преждевременной
терминации трансляции й, вероятно, нефункциональному белку,
то такой сценарий исключается из рассмотрения, и наблюдаемое
различие между кодонами расценивается как результат двух
несинонимичных замен.
Используя модель Джукса—Кантора, можно учесть и множе-
ственные замены в одной позиции:
Ds =
„ 3, ( 4
Dn = — — hi I 1 — -рп
1 у о
Существуют и другие методы для расчета синонимичных
и несинонимичных дистанций. Модифицированный метод
Неи-Годжобори учитывает неодинаковую частоту транзиций
и трансверсий. Метод Ли—By—Луо (Li—Wu—Luo method)
относит каждую позицию последовательности по ее вырож-
денности к одному из трех классов: 0-кратно вырожденные
(все три возможные замены в этой позиции будут несино-
нимичными), 2-кратно вырожденные (одна или две замены
будут синонимичными) и 4-кратно вырожденные (все три
замены будут синонимичными) позиции [Li et al., 1985].
Все 0-кратно вырожденные позиции и две трети 2-кратно
вырожденных позиций учитываются как несинонимичные,
треть 2-кратно вырожденных позиций и все 4-кратно вы-
рожденные позиции — как синонимичные. Наблюдаемые тран-
зиции и трансверсии учитываются для каждого из трех
классов позиций отдельно. Развитием метода Ли—By—Луо
является метод Памило—Бианчи—Ли (Pamilo—Bianchi—Li
method), отличающийся подходом к учету транзиций и транс-
версий [Ы, 1993; Pamilo and Bianchi, 1993]. Предложены также
и другие модификации метода Ли—By—Луо [Сотегоп, 1995].
Следует также отметить, что из-за большого стандартного от-
клонения ни один из существующих методов не дает надежных
результатов при ps и р„, превышающих 0,4.
При подсчете среднего отношения Dn/Ds для группы после-
довательностей можно использовать два алгоритма. В первом
случае можно подсчитать отношения Dn/Ds для каждой пари
3.4. Аминокислотные дистанции 85
последовательностей группы, а их среднюю величину опреде-
лить как среднее отношение для группы. Во втором случае
можно подсчитать отдельно Dn и Ds для каждой пары после-
довательностей группы, найти средние Dtl и Ds для группы,
и их отношение определить как среднее отношение для группы.
Второй алгоритм считается более аккуратным. В частности,
он учитывает и те попарные сравнения последовательностей,
для которых D.o = О (первый алгоритм не учитывает такие
сравнения — деление на О).
3.4. Аминокислотные дистанции, матрицы
вероятностей аминокислотных замещений
При разработке методов установления аминокислотных дистан-
ций используются рассуждения, во многом аналогичные тако-
вым для нуклеотидных дистанций, с учетом того, что конкрет-
ную позицию в последовательности может занимать одна из
20 аминокислот. Так, можно говорить о наблюдаемой аминокис-
лотной дистанции, p-дистанции, т. е. пропорции неодинаковых
аминокислот, о дистанциях истинной и расчетных.
При учете множественных замещений в одной позиции в про-
стейшем случае предполагают, что число замещений для раз-
личных позиций варьирует согласно распределению Пуассона.
Такая дистанция называется исправленной по Пуассону (Poisson-
corrected):
D = — 1п(1 — р).
Можно учитывать и различия в частоте замещений между
различными позициями, Г-дистанцию [Net et al., 1976] —
D = a((l-p)-1/“-l),
и различную частоту встречаемости различных аминокислот
ь анализируемых последовательностях. В этом случае говорят
° дистанции, основанной на модели равного вклада (equal input
model):
£> = -Mn(l-|Y b=l-^gi2,
' ' i
где g, — частота аминокислоты i.
Для установления аминокислотных дистанций широко ис-
пользуются и подходы, основанные на матрицах вероятностей
86 Глава 3. Генетические дистанции и эволюционные модели
конкретных аминокислотных замещений. Эти подходы основаны
на экспериментальных наблюдениях и рассуждениях о том, что
для каждой конкретной аминокислоты вероятность (частота) как
ее сохранения, так и замещения каждой другой аминокислотой
неодинакова. Так, гидрофобные аминокислоты более вероятно
будут замещаться гидрофобными, поскольку замещение гидро-
фильными аминокислотами может вести к нарушению функци-
онирования белка. Цистеин часто участвует в создании дисуль-
фидного мостика, что важно для поддержания структуры белка,
поэтому его замещение любой другой аминокислотой — малове-
роятно. Можно исходить и из различий в кодонах, кодирующих
аминокислоты. Так, замещение метионина (ATG) триптофаном
(TGG) требует двух конкретных нуклеотидных замен в первой
и второй позициях, а замещение метионина изолейцином (АТТ,
АТА, АТС) — одной замены в третьей позиции, при этом —
любой замены. Соответственно логично считать, что второе
замещение будет происходить чаще.
Таким образом, исходя из различных рассуждений и/или экс-
периментальных наблюдений, для каждой аминокислоты можно
оценить вероятность ее сохранения или замещения каждой дру-
гой аминокислотой в различных группах гомологичных белков.
Соответственно каждому событию (сохранение аминокислоты
или ее замещение каждой другой аминокислотой) можно при-
своить определенные значения (положительные или штрафы),
в зависимости от вероятности этих событий. Эти значения
можно представить в виде таблицы, матрицы и использовать
при расчете аминокислотных дистанций. При таком подходе
менее вероятные (менее частые) замещения будут учитывать-
ся как значительно больше увеличивающие дистанцию между
последовательностями, чем более вероятные замещения.
Исторически первыми матрицами, основанными на вероят-
ностях замещений аминокислот, были матрицы класса РАМ
(point accepted mutations), рассчитанные Дэйхофф на основе
анализа частоты различных замещений в близкородственных
последовательностях [Dayhoff, 1972; Dayhoff, 1978]. Эти мат-
рицы отражают вероятности сохранения каждой аминокисло-
ты и ее замещения каждой другой аминокислотой в случа-
ях, если замещается 1 из 100 (РАМ1), 10 из 100 (РАМ10).
50 из 100 (РАМ50) аминокислот и т. д. Дэйхофф рассчитала
матрицы до РАМ250, РАМ250 = (РАМ1)250 (естественно, что
при учете множественных замещений в одной позиции на 100
аминокислот приходится более 100 замещений). В настоящей
3.4. Аминокислотные дистанции 87
время эти матрицы широко используются, как и матрицы
более высокого порядка (скажем, РАМ350). Отметим, что на-
дежность матриц столь высокого порядка подвергается сомне-
ниям.
Используемый в расчетах матрицы математический аппарат
достаточно прост, но в рамках нашей книги ограничимся упоми-
нанием того, что вероятности замещений в такой матрице вы-
ражаются с помощью методов логистической регрессии. Вероят-
ность замещения аминокислоты г аминокислотой j оценивается
по значению (log-odds score) StJ, выражаемому как
8ц = log
Мд
PiPj'
где Pi и pj — частоты аминокислот i и j, a —вероятность
замещения аминокислоты i аминокислотой j.
Таблица 3.2
Пример матрицы РАМ — РАМ250
А R N D С Q Е G н I L К М F Р S Т W Y V *
А 2 —2 0 0 —2 0 0 1 -1 -1 —2 -1 -1 -3 1 1 1 -6 -3 0 -8
R 6 0 -1 -4 1 -1 -3 2 —2 -3 3 0 —4 0 0 -1 2 —4 —2 -8
N 2 2 —4 1 1 0 2 —2 -3 1 —2 -3 0 1 0 —4 —2 —2 -8
D 4 -5 2 3 1 1 —2 —4 0 -3 -6 -1 0 0 —7 —4 —2 -8
С 12 -5 -5 -3 -3 —2 -6 -5 -5 —4 -3 0 —2 -8 0 —2 -8
Q 4 2 -1 3 —2 —2 1 -1 -5 0 -1 -1 -5 —4 —2 -8
Е 4 0 1 —2 -3 0 —2 -5 -1 0 0 —7 —4 —2 -8
G 5 —2 -3 —4 —2 -3 -5 0 1 0 —7 -5 -1 -8
Н 6 —2 —2 0 —2 —2 0 -1 -1 -3 0 —2 -8
I 5 2 —2 2 1 —2 -1 0 -5 -1 4 -8
L 6 -3 4 2 -3 -3 —2 —2 -1 2 -8
К 5 0 -5 -1 0 0 -3 —4 —2 -8
М 6 0 —2 —2 -1 —4 —2 2 -8
F 9 -5 -3 -3 0 7 -1 -8
Р 6 1 0 -6 -5 -1 -8
S 2 1 —2 -3 -1 -8
т 3 -5 -3 0 -8
W 17 0 -6 -8
Y 10 —2 -8
у 4 -8
1
Примечание: ’ — стоп-кодон.
88 Глава 3. Генетические дистанции и эволюционные модели
В табл. 3.2 приведен пример матрицы РАМ — матрица
РАМ250. Как видно, вероятности сохранения триптофана и ци-
стеина велики для этих событий, значения составляют 17 и 12
соответственно, а вероятность сохранения аланина ниже — зна-
чение 2. Аланин будет равновероятно замещен глицином, проли-
ном, серином или треонином (в каждом случае — значение 1).
Матрицы РАМ можно использовать для расчета эволюци-
онных дистанций между аминокислотными последовательно-
стями— РАМ-дистанции. Особенное распространение основан-
ные на матрицах вероятности замещений подходы получили
при разработке алгоритмов выравнивания последовательностей,
рассмотренных в разд. 2.3 [Altschul, 1991].
Использованный Дэйхофф при разработке матриц клас-
са РАМ методологический подход сравнения близкородствен-
ных последовательностей не всегда оптимален при анализе
не близкородственных последовательностей. Эта проблема ре-
шается в матрицах класса BLOSUM (block substitution matrix)
Хеникоффа—Хеникофф [Henikoff and Henikoff, 1992]. Разработ-
ка матриц этого класса основывается на рассмотрении только
консервативных участков (блоков) не близкородственных после-
довательностей. Эти участки расцениваются как функциональ-
но значимые. Используются матрицы BLOSUM62, BLOSUM50,
BLOSUM30 и др. (в отличие от матриц класса РАМ, в матрицах
класса BLOSUM большее число соответствует большей схоже-
сти последовательностей) [Eddy, 2004d].
Матрицы классов РАМ и BLOSUM являются наибо-
лее часто используемыми. Кроме них предложены матри-
цы JTT [Jones et al., 1992а], Gonnet [Gonnet et al., 1992],
WAG [Whelan and Goldman, 2001] и др.
3.5. Учет делеций и отсутствующей информации
Наряду с заменами, вставки и делеции являются эволюционны-
ми событиями. Соответственно их можно учитывать в формулах
для расчета генетических дистанций [Saitou and Ueda, 1994],
в филогенетическом анализе вообще [Diallo et al., 2007]. Однако
в отличие от нуклеотидных замен сколь-нибудь надежная ко-
личественная оценка вероятностей вставок и делеций, в общем
случае, весьма затруднена, если вообще возможна. Поэтому, как
правило, при филогенетическом анализе не учитывают присут-
ствующие в последовательностях пробелы, и анализируют толь-
ко совпадающие и несовпадающие нуклеотиды (аминокислоты).
3.5. Учет делеций и отсутствующей информации 89
диалогично подходят и к участкам, последовательность нуклео-
тидов (аминокислот) которых неизвестна (скажем, не могла быть
определена из-за экспериментальных проблем). Собственно, та-
кие участки, называемые отсутствующей информацией (missing
information), являются экспериментальными пробелами.
При анализе нескольких последовательностей существуют
два подхода к исключению пробелов: их полное или попарное
исключение (complete or pairwise gap deletion). Продемонстриру-
ем разницу между этими подходами на примере анализа трех
последовательностей:
111111
123456789012345
1 GATGTTACTTGGCAT
2 AACGCTACTTAGCTT
3 G777TTACTTGG---
Как видно, у последовательности 3 отсутствует информация
о нуклеотидах в позициях 2-4, и имеется пробел в позициях
13-15. При полном исключении пробелов позиции 2-4 и 13-15
не будут учитываться при попарных сравнениях любых по-
следовательностей группы, в том числе и при установлении
генетических дистанций между последовательностями 1 и 2.
При попарном исключении пробелов позиции 2-4 и 13-15 будут
учитываться при установлении дистанций между последователь-
ностями 1 и 2 и не будут учитываться при сравнении последо-
вательностей 1 и 2 с последовательностью 3. Преимуществом
попарного исключения пробелов является то, что этот подход
более полно учитывает имеющуюся генетическую информацию.
Однако при этом анализ различных последовательностей ведется
на различных участках, что можно считать недостатком такого
подхода.
Глава 4
ФИЛОГЕНЕТИЧЕСКИЙ АНАЛИЗ
4.1. Филогенетические деревья
Все формы жизни на Земле, как существующие, так и вымер-
шие, имеют общее происхождение, и их эволюционная исто-
рия— это порядок их дивергенций от общих предков (рис. 4.1).
Чем менее давно две формы жизни дивергировали от общего
предка — тем они более родственны между собой.
Задачей филогенетического анализа является установление,
реконструкция эволюционной истории — родственных связей,
отношений между формами жизни — и датирование эволюци-
онных событий, моментов дивергенций. В филогенетических
исследованиях эволюционные отношения между формами жизни
представляют в виде филогенетических, или эволюционных,
деревьев (phylogenetic, evolutionary trees).
Структура филогенетического дерева представлена на
рис. 4.2, а. Филогенетическое дерево состоит из внутренних
и внешних ветвей (branches), узлов (nodes), и, если иссле-
дователем выбрана соответствующая опция, — корня (root)
(рис. 4.2, а, б). Порядок всех ветвей дерева называют его тополо-
гией (topology). Внутренние ветви соединяют внутренние узлы,
внешние ветви ведут непосредственно к объектам исследования
(говорят также о внешних узлах, или листьях дерева, leaves).
Объектами филогенетического исследования могут быть гены
или их участки, нуклеотидные или аминокислотные последова-
тельности, организмы, популяции, индивидуумы, штаммы виру-
сов и т. д. Эти объекты исследования называют оперативными
таксономическими единицами, OTU (operational taxonomic units).
Деревья с корнем отражают направление эволюции, порядок
почкования, ответвления (branching) различных эволюционных
линий (см. рис. 4.2, а). Деревья без корня показывают родствен-
ные отношения между анализируемыми последовательностями,
но не направление эволюции (см. рис. 4.2,6).
Отметим, что направление от корня к листьям дерева есть
направление по нисходящей линии (аналогично генеалогическо-
му дереву, от предков к потомкам). Направление от листьев
к корню — восходящая линия от потомков к предкам.
4.1. Филогенетические деревья 91
PEDIGREE OF MAN.
Hoofed Animals
(Ungulata)
| MAN |
| Gorillgy^fO^ang |
| Chimpanzeejx^^g^ Gibbon]
| Ape-Men] Д
les | for
| Whal'e'spfo^' Sloth^
[Pouched Animals!
Semi-Apes (Leinuroiden)
Beasts of Prey|
Primitive Mammals
(Promammalia)
S
[Beaked Animals.]
tn .2
2 «
s
S
a
s
CC
s
Osseous Fishes
(Toleostei)
Ж
Mud-Fish
(Protopteri)
v-, Z&- ,, | Amphibia |
Mud-Fish
BirdS
(Aves) ——
„ X‘,7—। Tortoises
Reptiles |
| Snakes|
fcj Crocodiles |
[Lizards ~ ‘
| Myxine]
У -ЛЛ Primitive Fishes (Selachii)
Jawless Animals (Cyclostoma)
tn
(S
fl)
CC
+->
CC
| Sponges]
Star-Animals
(Echinoderma)
- nil---------
Sea-Nettles
(Acalephae)
Skull-less Animals
(Aerania)
]~insects~[
| Crustaceans |
| Amphioxus|
| Ascidians |
.J Soft Animals
<s
Monera
7Ш1Ш1,
£
c
Plant-Animals
(Zoophyta)
CL
Рис. 4.1. Древо жизни по Геккелю [Haeckel, 1879]
eS Ringed
te-Л Primitive Worms! (Archelminthes) I
Egg-Animals
(Ovularia)
.1-1 a <d
? <u P >
Worms
(Vermes)
Gastreada
hiifc* Synamoebae
Amoebae
Infusoria
92
Глава 4. Филогенетический анализ
Рис. 4.2. Филогенетические деревья, отражающие эволюционные от-
ношения между OTU. (а) Показаны элементы топологии и масштаб
дерева. Выделены горизонтальные ветви, соединяющие OTU1 и OTU5.
Их суммарная длина равна эволюционной дистанции между ОТШ и
OTU5. (б) То же дерево без корня, (в) Филогенетические отношения
между последовательностями А-I. Показаны примеры групп последо-
вательностей, формирующих филогенетические кластеры. Узлы (после-
довательности) 1-8 являются предками кластеров последовательностей,
расположенных ниже в дереве. Так, узел 3 является последним общим
предком последовательностей A-D
4.1. Филогенетические деревья 93
Внутренний узел представляет собой последнего общего
предка для последовательностей, ответвляющихся от (т. е. на-
ходящихся ниже) этого узла (см. рис. 4.2,в). Так, узел 3
на рис. 4.2, в является последним общим предком последователь-
ностей А-D. Внутренние узлы отражают моменты дихотомии,
бифуркации (bifurcation) между двумя нижерасположенными
эволюционными линиями: после этого момента, их эволюция
шла независимо друг от друга, произошла дивергенция двух
эволюционных линий (если анализируются биологические виды,
то говорят о процессе видообразования). Так, узел 3 на рис. 4.2, в
соответствует разделению эволюционных линий А-С и D. Чем
меньше суммарная длина ветвей, соединяющих две последова-
тельности, чем меньше внутренних узлов между ними, тем бо-
лее они родственны друг другу. Корень дерева является общим
предком для всех анализируемых последовательностей. Для п
OTU, общее число ветвей в филогенетическом дереве с корнем
составит 2(71—1), из которых п ветвей будут внешними, а п - 2 —
внутренними.
Возможность одновременного разделения на три (или более)
линии, политомия (polytomy), является предметом дискуссии:
действительно ли эти линии отделились друг от друга в один
и тот же момент времени, или же это артефакт, следствие того,
что временной промежуток между разделениями этих линий
был небольшим, и существующие данные и методы не позво-
ляют разделить во времени эти события?
Рассмотрим различные варианты филогенетических деревьев
для трех OTU — последовательностей А, В и С (рис. 4.3). Отме-
тим, что топология деревьев на рис. 4.3, а, б одинакова, посколь-
ку оба этих дерева показывают филогенетические отношения
между тремя последовательностями, порядок их бифуркаций,
одинаковым образом. Такие деревья называют конгруэнтными.
Для трех последовательностей возможно три неконгруэнтных
дерева, в которых последовательности А и В (см. рис. 4.3, а, б), А
и С (рис. 4.3,в), В и С (рис. 4.3,г) более родственны друг другу.
Таким образом, для трех OTU существуют три различных
(неконгруэнтных) дерева с корнем и одно дерево без корня.
При увеличении числа анализируемых OTU число возможных
Деревьев возрастает согласно формулам
(2п-3)! (2Т7-5)!
R 2п-2(тг — 2)! U 2"-3(ti —3)!’
94 Глава 4. Филогенетический анализ
Рис. 4.3. Филогенетические деревья для трех OTU. (а-г) — прямые
масштабированные деревья с корнем, (5) — прямоугольное масштаби-
рованное дерево с корнем, (е) — прямоугольное немасштабированное
дерево с корнем, (ж) — масштабированное дерево без корня. Топологии
деревьев (а) и (б) одинаковы и отличаются от топологий деревьев
(в) и (г)
где п — число анализируемых OTU, a Nr и Nu — число воз-
можных деревьев с корнем и без корня соответственно. Уже
для десяти OTU число возможных деревьев с корнем и без корня
превышает 34 миллиона и 2 миллиона соответственно. Для 100
OTU число возможных деревьев с корнем превышает расчетное
число атомов в видимой части Вселенной. Только одно из этих
деревьев является истинным.
По выбору исследователя длина ветвей дерева может от-
ражать, т. е. быть пропорциональной (рис.4.3,а-5 и ж), или
не отражать (рис. 4.3, е) эволюционную дистанцию (число нук-
леотидных замен, число других эволюционных событий) или
время после дивергенции OTU. В первом случае говорят, что
дерево масштабировано (scaled), во втором — немасштабировано
(unsealed). В немасштабированном дереве длина всех ветвей оди-
накова, такие деревья показывают только порядок бифуркаций.
Деревья с корнем могут быть изображены как прямые
(рис. 4.3,а-г), так и прямоугольные (рис. 4.3,5 и ё). В пря-
мых масштабированных деревьях длина ветвей отражает число
эволюционных событий. В прямоугольных деревьях значимы
только длины горизонтальных ветвей, а вертикальные ветви об-
4.1. Филогенетические деревья 95
дегчают презентацию дерева. Таким образом, в прямоугольных
масштабированных деревьях для определения эволюционных
дистанций между двумя OTU нужно измерить сумму длин всех
соединяющих их горизонтальных ветвей. В качестве примера
яа рис. 4.2,а все горизонтальные линии, соединяющие OTU1
и OTU5, выделены. Их суммарная длина отражает эволюцион-
ную дистанцию между 0TU1 и OTU5.
Для определения положения корня дерева можно использо-
вать внешнюю группу (outgroup, аутгруппа) — одну или несколь-
ко OTU, отпочковавшихся от общего дерева заведомо раньше
(но желательно не намного раньше) анализируемых OTU. Так,
при изучении филогенетических отношений между плацентар-
ными млекопитающими в качестве внешней группы можно
использовать сумчатых млекопитающих, при изучении эволю-
ционных отношений между человеком, гориллой и шимпанзе —
орангутана. Важность роли внешней группы в филогенетиче-
ском анализе подробно обсуждается в разд. 4.3.2 и 5.3.2.
В зависимости от топологии дерева или его части гово-
рят о звездообразных (star-like, или кустообразных, bush-like)
и кактусообразных (cactus-like) филогенетических отношениях
между анализируемыми OTU. Различия между этими тополо-
гиями представлены на рис. 4.4, на примере анализа после-
довательностей астровирусов [Lukashov and Goudsmit, 2002а].
Для звездообразной топологии характерны короткие внутренние
ветви при относительно длинных внешних ветвях, т. е. близость
к политомии, при которой различные эволюционные ветви от-
почковываются практически из одной точки. Такие филогене-
тические отношения описывают случаи эволюционной радиации
(evolutionary radiation) — значительного и быстрого (в масшта-
бах времени эволюции) возрастания генетического разнообразия
в некой группе живых систем. Примером эволюционной радиа-
ции является резкое увеличение разнообразия плацентарных
млекопитающих в палеоцене. Звездообразные филогенетические
отношения наблюдаются и между вариантами изменчивых ви-
русов (скажем, ВИЧ-1) в ходе эпидемии. Для кактусообразной
топологии характерно наличие длинных внутренних ветвей —
Ствола, почкование внешних ветвей от которого проходит через
значительные промежутки времени.
В филогенетических деревьях выделяют кластеры (cluster),
°бъединяющие группы OTU, имеющие общего предка. Иначе та-
кУю группу, кластер, называют кладой (clade). Так, на рис. 4.2,в,
Последовательности А и В формируют кластер, или кладу, после-
96 Глава 4. Филогенетический анализ
HAstV 3 AF117209
HAstV 7 Y08632
HAstV 1 L23513
HAstV 5 AB037274
HAstV 6 Z46658
-HAstV 2 L1Q745
— HAstV 4 233883
HAstV 8 AF260508
FAstV AF056197
----PAstV Y15938
— OAstV Y15937
— TAstV 2 AF206663
— TAstV 1 Y15936
----ANV 2 AB046864
— ANV AB033998
звездообразная
филогения
кактусообразная
филогения
Рис. 4.4. Примеры звездообразной и кактусообразной топологии в фи-
логенетическом дереве астровирусов. Представлены деревья без кор-
ня (а) и с корнем (б), построенные при анализе orf2 астровирусои
человека серотипов 1-8 (HAstVl-8), кошек (FAstV), свиней (PAstV)>
овец (OAstV), индюков (TAstV-1 и 2), и вирусов нефрита птиц (ANV и
ANV-2). Филогенетические отношения между астровирусами человека
характеризуются звездообразной топологией, для отношений межДУ
астровирусами животных и птиц характерна кактусообразная тополо
гия. По Лукашову и Годсмиту [Lukashov and Goudsmit, 2002а]
4.1. Филогенетические деревья 97
довательности А-G формируют кластер, а, скажем, последова-
тельности D-G — кластер не формируют. Группу OTU называют
монофилетической (monophyletic), если эта группа объединяет
все OTU, происходящие от одного общего предка (т. е. кла-
стер последовательностей). Скажем, группы последовательно-
стей А-D или А-G на рис. 4.2, в являются монофилетическими
группами, объединяющими всех потомков общих предков —
узлов 3 и 4 соответственно. Сам общий предок также входит
в монофилетическую группу. Если группа состоит из OTU, имею-
щих разных последних общих предков, то она является полифи-
летической (polyphyletic). Скажем, группа последовательностей
д, В, Н и I на рис. 4.2, в является полифилетической группой,
поскольку включает в себя последовательности, имеющие раз-
ных общих предков: последовательности А и В, для которых
последним общим предком был узел 1, и последовательности
Н и I, для которых последним общим предком был узел 7.
Если же группа состоит из OTU, имеющих общего предка, но
не включает в себя все OTU, происходящие от этого предка,
то такая группа является парафилетической (paraphyletic). Так,
группы последовательностей В-D и С-G на рис. 4.2, в являются
пар< филетическими. Обе группы имеют одного общего предка —
узлы 3 и 4 соответственно, но не включают в себя все по-
следовательности, происходящие из этих узлов. В первом слу-
чае в группу не включена последовательность А, разделяющая
с группой В-D общего предка, узел 3, во втором случае —
последовательности А и В, разделяющие с группой С-G общего
предка, узел 4. Монофилетическая группа должна включать всех
потомков одного общего предка. Парафилетическую группу мож-
но получить путем исключения из монофилетической группы
одной монофилетической подгруппы. Эти определения важны
Для понимания принципов эволюционной классификации форм
Жизни (разд. 4.7, где вопросы моно-, поли- и парафилии подробно
Рассмотрены на примерах конкретных форм жизни).
Отметим, что филогенетические отношения между какими-
либо генами в анализируемой группе форм жизни могут и отли-
чаться от филогенетических отношений между самими формами
жизни, их эволюционными линиями. Так, например, один из ге-
нов может иметь рекомбинантное происхождение (разд. 5.1),
а Другие — нет. Таким образом, филогенетические деревья, по-
кроенные для некой группы форм жизни при анализе различ-
ных участков их генома, могут иметь и различные топологии
(этот вопрос будет обсуждаться в разд. 4.6).
7 ’ли
98 Глава 4. Филогенетический анализ
Реконструкция эволюционных отношений между формами
жизни на основании сравнения их наследственной информации,
построение филогенетического дерева путем анализа генети-
ческих последовательностей, является математической задачей.
Методы построения филогенетических деревьев делятся на две
группы - дистанционные методы (distance methods, называемые
также матричными) и методы анализа дискретных признаков
(discrete characters).
4.2. Дистанционные методы построения
филогенетических деревьев
4.2.1. Принципы дистанционных методов
Первым этапом построения филогенетического дерева с помо-
щью дистанционных методов является установление попарных
эволюционных дистанций между анализируемыми последова-
тельностями, представляемых в виде матрицы дистанций (гл. 3).
Таким образом, при построении дерева с помощью любого ди-
станционного метода важен выбор эволюционной модели, оп
тимального метода расчета эволюционных дистанций между
последовательностями. Дистанционные методы позволяют стро-
ить филогенетические деревья на основе и других, непрямых,
данных о генетических дистанциях между анализируемыми
последовательностями — таких, как данные ДНК-гибридизации,
рестрикционного анализа, иммунной реактивности и т. д., необ-
ходимо лишь представить эти данные в виде матрицы дистан-
ций. После этого матрица дистанций переводится в графиче-
ский формат — собственно дерево — с использованием различ-
ных алгоритмов.
Для этой операции предложено множество подходов. От-
метим, что конкретные алгоритмы одного и того же метода
построения деревьев (и дистанционных методов, и методов
анализа дискретных признаков), использованные в различных
компьютерных программах, могут несколько отличаться друг
от друга.
Алгоритмы филогенетических методов в принципе одинако-
вы для нуклеотидных и аминокислотных последовательностей.
Самым простым дистанционным методом построения филоге-
нетических деревьев является метод невзвешенного попарного
группирования со средним арифметическим UPGMA (unweighted
pair group method with arithmetic mean). В настоящее вре-
4.2. Дистанционные методы построения деревьев 99
мя, при наличии гораздо более эффективных методов, метод
UPGMA практически не используется. Тем не менее его рассмот-
рение важно для понимания логики филогенетических методов
вообще.
4.2.2. Метод UPGMA
Изначально предложенный Сокалом и Мичинером для других
целей [Sokal and Michiner, 1958] метод UPGMA может исполь-
зоваться и для построения филогенетических деревьев на основе
анализа генетических дистанций между последовательностями.
Этот метод подразумевает строгое соблюдение принципа моле-
кулярных часов в эволюции анализируемой группы последова-
тельностей.
Алгоритм метода базируется на использовании после-
довательного группирования, при котором дерево строится
в несколько этапов, шаг за шагом. Рассмотрим принципы этого
метода на примере анализа пяти нуклеотидных последователь-
ностей, представленных на рис. 4.5. Первым этапом построения
дерева является идентификация в анализируемой группе двух
последовательностей, эволюционная дистанция между которыми
наименьшая. Как видно из представленной на рис. 4.5 матрицы
попарных дистанций, среди пяти анализируемых последователь-
ностей наименьшая эволюционная дистанция наблюдается меж-
ду последовательностями А и В, Dab = 6,0699. Последовательно-
сти А и В соединяют с их общим предком (узлом дерева) вет-
вями одинаковой длины, равной половине Dab (рис. 4.5, этап 1).
В дальнейшем эти две последовательности рассматривают как
единое целое — так называемую композитную последователь-
ность АВ.
Следующим шагом является идентификация среди остав-
шихся последовательностей той, которая имеет наименьшую
дистанцию с композитной последовательностью АВ, т. е. с по-
следовательностями А и В в среднем. Для этого рассчитывается
новая матрица эволюционных дистанций, в которой для каждой
оставшейся последовательности i рассчитывают ее дистанцию
°т композитной последовательностью АВ согласно формуле
n DAi + Dbi
U(AB\i = ---------•
Как видно из рис. 4.5, из трех оставшихся последовательно-
стей С-Е последовательность С имеет наименьшую дистанцию
100 Глава 4. Филогенетический анализ
анализируемые последовательности
A ATTCGTGAGAATGCTATCCGCGAGAATGCC
В А----------------------Т
С —СА-А------------------Т
D —CA-G------А------А----А
Е —С—G—А-----G—Т-------С—А
этап 1
В С D Е
А 0,0699 0,1473 0,2326 0,2842
В 0,1085 0,2342 0,2803
С 0,1473 0,2842
D 0,2456
этап 2 -В(АВ„- D^Db‘
В D Е
АВ 0,1279 0,2334 0,2822
В 0,1473 0,2842
С 0,2456
_„QrT О П — ^Ai+DBl+Da
этап <5 - -------g---
D Е
АВС 0,2047 0,2829
D 0,2456
этап 4 — D(abcd)e —
Dae + Рве + Dce + Dde
4
E |
0,2736 ।
_________I----------A
I------------------В
----------------------------i-c
--------------------------------------i-------------------------------------i------------------------------------D
--------------------------------------?-i i-E
—+------I I I t 1 I -I
0.12 0.10 0.08 0.06 0.04 0.02 0.00
Рис. 4.5. Построение филогенетического дерева для последовательно-
стей А-Е методом UPGMA. Дистанции между последовательностями
рассчитаны согласно модели Кимуры
4.2. Дистанционные методы построения деревьев 101
от композитной последовательности АВ, т. е.
D(ab)c = 0.1279 < D(AB)D < D(AB)E.
Соответственно последовательности С и АВ соединяют вет-
вями равной длины с их общим предком (рис. 4.5, этап 2),
и в дальнейшем эти - три последовательности рассматривают
как композитную последовательность АВС. Пересчет дистанций
и присоединение последовательностей повторяют (рис. 4.5, этапы
3 и 4) для всех оставшихся последовательностей, в данном слу-
чае — последней к дереву присоединяется последовательность Е.
В результате дистанции между всеми анализируемыми после-
довательностями А-Е и их общим предком при использовании
метода UPGMA будут одинаковыми.
Если изначально не две, а три или более последовательностей
имеют одинаковую дистанцию между собой, то первоначальный
кластер состоит из трех или более последовательностей, и их
дистанции от общего предка одинаковы. Более того, изначаль-
но — не одна пара (группа) последовательностей может иметь
одну и ту же наименьшую дистанцию между собой, но несколько
групп. В таком случае построение дерева начинается с несколь-
ких групп. Аналогично поступают, если на определенном этапе
не одна, а две или более последовательностей имеют одинаковую
дистанцию от рассматриваемой композитной последовательно-
сти: эти две или более последовательностей добавляют к кла-
стеру.
Такие случаи проиллюстрированы на рис. 4.6, а, где при ана-
лизе последовательностей А-I в качестве меры эволюционных
дистанций использовано наблюдаемое число нуклеотидных за-
мен между последовательностями. Здесь изначально три группы
последовательностей имеют наименьшую и одинаковую дистан-
цию между собой, одну замену: три последовательности — А, В
и С различаются между собой на один нуклеотид, и на один же
нуклеотид отличаются друг от друга последовательности D и Е
и последовательности D и F. При этом последовательности
Е и F различаются между собой на два нуклеотида. Таким
образом, последовательности А-С можно объединить в один
кластер, длина каждой ветви в котором будет составлять 0,5,
а последовательности D-F — в один кластер с равными длина-
ми ветвей объединить невозможно. Соответственно построение
Филогенетического дерева будет начинаться с двух кластеров:
кластера АВС (случай политомии, с точки зрения выбранной
102 Глава 4. Филогенетический анализ
анализируемые последовательности
A CTCTACTTGCGATTAATAACGCC
В ----С-------------
С ----G-------------
D —т----------------А
Е —Т----------------Т
F —т-----------с----д
G —Т---------А----Т-
Н -GT-G—С—Т---------
I -GT-T—С—А---------G
1 В С D Е F G Н I
А 1,и 1.Q' 2,0 2,0 3,0 3,0 5,0 6,0
В 1,0 3,0 3,0 4,0 4,0 5,0 6,0
С 3,0 3,0 4,0 4,0 4,0 6,0
D 1,0 1,0 3,0 5,0 5,0
Е 2,0 3,0 5,0 5,0
F 4,0 6,0 6,0
G 6,0 7,0
Н 3,0
—।--------1-----1--------1------1------1
2.5 2.0 1.5 1.0 0.5 0
Рис. 4.6. Построение филогенетического дерева для последовательно-
стей А-I методом UPGMA. В качестве меры эволюционных дистан-
ций между последовательностями использовано число наблюдаемых
различий
4.2. Дистанционные методы построения деревьев 103
эволюционной модели, меры эволюционных дистанций) и кла-
стера DE (либо DF, выбор одного из этих двух вариантов будет
определяться порядком последовательностей в анализируемой
группе). Длина ветвей в каждом кластере будет составлять
половину замены. Соответственно суммарная длина ветвей, со-
единяющих две любые последовательности в каждом из этих
кластеров, составит одну замену. На этапе 2 для всех остальных
последовательностей анализируемой группы рассчитываются ди-
станции между ними и композитными последовательностями
АВС и DE. Одна из этих оставшихся последовательностей, после-
довательность F, имеет наименьшую дистанцию от композитной
последовательности DE и будет присоединена к этому кластеру.
На этапе 3 рассчитывается новая матрица дистанций между ком-
позитными последовательностями АВС и DEF и оставшимися
последовательностями G, Н и I. Здесь наименьшая дистанция
в три замены встречается в двух случаях: между композитны-
ми последовательностями АВС и DEF и между оставшимися
последовательностями Н и I. Соответственно на этом этапе
будут сформированы два кластера: А-F и HI. На этапе 4
к кластеру А-F будет присоединена последовательность G. На-
конец, на этапе 5 кластеры А-G и HI будут соединены между
собой.
При несоблюдении модели молекулярных часов в эволюции
анализируемой группы последовательностей использование ме-
тода UPGMA приводит к ошибкам в топологии дерева. Так,
в рассмотренном на рис. 4.6 случае метод подразумевает, что
последними эволюционными событиями в анализируемой группе
последовательностей явилось ответвление линий АВС и DE
и последующая диверсификация эволюционных линий в этих
кластерах (построение дерева, объединение последовательностей
в кластеры начинается с последних эволюционных событий).
Основанием для такого вывода является накопление минималь-
ного числа нуклеотидных замен в этих линиях. Естественно, что
это условие может и не соблюдаться. Так, например, возможно,
Нто последовательности D и Е отпочковались от общего дерева
Достаточно давно, но после этого события скорость их эволюции
была гораздо меньше, чем скорость эволюции, скажем, после-
довательностей в кластере АВС. В этом случае при несоблюде-
нии модели молекулярных часов использование метода UPGMA
ае позволит установить истинную топологию филогенетического
Дерева.
104 Глава 4. Филогенетический анализ
4.2.3. Метод трансформированной дистанции
Являясь развитием метода UPGMA, метод трансформированной
дистанции (transformed distance method) позволяет (в некото-
рой степени) учесть несоблюдение модели молекулярных ча-
сов в анализируемой группе последовательностей [Farris, 1977;
Klotz et al., 1979]. В связи с наличием более точных, надежных
филогенетических методов, метод трансформированной дистан-
ции широкого распространения не получил, однако его рассмот-
рение помогает понять логику разработки филогенетических
методов, роль внешней группы — в частности.
Рассмотрим ту же группу последовательностей, что
и при описании метода UPGMA (см. рис. 4.6). Однако будем ис-
ходить из того, что задачей исследования является установление
эволюционных отношений между последовательностями А-G и
известно, что последовательности Н и I отпочковались от эволю-
ционного дерева раньше, чем произошла дивергенция в группе
А-G, т. е. последовательности Н и I являются внешней группой
по отношению к группе А-G. Скажем, последовательности A-G
получены от высших, а последовательности Н и I — от низших
приматов. В таком случае матрицу попарных эволюционных
дистанций между анализируемыми последовательностями A-G
можно рассчитать с учетом их дистанций от внешней группы,
т. е. композитной последовательности HI, согласно формуле:
Dij - Dt(HI) - Dj{HI}
uij =---------2---------+ U(HI)
где i и j — две различные последовательности группы A-G,
—дистанция между этими последовательностями, D^ni)
и DjfHi) —их дистанции от внешней группы, a D^j^ —средняя
дистанция между всеми анализируемыми последовательностями
и внешней группы, введенная для того, чтобы трансформирован-
ная дистанция D'^ являлась положительной величиной (рис. 4.7)-
Логика такого преобразования заключается в следующем-
Если эволюция всех последовательностей анализируемой группы
происходила согласно модели молекулярных часов, то неза-
висимо от индивидуальных дистанций между любыми двумя
последовательностями в группе А-G дистанция между каждой!
из этих последовательностей и общим предком группы А-6
должна быть одинаковой. Естественно, что в общем случав!
последовательность этого общего для группы А-G предка неиз-
вестна. Однако вместо этого можно рассчитать дистанцию междУ
последовательностями А-G и последовательностями внешней!
4.2. Дистанционные методы построения деревьев 105
Матрица генетических дистанций до трансформации:
В С D Е F G Н ~Т1
А 1.0 1.0 2,0 2,0 3,0 3,0 5,0 6,0
В 1.0 3,0 3,0 4,0 4,0 5,0 6,0
С 3,0 3,0 4,0 4,0 4,0 6,0
D 1,0 1,0 3,0 5,0 5,0
Е 2,0 3,0 5,0 5,0
F 4,0 6,0 6,0
G 6,0 7,0
Н 3,0
в среднем для последовательностей A-G
5,0
5.0
вл»
в,5
нГ]
5,5.
5.5
5.М
Матрица генетических дистанций после трансформации
согласно формуле
п ч--------5-----+ Мял-
В С D Е F G
А 0,50 0,75 1,25 1,25 1,25 1,00
В 0,75 1,75 1,75 1,75 1,50
С 2,00 2,00 2,00 1,75
D 1,00 0,50 1,25
Е 1,00 1,25
F 1,25
4.7. Построение филогенетического дерева для последовательно-
стей А-G методом трансформированных дистанций с использованием
Последовательностей Н и I в качестве внешней группы. Проведен
анализ последовательностей, представленных на рис. 4.6. В качестве
МеРы эволюционных дистанций между последовательностями также
Использовано число наблюдаемых различий
106 Глава 4. Филогенетический анализ
группы Ни I. Если какая-либо последовательность г груп-
пы А-G отпочковалась от дерева достаточно давно, но после
этого скорость ее эволюции была меньше, чем скорость эво-
люции остальных последовательностей, то дистанция между
последовательностью i и внешней группой будет небольшой,
а дистанции между остальными последовательностями и внеш-
ней группой — большими. Этот вопрос рассматривается также
в разд. 5.3.2.
Рассмотрим этот подход для случая с последовател ностями
А-С (см. рис. 4.7). Все эти три последовательности отличают-
ся друг от друга на один нуклеотид. Соответственно при ис-
пользовании метода UPGMA эти последовательности будут от-
почковываться из одного узла (случай политомии на рис. 4.6).
Однако отметим, что дистанции последовательностей А и В
от внешней группы, композитной последовательности HI, со-
ставляет 5,5, а дистанция последовательности С от внешней
группы меньше, 5,0. Таким образом, можно сделать вывод, что
скорость эволюции линии С меньше, чем скорость эволюции ли-
ний А и В. Поскольку дистанции между последовательностями
А, В и С одинаковы и скорость эволюции линии С меньше,
то для накопления того же числа замен в линии С требова-
лось больше времени. Соответственно можно сделать вывод,
что линия С отпочковалась от общего для последовательностей
А-С предка раньше, чем произошла дивергенция линий А и В.
Это и учитывается в методе трансформированных дистанций
(рис. 4.7). Поскольку дистанция между последовательностью С
и внешней группой меньше, чем у последовательностей А и
В, то при расчете трансформированных дистанций D'AC и D'BC
из Dac и Dbc будет вычитаться меньшее число, чем при расчете
.ОдВ, т. е. D'Ac и D'bc будут больше. Соответственно при исполь-
зовании метода UPGMA с такой трансформированной матрицей
попарных дистанций последовательности А и В будут присо-
единены к дереву в первую очередь, а последовательность С —
на следующем этапе, т. е. последовательность С будет отпоч-
ковываться от дерева ближе к корню. Таким образом, разде-
ление эволюционных линий А-С не будет случаем политомии.
Топология дерева будет приближена к истинным филогенетиче-
ским отношениям в анализируемой группе последовательностей-
Отметим и другие отличия в топологии дерева при исполь-
зовании метода трансформированной дистанции. В частности,
последовательность D более родственна последовательности F»
чем Е.
4.2. Дистанционные методы построения деревьев 107
д.2.4. Метод минимума эволюции
рДетод минимума эволюции, ME (minimum evolution), исходит
из базового принципа молекулярной эволюции — того, что наи-
более вероятным сценарием эволюции группы последовательно-
стей является тот, который требует минимального числа эволю-
ционных событий [Cavalli-Sforza and Edwards, 1967; Saitou and
[manishi, 1989; Rzhetsky and Nei, 1993].
В филогенетическом дереве сумма длин всех его вет-
вей, S, будет отражать общее число всех эволюционных со-
бытий в истории анализируемой группы последовательностей.
Таким образом, применительно к филогенетическому дереву
базовый принцип молекулярной эволюции подразумевает, что
среди всех возможных топологий дерева истинной наиболее
вероятно является та топология, для которой сумма длин
всех ветвей — наименьшая. Естественно, что при этом обя-
зательным условием является статистически достоверное от-
личие S этого дерева от S остальных деревьев. Если сум-
мы длин ветвей нескольких деревьев не имеют статистически
достоверных отличий друг от друга, то все эти деревья —
равновероятны. Методы установления длин ветвей обсуждены
в разд. 4.2.7.
Таким образом, метод минимума эволюции подразумевает
построение всех теоретически возможных деревьев и выбор
из них дерева с наименьшей суммой длин всех ветвей. Есте-
ственно, что с увеличением числа последовательностей в анали-
зируемой группе прямое использование такого подхода требует
значительного времени, поскольку число теоретически возмож-
ных топологий резко возрастает. Для решения этой проблемы
предложен ряд подходов, основанных на так называемых связях
между соседями (neighbors-relation methods). В алгоритмах этих
подходов предусматривается промежуточная выбраковка, отсев
топологий, не ведущих к минимизации S. Рассмотрим сначала
подходы к определению топологии дерева, а затем — методы
Установления длин ветвей этого дерева.
4-2.5. Методы связей между соседями
Соседями в филогенетическом анализе называют две последова-
тельности, соединенные через один узел. Так, на рис. 4.8 соседя-
1411 являются последовательности А и В и последовательности
С и D, а, скажем, последовательности А и С, соединенные
Через два узла, соседями не являются. Однако композитная
108 Глава 4. Филогенетический анализ
Рис. 4.8. Филогенетическое дерево последовательностей A-D
последовательность АВ будет являться соседом последователь-]
ностей С и D.
Предположим, что филогенетическое дерево на рис. 4.8 пред4
ставляет истинные эволюционные отношения между последова-
тельностями А-D. Для этих четырех последовательностей сумма
дистанций АС и BD будет равна сумме дистанций AD и ВС:
Dac + Dbd = Dad + Dbc — a + b + c + d + 2x = Dab + Dcd + 2a;,
и будут справедливы следующие неравенства:
Dab + Dcd < Dac + Dbd,
Dab + Dcd < Dad + Dbc-
Эти правила вместе называются условием четырех точек
(four-point condition) [Випетап, 1974].
Соответственно, если эволюционные отношения между по-
следовательностями А-D неизвестны, то это условие можно ис-
пользовать для установления соседей в филогенетическом дереве
анализируемых последовательностей. Собственно, определение
соседей среди последовательностей А-D и является установле-
нием топологии филогенетического дерева этих последователь-
ностей, эволюционных отношений между ними.
Для анализа более чем четырех последовательностей, Сат-
татом и Тверским был предложен следующий метод [Sattath
and Tversky, 1977]. Для каждой четверки последовательностей
в анализируемой группе, скажем, последовательностей i, j, т
и п, рассчитываются суммы дистанций DtJ + Dmn, Dlm + Djn
и Din + Djm. Далее определяется наименьшая из них, пусть
это будет первая сумма. Таким образом, согласно условию четы-
рех точек, в анализируемой четверке последовательности г и j
4.2. Дистанционные методы построения деревьев 109
Л последовательности т и п являются соседями. Этим парам
последовательностей присваивается значение 1, а остальным
четырем парам — i и п, i и т, j и п, j и т — значение 0.
Такая операция повторяется для всех возможных четверок
последовательностей анализируемой группы, для каждой пары
последовательностей группы суммируется общий счет, и пара
последовательностей с наибольшим общим счетом объединяется
в первый кластер дерева и используется как композитная по-
следовательность для следующего аналогичного сравнения всех
возможных четверок.
Другой метод связей между соседями, называемый методом
добрососедства (neighborliness method), был предложен Фит-
чем [Fitch, 1981]. Наибольшее распространение получил метод
присоединения соседей, NJ (neighbor-joining method), разрабо-
танный Саитоу и Неи [Saitou and Nei, 1987].
4.2.6. Метод присоединения соседей
Метод присоединения соседей является наиболее распространен-
ным дистанционным методом построения деревьев. Рассмотрим
алгоритм этого метода, начиная с логики определения топологии
дерева (рис. 4.9). Математический аппарат установления длин
ветвей будет обсуждаться в следующем разделе.
Пусть анализируемая группа состоит из N последовательно-
стей (на рис. 4.9 показан случай с N = 8). Метод присоединения
соседей начинает построение дерева со звездообразной тополо-
гии, т. е. с отсутствующими кластерами последовательностей,
отсутствующими внутренними ветвями (рис. 4.9, а). Следующим
этапом является отделение двух последовательностей — пред-
положим, последовательностей 1 и 2 — от остального дерева
^ис. 4.9. Построение филогенетического дерева методом присоединения
соседей
110 Глава 4. Филогенетический анализ
(рис. 4.9, б). При этом возникают два узла — X и У — и внут1
ренняя ветвь между ними. Узел X будет являться последним!
общим предком последовательностей 1 и 2, а У — последним]
общим предком остальных шести последовательностей 3-8. Для
такого дерева сумма длин всех ветвей будет равна
512 = 2(ЛТ —2) (Dlk + °2к> + 2Dl2+W^2 Dij'
где к — шесть остальных последовательностей (кроме последова-
тельностей 1 и 2).
Таким образом рассматривается каждая возможная пара
последовательностей, всего существует N(1 — N)/2 пар после-
довательностей. Из всех возможных пар последовательностей
выбирается пара, дающая наименьшую сумму длин ветвей. Эти
последовательности далее рассматриваются как композитная
последовательность, рассчитывается новая матрица дистанций
для Д’ — 1 последовательностей, выбирается новая пара последо-
вательностей, дающая наименьшую сумму длин ветвей дерева,
и процедура повторяется до тех пор, пока не будут установлены
все Д' — 3 внутренние ветви. Так строится дерево без корня. Для
построения корня требуется внешняя группа, и корнем дерева
будет являться узел, в котором внешняя группа присоединяется
к анализируемой группе. Если внешняя группа в анализе не ис-
пользуется, то за корень принимается сере ,ина самой длинной
ветви дерева.
Отметим принципиальное отличие метода присоединения со-
седей от метода UPGMA. В методе UPGMA в кластер объедини!
ются две последовательности, имеющие наименьшую дистанции]
между собой, т. е. самую короткую ветвь, независимо от длин
остальных ветвей дерева, а в методе присоединения соседей
в кластер объединяются последовательности, дающие наимень-
шую сумму всех ветвей дерева, т. е. учитываются и длины
остальных ветвей. При этом в отличие от метода UPGMA длины
ветвей последовательностей, выходящих из одного узла, могут
быть и не равны между собой. Так, на рис. 4.10 представлены
филогенетические отношения между теми же последовательно-
стями А-I, что и на рис. 4.6, но реконструированные методоьч
присоединения соседей. Как видно, использование метода при-
соединения соседей для тех же последовательностей приводит
к дереву с другой топологией. В частности, последовательность G
ответвляется от дерева не до, а после разделения эволюционных
4.2. Дистанционные методы построения деревьев 111
Рис. 4.10. Филогенетическое дерево, построенное методом присоедине-
ния соседей. Использованы те же последовательности А-I, что и при
построении дерева методом UPGMA на рис. 4.6. При использовании
метода присоединения соседей, длины ветвей, выходящих из каждого
внутреннего узла, в общем случае, не будут равны между собой
линий А-С и D-F. Отметим также и значительные отличия
в длинах ветвей, выходящих из одного узла — в частности,
нулевую длину внешней ветви, ведущей к последовательности D.
В дальнейшем, Студир и Кепплер [Studier and Keppler, 1988]
предложили вместо S минимизировать параметр Q, определяе-
мый формулой
N N
Q12 = (А — 2)£>12 — У2 ^2l’
i=l i=l
что выгоднее с точки зрения быстроты расчетов, а Гаску-
ел [Gascuel, 1994] показал, что
S'2 = 2<N-2)<?'2“ кН'/’"'
4.2.7. Установление длин ветвей
До сих пор при рассмотрении филогенетических методов мини-
мума эволюции и связей между соседями мы обсуждали логику
Установления топологии дерева, не говоря об алгоритмах расчета
Длин ветвей, которые используются для определения топологии.
Ь этих филогенетических методах используются одни и те же
алгоритмы расчета длин ветвей. Наиболее распространенными
Подходами являются метод Фитча-Марголиаша и стандартный
статистический метод наименьших квадратов.
112 Глава 4. Филогенетический анализ
а
В
Рис. 4.11. Установление длин ветвей филогенетического дерева
Рассмотрим сначала метод Фитча-Марголиаша [Fitch and
Margoliash, 1967]. Для этого продолжим рассмотрение метода
присоединения соседей с того момента, когда от дерева отделили
последовательности 1 и 2 (рис. 4.9,6). Для начала предположим!
что мы анализируем не восемь, а только три последовательно-
сти 1-3 (рис. 4.11,а). Обозначим последовательности 1-3 за А-С
соответственно (для удобства объяснения, что станет понятно
далее). В дереве для трех последовательностей будут только
три внешние ветви. Обозначим их неизвестные длины за J', У
и z — для ветвей, ведущих к последовательностям А, В и С
соответственно. Таким образом, генетические дистанции между
тремя последовательностями будут равны
(1дв = х + у. dAC=x+z. dBD=y + z.
Поскольку генетические дистанции между последовательно!
стями рассчитаны, то неизвестные длины всех трех ветвей этого
дерева можно найти следующим образом:
dAB+dAc — d-вс dAB+dBc — dAC
.с =--------------, у =---------------,
2 у 2
dAC + dec — d де
г~ 2
Здесь jc, у и z будут равны длинам ветвей, ведущих к посЛе"
довательностям А, В и С — ветвей , h2 и Ь3 соответственно.
4.2. Дистанционные методы построения деревьев 113
Теперь рассмотрим пример с большим числом последо-
вательностей — пусть их будет пять, последовательности 1-5
(рис. 4.11, б). Как и для трех последовательностей, обозначим
последовательность 1 за А, последовательность 2 — за В, а за
последовательность С примем композитную последовательность
3^5. В таком случае можно использовать вышеприведенные
формулы для трех последовательностей. Выражение для d^B
не изменится, а с1ас и dBc будут равны следующим выражениям:
, , <Лз + с(14 + ^15
<1АС — «1(345) — -------g-------,
, _ ^23 + <?24 + <?25
«ВС — «2(345) —
3
Отсюда находим длины ветвей bi = х и Ьг = у (рис. 4.11,6).
Далее, рассматриваем последовательности 1 и 2 как композит-
ную последовательность А, последовательность 3 — как после-
довательность В, и последовательности 4-5 — как композитную
последовательность С. Таким же образом, мы рассчитываем
длины ветвей этого дерева, учитывая, что в данном случае
, , , Ь1 + 5г
у = Оз, X — Об Ч-----------—
Учитывая, что bi и Ьг были рассчитаны ранее, можно рас-
считать Ьб. Аналогичную процедуру повторяют до установления
длин всех ветвей. Отметим, что длина какой-либо ветви может
принимать и отрицательное значение. В таком случае ее прини-
мают равной 0.
Теперь обсудим подход к установлению длин ветвей на осно-
вании метода наименьших квадратов. Рассмотрим то же дерево
Для пяти последовательностей (рис. 4.11,6). Для этого дерева ди-
станции между анализируемыми последовательностями можно
записать следующим образом:
di2 — Ь1 + Ьг + «12.
^13 = bi + Ьз + Ьб + t 13.
^14 = bl + ь^ + Ьб + by + С14,
^15 = bi + Ьб + be + br + «15.
^23 = Ьг + Ьз + Ьб + «23,
^24 = Ьг + Ьд + Ьб + Ьт + 624,
^25 = 5г Ьб + Ьб + bj + «25,
d34 = Ьз + Ьл + Ь? + «34-
^35 = Ьз + Ьб + bj + «35,
^45 = Ьл + Ьб + «45-
114 Глава 4. Филогенетический анализ
где eij — ошибки экспериментальной выборки, имеющие средне®
значение, равное 0, и дисперсию V(dtJ).
Приблизительные значения длин каждой ветви 6,—bt опр®.I
деляются согласно стандартному подходу метода наименьших
квадратов, направленному на минимизацию сумм
' (^ij ~ dij) -
г,3 '
где d\2 = bi + с?1з = bi + t>2 4- Ьб ит. д.
Ржецкий и Неи предложили простой алгоритм для приме-
нения метода наименьших квадратов при установлении длин!
ветвей филогенетического дерева [Rzhetsky and Net, 1993]. Для
описания логики этого подхода рассмотрим дерево на рис. 4.11, в,
в котором последовательности А-D являются (композитными)
последовательностями дерева на рис. 4.11, б. Например, для вет-
ви Ьб на рис. 4.11, б, А — это последовательность 1, В — 2, С — 3,
a D — композитная последовательность 4-5. Для такого дерева,!
dAB~dcD определяются согласно рассмотренной выше логике,
и длина ветви z определяется как
4- + (1 — ?)(^вс + f/ло) — Лав — den
Z~ 2
где
пвпс + nAnD
(пл 4- пв)(пс 4- nD)
а па, пв, пс и пв — число индивидуальных последовательно-
стей, из которых состоят композитные последовательности A-D
соответственно.
Филогенетический анализ с использованием дистанционных
методов может быть выполнен при использовании целого ряда
филогенетических компьютерных программ, включая, напри-
мер, простую и надежную программу MEGA (разд. 6.5).
4.3. Методы анализа дискретных признаков
4.3.1. Принципы методов анализа дискретных признаков
В отличие от дистанционных методов филогенетического анали-
за, базирующихся на анализе эволюционных дистанций междУ
последовательностями, методы анализа дискретных признаков 1
рассматривают различия между последовательностями в кон-
кретных позициях. Целью методов анализа дискретных при-
знаков является реконструкция сценария, наиболее вероятно
4.3. Методы анализа дискретных признаков 115
объясняющего порядок конкретных нуклеотидных замен (ами-
нокислотных замещений) в эволюционной истории анализируе-
мой группы последовательностей. Наиболее распространенными
методами анализа дискретных признаков являются методы мак-
симальной экономии, MP (maximum parsimony — максимальная
экономия, расчетливость, бережливость) и максимального прав-
доподобия, ML (maximum likelihood).
4.3.2. Метод максимальной экономии
Метод максимальной экономии направлен на поиск филогенети-
ческого дерева, требующего наименьшего числа эволюционных
изменений (нуклеотидных замен, аминокислотных замещений)
для объяснения наблюдаемых между анализируемыми последо-
вательностями различий. Этот метод подразумевает, что для ана-
лизируемой группы последовательностей может существовать
и несколько различных деревьев, равновероятно объясняющих
наблюдаемые различия. Изначально метод максимальной эконо-
мии был предложен для анализа аминокислотных последова-
тельностей [Eck and Dayhoff, 1966], а впоследствии модифи-
цирован Фитчем для анализа нуклеотидных последовательно-
стей (Fitch, 1977].
В основе этого метода лежит анализ нуклеотидов или амино-
кислот, находящихся в так называемых филогенетически инфор-
мативных позициях последовательностей (parsimonious sites).
Информативными называются те позиции, состояние которых,
т.е. находящиеся в этой позиции нуклеотиды или аминокисло-
ты, позволяет отдать предпочтение тому или иному филогенети-
ческому дереву (сценарию эволюции). Информативными являют-
ся те позиции, в которых у анализируемых последовательностей
находятся два или больше различных нуклеотидов (аминокис-
лот) и как минимум два из них присутствуют у двух или более
последовательностей. Для объяснения этого рассмотрим пример
с четырьмя нуклеотидными последовательностями:
123456789
1 CTGGCTGTA
2 CAAGCTGTC
3 САСААТТТА
4 САТСАТТАС
Как известно, для четырех последовательностей возможны
тРи различных дерева без корня (рис. 4.12): деревья, в которых
116 Глава 4. Филогенетический анализ
123456789
1 CTGGCTGTA
2 CAAGCTGTC
3 САСААТТТА
4 САТСАТТАС
—???!“!?!
Рис. 4.12. Анализ четырех последовательностей методом максимальной
экономии. Анализ первых четырех позиций приведенных последо*
вательностей не позволяет отдать предпочтение ни одному из трех
возможных деревьев: при любой топологии дерева минимально необхо-
димое число замен (показаны точками) одинаково (а-г). Анализ пятой
позиции позволяет отдать предпочтение первому дереву (выделено),
поскольку такой сценарий эволюции более экономен: он предполагает
одну замену, а другие сценарии — две ((?). Знаками «—», ♦?» и •!*
обозначены инвариантные, вариабельные неинформативные и филоге-
нетически информативные позиции соответственно
4.3. Методы анализа дискретных признаков 117
родственными парами последовательностей (соседями) являются
пары 1 и 2 — и 3 и 4, 1 и 3 — и 2 и 4, и 1 и 4 — и 2 и 3. Поскольку
в первой позиции все четыре анализируемых последовательно-
сти имеют один и тот же нуклеотид (С), то анализ этой позиции
не дает оснований отдать предпочтение какому-либо из трех
возможных деревьев (рис. 4.12, а). Таким образом, инвариантные
позиции (одинаковые у всех последовательностей) не являются
информативными.
Во второй позиции последовательность 1 имеет нуклео-
тид Т, а последовательности 2-4 — нуклеотид А. Самым про-
стым (т. е. наиболее вероятным) сценарием эволюции анализиру-
емой группы последовательностей является одна замена в этой
позиции: замена А на Т у последовательности 1 (рис. 4.12, б).
Очевидно, что эта замена в эволюционной истории последо-
вательности 1 равновероятна при любом из трех возможных
сценариев эволюции последовательностей 1-4. Эта замена могла
равновероятно произойти в эволюционной линии последователь-
ности 1 независимо от того, какой из остальных последова-
тельностей 2-4 последовательность 1 более родственна. Таким
образом, анализ этой гетерогенной позиции не дает оснований
для заключения, какой именно из последовательностей 2-4
последовательность 1 более родственна, т. е. не позволяет отдать
предпочтение ни одному из трех возможных деревьев. Анало-
гично не дает таких оснований и анализ позиции 3, в кото-
рой все четыре последовательности имеют четыре различных
нуклеотида (рис. 4.12, в). У общего предка четырех анализируе-
мых последовательностей в этой позиции мог находиться любой
из четырех нуклеотидов, и наиболее простым сценарием эволю-
ции является сохранение этого нуклеотида у одной из последо-
вательностей (неизвестно, какой) и независимые замены этого
нуклеотида в каждой из трех оставшихся последовательностей.
Таким образом, равновероятны четыре самых простых сценария
эволюции, каждый из которых требует трех замен, и анализ
этой позиции не дает оснований отдать предпочтения ни одному
из возможных деревьев.
В позиции 4 первые две последовательности имеют одинако-
вый нуклеотид, G, последовательность 3 — нуклеотид А, а по-
следовательность 4 — нуклеотид С (рис. 4.12, г). Таким образом,
Можно предположить, что у общего предка последовательностей
1~4 в этой позиции находился нуклеотид G, и в эволюционной
истории группы произошли две замены, в линиях 3 и 4. Тем
не менее анализ и этой позиции не дает оснований отдать
118 Глава 4. Филогенетический анализ
предпочтение ни одному из трех возможных деревьев: любое!
из них соответствует наиболее простому сценарию эволюции j
этой позиции, требующему двух независимых замен в последо-
вательностях 3 и 4. Поскольку анализ позиций 2-4, несмотря
на наличие в них нуклеотидных замен, не дает оснований]
отдать предпочтение одному из трех возможных деревьев, та-
кие позиции также не являются информативными. В отли-
чие от дистанционных филогенетических методов, в которых
замены в позициях 2-4 влияют i?a генетические дистанции'
между последовательностями и, таким образом, учитываются
при построении дерева, все эти неинформативные позиции
не учитываются при построении дерева методом максимальной
экономии.
В позиции 5 у первых двух последовательностей находится
нуклеотид С, а у двух оставшихся последовательностей — нук-
леотид А (рис. 4.12,5). Наиболее простым сценарием эволюции
этой позиции будет наличие нуклеотида А или С у общего
предка всех четырех последовательностей и одна последующая
замена: либо замена А —»С у общего предка последовательностей
1 и 2, либо замена С —»А у общего предка последовательностей
3 и 4, т. е. одна замена в эволюционной истории анализируемой
группы последовательностей. Таким образом, анализ этой пози-
ции позволяет отдать предпочтение эволюционному сценарию,
в котором родственными парами последовательностей являются
пара 1 и 2 и пара 3 и 4. Если же предполагать, что родственными
парами последовательностей являются, скажем, пара 1 и 3 и па-
ра 2 и 4, то для объяснения наблюдаемых рас.'.ичий требуется
не одна, а две замены: одна замена в первой паре и одна —
во второй паре, что менее вероятно. Таким образом, анализ
этой позиции позволяет отдать предпочтение одному из трех
возможных деревьев (выделено на рис. 4.12,5). Соответственно
такие позиции являются информативными.
Рассматриваемые последовательности имеют три филогене-
тически информативных позиции. Анализ позиций 5 и 7 дает
основание отдать предпочтение дереву, в котором родственными
являются пара 1 и 2 и пара 3 и 4, анализ позиции 9 — дереву,
в котором родственными являются пара 1 и 3 и пара 2 и 4. Та-
ким образом, максимально экономным деревом является дерево,
в котором родственными последовательностями являются пара 1
и 2 и пара 3 и 4.
При возрастании числа анализируемых последовательностей
принцип анализа остается тем же самым. Естественно, что
4.3. Методы анализа дискретных признаков 119
его процедура усложняется в связи с необходимостью учиты-
вать большее число различных возможных деревьев. Алгоритм
пОчска максимально экономного дерева был предложен Фит-
чем [Fitch, 1971].
Рассмотрим алгоритм метода максимальной экономии на при-
зере анализа шести нуклеотидных последовательностей 1-6,
в некой позиции которых находятся следующие нуклеотиды: С,
д, G, А, Т и Т. Поскольку в этой позиции находятся как мини-
мум два различных нуклеотида, как минимум два из которых
присутствуют у как минимум двух последовательностей — это
нуклеотиды А и Т, то эта позиция является информативной.
Такие позиции используются в анализе.
Начнем с рассмотрения некоего — любого из возможных
для этих последовательностей — филогенетического дерева.
Пусть это будет дерево на рис. 4.13, а.
Поскольку базовым принципом метода максимальной эко-
номии является поиск филогенетического дерева, требующего
наименьшего числа замен для объяснения наблюдаемых между
анализируемыми последовательностями различий, необходимо
подсчитать число замен, происшедших в случае, если эволюция
анализируемой группы последовательностей проходила согласно
предполагаемому на рис. 4.13, а сценарию. Для этого следует
рассчитать нуклеотиды, вероятно находившиеся в узлах (общих
предках) 7-11.
Поскольку в анализируемой позиции последовательностей 1
и 2 находятся нуклеотиды С и А, то нуклеотид в узле 7 не может
быть установлен однозначно. Равновероятны два сценария, когда
у общего предка последовательностей 1 и 2 в этой позиции был
либо нуклеотид С, либо нуклеотид А, после чего имела место
одна замена: либо замена С —► А в последовательности 2, либо
замена А—»С в последовательности 1. Такая невозможность од-
нозначно установить наиболее вероятный нуклеотид в последо-
вательности общего предка математически требует объединения
(union) нуклеотидов потомков (нисходящих ветвей). Аналогично
во может быть однозначно установлен нуклеотид у общего пред-
ка последовательностей 3 и 4 (узел 8): это мог быть либо G, либо
А, после чего в одной из эволюционных линий — либо в линии 3,
либо в линии 4 — произошла одна замена (второе объединение).
Соответственно у общего предка последовательностей 3, 4 и 5
(узел 9) в этой позиции находился либо G, либо А, либо Т, после
чего произошли две нуклеотидные замены, в двух из трех линий
(третье объединение).
120 Глава 4. Филогенетический анализ
Рис. 4.13. Построение филогенетического дерева мет дом максимальной
экономии. Представлено дерево, требующее четырех замен (а и б), и два
возможных наиболее экономных дерева, каждое из которых требует
трех замен (в и г)
Переходя к общему предку последовательностей 1-5
(узлу 10), мы видим, что одна из эволюционных линий,
отпочковавшаяся от этого узла (линия последовательностей 1
и 2, узел 7) имела в рассматриваемой позиции либо С, либо А,
а другая линия (линия последовательностей 3-5, узел 9) —
либо G, либо А, либо Т. Если исходить из того, что у общего
предка последовательностей 1-5 в анализируемой позиции
находился нуклеотид А, то такой сценарий предполагает
наименьшее, наиболее экономное, число последующих замен:
А—»С в последовательности 1, A—»G в последовательности 3
и А —»Т в последовательности 5 — всего, таким образом, три
4.3. Методы анализа дискретных признаков 121
замены в нисходящих линиях. Если же исходить из того,
что у общего предка последовательностей 1-5 в анализиру-
емой позиции находился другой нуклеотид, то объяснение
наблюдаемых между последовательностями 1-5 различий
потребует большего числа замен, т. е. не будет соответствовать
принципу максимальной экономии. Скажем, если у общего
предка последовательностей 1-5 в рассматриваемой позиции
находился нуклеотид С, то в нисходящих линиях 2-5 произошли
четыре замены, что менее вероятно. Соответственно, исходя
из принципа максимальной экономии, (пока) следует однозначно
полагать, что в анализируемой позиции узла 10 находился
нуклеотид А. Объединение здесь не требуется.
Переходя к узлу 11, т. е. к общему для всех анализируемых
последовательностей предку, мы видим, что в одной из двух
нисходящих ветвей этого узла в рассматриваемой позиции на-
ходился нуклеотид А (узел 10, общий предок последователь-
ностей 1-5), а в другой — находится нуклеотид Т (последова-
тельность 6). Соответственно необходимо еще одно, четвертое,
объединение, и следует сделать вывод о том, что общий предок
всех шести последовательностей имел либо А, либо Т в ана-
лизируемой позиции. Если узел 11 имел А в анализируемой
позиции, то в последовательности 6 произошла замена А—>Т,
четвертая для всей анализируемой группы. Отметим, что воз-
можное наличие нуклеотида Т в анализируемой позиции узла 11
меняет данную нами ранее оценку вероятности нахождения А
в этой позиции узла 10: для всех шести последовательностей
вместе, наличие А в узле 10 подразумевает то же число замен
в эволюции всей анализируемой группы (четыре замены), что и
наличие Т: в случае нахождения в узле 11 нуклеотида Т, замены
в последовательности 6 не было, а в последовательностях 1-5
было на одну замену больше, в сумме — те же четыре замены.
Таким образом, после анализа всех шести последовательностей
следует сделать вывод о том, что, если дерево на рис. 4.13, а от-
ражает истинные филогенетические отношения между последо-
вательностями 1-6, то в рассматриваемой позиции в узле 10 мог
находиться с равной вероятностью как А, так и Т (рис. 4.13,6).
Математически говоря, во внутреннем узле находится нук-
леотид, являющийся пересечением множеств нуклеотидов в нис-
ходящих линиях. Если же это пересечение нулевое, т.е. в нисхо-
дящих линиях нет одинаковых нуклеотидов, то в узле находится
объединение множеств нуклеотидов в нисходящих линиях.
122 Глава 4. Филогенетический анализ
Поскольку в рассматриваемом случае в информативной пози-
ции у шести последовательностей находятся четыре различных
нуклеотида, то в эволюционной истории этих последователь-
ностей минимально необходимыми являются три замены. Ддя
рассматриваемого случая возможно несколько альтернативных
одинаково экономных деревьев, требующих не четыре, как
на рис. 4.13, а, а три объединения (соответственно три замены),
т. е. максимально экономных, наиболее вероятных дерева. Два
таких дерева представлены на рис. 4.13, в и г. Вообще, в отличие,
например, от метода присоединения соседей, метод максималь-
ной экономии может приводить и к нескольким деревьям, что
является его преимуществом, поскольку позволяет сравнить
альтернативные топологии.
Естественно, что в практических случаях число информатив-
ных позиций в анализируемой группе последовательностей —
(много) больше одной. Собственно, чем больше информативных
позиций, тем, в общем случае, надежнее филогенетическое дере-
во: малое число информативных позиций, как правило, приводит
к слишком многим альтернативным равновероятным деревьям,
без возможности выбрать одно из них. Алгоритм построения де-
рева при большем числе информативных позиций не отличается
от рассмотренного на рис. 4.13 случая для одной информативной
позиции. Для каждой возможной топологии рассматривается
минимально необходимое число замен в каждой информативной
позиции с использованием того же правила пересечения или
объединения для каждой позиции отдельно, и итоговое необ-
ходимое число замен суммируется по всем позиг ям.
Таким образом, процедура поиска максимально экономно-
го дерева заключается в следующем. Во-первых, необходи-
мо идентифицировать все информативные позиции. Во-вторых,
для каждого возможного дерева следует рассчитать минималь-
ное необходимое число замен в каждой информативной позиции
и просуммировать их по всем позициям (одна ли это позиция,
или же их много больше, на принципы алгоритма это не вли-
яет). Наконец, из всех возможных деревьев нужно выбрать
то, для которого эта сумма минимальна (возможен и выбор
нескольких деревьев с одинаковой минимальной суммой). При
построении дерева методом максимальной экономии длины его
ветвей будут пропорциональны минимально необходимому числу
замен при переходе от одного узла к другому.
Построение филогенетического дерева с помощью метода
максимальной экономии может быть выполнено при использова- I
4.3. Методы анализа дискретных признаков 123
ции ряда филогенетических компьютерных программ, включая
pHYLIP и PAUP* (разд. 6.5).
4.3.3. Метод максимального правдоподобия
Этот метод, изначально предложенный для анализа данных
о частотах генов [Cavalli-Sforza and Edwards, 1967], был впо-
следствии модифицирован Фельзенштейном для анализа ами-
нокислотных и нуклеотидных последовательностей [Felsenstein,
1973; Felsenstein, 1981].
В отличие от метода максимальной экономии метод макси-
мального правдоподобия, как и дистанционные филогенетиче-
ские методы, базируется на использовании моделей эволюции
(гл. 3). Однако в отличие от дистанционных методов метод мак-
симального правдоподобия использует эти модели для построе-
ния филогенетического дерева, исходя из оценки вероятности
(правдоподобия) нахождения каждого конкретного нуклеотида
(аминокислоты) в каждой конкретной позиции.
Говоря несколько упрощенно, различие между понятиями
♦вероятность» и ♦правдоподобие» состоит в том, что первое
понятие позволяет предсказать возможный исход случайного
эксперимента при известном числе параметров, а второе — опре-
делить неизвестное число параметров на основании известных
результатов эксперимента. Скажем, если известно число граней
правильного многогранника (т. е. число параметров), то можно
определить, чему равны вероятности различных исходов бросков
этого многогранника. Так, для шестигранной игральной кости
вероятность любого исхода броска будет равна 1/6. Однако
предположим, что число граней некой игральной кости нам
неизвестно. В таком случае многократно повторяя эксперименты
с броском этой игральной кости, можно сделать предположение
° числе граней и определить правдоподобие этого предполо-
жения. Так, многократно подбрасывая некую игральную кость
с неизвестным числом граней и наблюдая, что число различных
Исходов бросков кости равно шести, можно сделать предполо-
жение о том, что кость шестигранная. Правдоподобие этого
предположения будет расти по мере того, как повторные броски
будут продолжать приводить к одному из шести исходов, но
Исчезнет, если очередной бросок закончится новым, седьмым,
Исходом.
В известном смысле, функция вероятности направлена в бу-
^УЩее, на установление предполагаемых исходов при известных
124 Глава 4. Филогенетический анализ
начальных условиях (начальные условия известны, а исход, ре.
зультаты могут быть оценочно установлены), а функция правдо.
подобия — в прошлое (известны результаты — следует оценочно
установить начальные условия).
Пусть эволюция некой анализируемой нами группы нуклео.
тидных последовательностей 1-4 адекватно описывается некой
эволюционной моделью, скажем, однопараметрической моделью
Джукса—Кантора (разд. 3.2.1). Таким образом, вероятность со-
хранения или замены нуклеотида i в некой позиции за время t
определяется приведенными в разд. 3.2.1 формулами. Нашей
целью является построение филогенетического дерева для по-
следовательностей 1-4, в некой позиции которых находятся
нуклеотиды i, j, к и I соответственно.
Рассмотрим некое филогенетическое дерево для этих по-
следовательностей (рис. 4.14, а). Будем исходить из того, что у
общего предка всех четырех последовательностей в этой по-
зиции находился нуклеотид .с. Тогда вероятность нахождения
в этой позиции, скажем, нуклеотида I у последовательности 4
будет равна Pxi(t\ 4-+ <з). а общая вероятность нахождения
конкретных нуклеотидов г — I в анализируемой позиции после-
довательностей 1-4 —
Р = Pxl(tl + ^2 + t3) Pxy(t 1 )Р yk(t2 + t3)PyZ(t2)Pzi(ts)PZJ(t3),
Рис. 4.14. Построение филогенетического дерева методом максималыЯ
го правдоподобия
4.3. Методы анализа дискретных признаков 125
где вероятности всех замен рассчитываются согласно использу-
емой нами в данном случае модели Джукса—Кантора.
Естественно, что мы не знаем, какой именно нуклеотид
находился в этой позиции у общего предка. Однако мы можем
предположить, что вероятность нахождения нуклеотида х в этой
позиции равняется некой величине д,г. Обычно за дх прини-
мают частоту нахождения нуклеотида х в современных (ана-
лизируемых) последовательностях. На этом этапе от функции
вероятности мы переходим к функции правдоподобия: на основа-
нии известного результата эксперимента (эволюции) — нахожде-
ния конкретных нуклеотидов в конкретных позициях анализи-
руемых последовательностей, частоты различных нуклеотидов
в этих последовательностях, следует установить эволюционные
параметры.
Для рассматриваемого гипотетического дерева функция прав-
доподобия будет описываться следующей формулой:
к, I) = 53 gxPxi(ti + ^2 + t-л) х
Х 53 (^2 + ^з) 53 ^г(^2)^гг(^з)^г>(^з)-
У г
До сих пор мы исходили из предположения о постоянстве
скорости эволюции. Однако это предположение необязательно,
можно рассмотреть и различные скорости эволюции различных
ветвей. В таком случае для каждой ветви п можно учитывать
собственную скорость эволюции, Ап. Длина этой ветви ип будет
Равна Antn (рис. 4.14,6)
В приведенном выше примере мы рассматривали функцию
правдоподобия для одной нуклеотидной позиции. Если эволюция
всех позиций происходит независимо друг от друга, то функ-
ция правдоподобия для всех позиций последовательности будет
Равна произведению функций правдоподобия для каждой инди-
видуальной позиции. Пусть рассматриваемые на рис. 4.14 после-
довательности 1-4 состоят не из одного, а, скажем, из Л = 3
нуклеотидов. Тогда функция правдоподобия рассматриваемого
Филогенетического дерева будет определяться формулой
з
— JJ Ln,
N=l
индивидуальные Ln определяются согласно предыдущей
Формуле.
126 Глава 4. Филогенетический анализ
В этой формуле неизвестными являются Antn ветвей. Суще.
ствует математический аппарат, направленный на максимиза-
цию L — установление значений Antn, при которых значение £
максимально, шах£. После установления шах£ для этого гипо-
тетического дерева следует повторить процедуры максимизации
£ для всех других возможных деревьев и выбрать дерево с
наибольшим значением шах£. При использовании метода мак-
симального правдоподобия длина всех ветвей дерева устанав-
ливается с помощью математического аппарата, направленного,
на максимизацию функции правдоподобия этого дерева.
Одной из наиболее совершенных компьютерных программ,
использующей возможности метода максимального правдоподо-
бия, является программа PAML (разд. 6.5). Отметим, что под-
ходы, основанные на анализе правдоподобия функций, широко
используются не только непосредственно при построении фило-
генетического дерева, но и при анализе наличия молекулярных
часов в эволюции группы последовательностей (разд. 5.3.3), уста-
новлении параметров эволюционных моделей (разд. 6.5) и т. д. (
4.4. Статистическая оценка дерева, бутстрэп-анализ
После построения филогенетического дерева с помощью любо-
го метода возникает вопрос: насколько достоверна топология
построенного дерева? Ряд филогенетических методов, включая
методы минимума эволюции, максимальной экономии и макси-
мального правдоподобия, позволяют получать несколько альтер-
нативных деревьев. В таких случаях в /бранную наилучшую
(согласно использованному математическому аппарату) тополо-
гию можно сравнить с другими лучшими топологиями (второй
лучшей, третьей лучшей и т. д.). Если между этими несколь-
кими лучшими топологиями нет статистически достоверных
различий, то все они равновероятны. Однако ряд филогенети-
ческих методов, скажем, широко используемый метод присо-
единения соседей, не предоставляет возможности рассмотрения
альтернативных топологий. Более того, статистические вопросы
в филогенетическом анализе не ограничиваются оценкой стати-
стической достоверности топологии дерева в целом, установле-1
нием только того, насколько достоверно выбранная топология
отличается от альтернативной.
Важным критерием оценки дерева является проверка на-
дежности каждой ветви дерева. Если статистические методы
не дают оснований для заключения о достоверном существо-!
4.4. Статистическая оценка дерева, бутстрэп-анализ 127
вании какой-либо ветви дерева, то, значит, нельзя исключить
и возможного отсутствия этой ветви, соответственно — возмож-
ности другой топологии дерева ниже этой ветви. Надежность
ветви дерева можно оценить с помощью различных статисти-
ческих методов. Так, можно рассчитать стандартную ошибку
длины ветви и с помощью статистических тестов установить
достоверность отличия длины ветви от 0. Если использованный
математический аппарат не дает оснований для заключения
о том, что длина ветви статистически достоверно отличается
от 0, т. е. что эта ветвь достоверно существует, то, естественно,
нельзя исключить и отсутствия этой ветви дерева.
Одними из самых распространенных подходов к оценке на-
дежности филогенетического дерева, его ветвей, являются ста-
тистические подходы, основанные на методах повторных вы-
борок (resampling). Среди таких методов наибольшее распро-
странение получил бутстрэп-анализ (bootstrap analysis, bootstrap
resampling) [Zharkikh and Li, 1992a; Zharkikh and Li, 1992b;
Zharkikh and Li, 1995]. Бутстрэп-анализ является универсаль-
ным, т. е. используемым не только в филогенетическом анализе,
статистическим методом. Для понимания особенностей приме-
нения этого метода в филогенетическом анализе рассмотрим
принципы бутстрэп-анализа подробнее.
Предположим, что анализируемая группа состоит из девяти
последовательностей длиной в 20 нуклеотидов каждая:
11111111112
12345678901234567890
А ТААТААТААТААТААТААТА
В TAATAATAATAATAATAATG
С ТААТААТААТААТААТАСТС
D ТААТААТААТААТТССССТТ
Е TAATAATAATAATGCCCCTT
F ТААТААТААТААСТТСССТТ
G ТААТААТААТААТТТАССТТ
Н ТААТААТАССССССССССТТ
I TAATAATAGGGGACCCCCTT
Филогенетическое дерево для этой группы последовательно-
стей, построенное методом присоединения соседей с использо-
ванием p-дистанций, представлено на рис. 4.15, а. Оценка ста-
тистической достоверности топологии этого дерева с помощью
128 Глава 4. Филогенетический анализ
Рис. 4.15. Бутстрэп-анализ в оценке статистической достоверности
филогенетического дерева. Для приведенных в тексте последовательно-
стей построено филогенетическое дерево с ис пользованием р-дистанций
и метода присоединения соседей (а). Проведен бутстрэп-анализ этого
дерева с использованием 100 случайных выборок, значения бутстрэп-
анализа указаны для каждого внутреннего узла. Кластеры со значени-
ями выше 90 были признаны достоверными (выделены) (б). Кластер
последовательностей А, В и С — статистически поддержан (значение
бутстрэп-анализа — 100), кластер последовательностей А и В — нет
(значение бутстрэп-анализа — 62), соответственно — в кластере АВС
возможны и другие порядки бифуркаций (в)
бутстрэп-анализа проводится следующим образом. В анализирУ'
емой группе последовательностей случайным образом выбирает
ся одна позиция — пусть это будет позиция 17 — и записываете^
как первая позиция новой группы последовательностей, перв°Ч
случайной выборки. Затем, случайным образом, выбирается еШе
4.4. Статистическая оценка дерева, бутстрэп-анализ 129
одна позиция — пусть это будет позиция 4 — и записывается как
вторая позиция этой первой случайной выборки. Эта процедура
повторяется 20 раз, согласно длине анализируемой группы по-
следовательностей. Таким образом, создается первая случайная
выборка:
11 1 12111 1111
74752894650207312691
А АТААААААААААТААТАТТА
В ATAAAAAAAAGATAATATTA
С ATAAAAAAAAGATAATATTA
D СТСААААТАСТАТСАТАСТА
Е CTCAAAAGACTATCATACTA
F СТСААААТАТТАТСАТАСТА
G СТСААААТАТТАТСАТААТА
Н СТСАААССАСТСССАТССТС
I CTCAAAGCACTGGCATGCTG
Поскольку позиции анализируемой группы последовательно-
стей выбираются случайным образом, то одна и та же позиция
может быть представлена в некой выборке и несколько раз.
Так, в рассматриваемой случайной выборке позиция 12 анали-
зируемых последовательностей отобрана дважды, позиция 17 —
трижды, а позиции 7, 13 и 18 — не отобраны ни разу.
Аналогичным образом создается и вторая, и последующие
случайные выборки. Вторая случайная выборка может выгля-
деть следующим образом:
1 211 1 1 1 11 111
09039937848810652571
А ТААТТАТТАТАААТААААТТ
В TAGTTATTATAAATAAAATT
С ТАСТТАТТСТСААТААААТТ
D ТАТТТАТТСТСААТАСАСТТ
Е ТАТТТАТТСТСААТАСАСТТ
F ТАТСТАСТСТСААТАТАТТТ
G ТАТТТАТТСТСААТАТАТТТ
Н ССТСТССТСТСАССАСССТТ
I GGTATGATCTCAGGACGCTT
Обычно при проведении бутстрэп-анализа создают
100-1000 случайных выборок. После этого для каждой выборки
130 Глава 4. Филогенетический анализ
строится филогенетическое дерево с использованием тех ясе
методов, что и для анализируемого дерева (т. е. в данном
случае — с использованием p-дистанций и метода присоединения
соседей). Все полученные деревья сравнивают с анализируемым!
деревом, определяя, есть ли в этих деревьях те же самые
внутренние узлы, что и в анализируемом дереве. Для каждого
внутреннего узла анализируемого дерева подсчитывают процент
(или абсолютное число) деревьев, в которых присутствует
тот же узел, т. е. все последовательности, находящиеся ниже
некоего узла в анализируемом дереве, находятся ниже этого
узла и в деревьях, построенных для случайных выборок.
Этот процент (или абсолютное число) называют значением
бутстрэп-анализа (bootstrap value) и записывают рядом с узлом.
Результаты бутстрэп-анализа с использованием 100 случайных
выборок для рассматриваемых последовательностей приведены
на рис. 4.15,6. Как видно, последовательности А и В, имеющие
в анализируемом дереве общий узел, объединены этим узлом
в 62 из 100 деревьев, построенных для случайных выборок.
В то же время общий для последовательностей А, В и С узел
анализируемого дерева присутствует в 100 из 100 деревьев,
построенных для случайных выборок. Таким образом, результа-
ты бутстрэп-анализа рассматриваемого дерева показывают, что
о существовании общего узла АВС можно говорить с большей
степенью уверенности, чем о существовании узла АВ. Как
правило, достоверно установленными узлами считают те, для ко-
торых значение бутстрэп-анализа превышает 70, лучше — 90.
В рассматриваемом примере, поскотьку последовательности
А, В и С расположены ниже общего для них узла в 100%
случаев, этот общий для последовательностей АВС узел следует
считать достоверно установленным, а топологию дерева ниже
узла АВС, т. е. наличие общего узла последовательностей А и В,
достоверно установленной считать нельзя. Ниже узла АВС воз-
можны и альтернативные топологии дерева, в которых вместе
группируются не последовательности А и В, а последователь-
ности А и С или В и С (рис. 4.15,в). Тем не менее совместное
группирование последовательностей А и В все равно является
наиболее вероятным сценарием.
Наибольшее распространение бутстрэп-анализ получил в со-
четании именно с методом присоединения соседей, что связано
с быстродействием этого метода, позволяющим легко проводить
анализ большого числа случайных выборок.
4.5. Другие филогенетические методы 131
4.5. Другие филогенетические методы
Описанные выше алгоритмы и методы являются наиболее на-
дежными, изученными и распространенными методами фило-
генетического анализа, широко используемыми при изучении
эволюционных отношений. Предложен и ряд других алгорит-
мов, среди которых в первую очередь следует упомянуть груп-
пу методов инвариантов (methods of invariants) [Cavender and
felsenstein, 1987; Lake, 1987; Cavender, 1989; Fu and Li, 1992; Fu,
1995] и метод головоломок квартетов (quartet puzzling) [Strimmer
and von Haeseler, 1996; Strimmer et al., 1997; Ranwez and
Gascuel, 2001; Schmidt et al., 2002; von Haeseler and Strimmer,
2004; Snir et al., 2008] (разд. 6.5).
В современных компьютерных программах для филогенети-
ческого анализа, в частности, при использовании метода мак-
симального правдоподобия, активно разрабатываются математи-
чески более сложные подходы к построению деревьев, основан-
ные на байесовской статистике, скрытых марковских моделях
и симуляциях Монте-Карло, работах Колмогорова, позволяющих
статистически оценивать вероятность того или иного эволюцион-
ного сценария [Eddy, 1996; Eddy, 1998; Huelsenbeck et al., 2002;
Holmes, 2003; Eddy, 2004a; Eddy, 2004b; Gasbarra et al., 2007].
Все описанные выше методы, традиционный филогенети-
ческий анализ вообще, представляют эволюционные отноше-
ния между анализируемыми OTU в виде филогенетического
дерева, определенного порядка ветвей. Логика такого подхода
подразумевает, что порядок бифуркаций всех анализируемых
эволюционных линий установлен однозначно, экспериментально
разрешен, и эволюция этих линий после их отделения от общего
предка происходит независимо — эти линии не взаимодейству-
ют, не обмениваются генетической информацией между собой.
Естественно, что оба этих положения могут и не соблюдать-
ся [Doolittle, 1999]. Может существовать неопределенность в от-
ношении порядка бифуркаций в эволюционной истории анали-
зируемой группы последовательностей. Любой вышеописанный
Филогенетический метод направлен на абсолютное разрешение
Филогенетических отношений, установление порядка бифурка-
ций. Однако интересно рассмотреть и возможную неопреде-
ленность филогенетических отношений между анализируемыми
Последовательностями, визуализировать те случаи, в которых
Филогенетические отношения не могут быть установлены одно-
значно. Кроме того, известно, что эволюция двух линий после
132 Глава 4. Филогенетический анализ
Рис. 4.16. Отображение филогенетических отношений между OTU с
мощью филогенетических сетей
7
их отделения от общего предка может включать в себя и обмен
генетической информацией между ними — скажем, между эти
ми линиями могут происходить рекомбинации, горизонтальный
перенос генов и т. д. В традиционном филогенетическом дереве
отображение этих феноменов не предусматривается.
Для учета этих явлений предложен другой подход к отобра
жению эволюционных отношений между последовательностями
базирующийся на понятии филогенетических сетей [Huson and
Bryant, 2006; Makarenkov et al., 2006]. Пример отображения
эволюционных отношений между последовательностями с по-
мощью филогенетических сетей представлен на рис. 4.16. Как
видно, при таком подходе предоставляется возможность про
анализировать и графически отобразить возможную неопреде
ленность в реконструкции эволюционн» гх событий, альтернатив
ные эволюционные пути, а также возможные взаимодействия
(обмен генетической информацией) при эволюции различных
линий. Наиболее проработанный на сегодняшний день алго
ритм отображения эволюционных отношений в виде филогене
тических сетей базируется на теории расщепления-разложения
(split-decomposition theory) [Випетап, 1971; Dopazo et al., 1993]
и включен в программу SplitsTree [Huson, 1998] (разд. 6.5)-
Однако характеристики этого алгоритма, как и других перечне
ленных в этом разделе подходов к филогенетическому анализу,
изучены пока недостаточно.
В разд. 4.1 уже был отмечен вопрос о том, что филоге
нетические отношения между генами каких-либо форм жиз
ни могут и не соответствовать филогенетическим отношениям
между этими формами жизни. Так, если у двух близкороД
ственных форм жизни некий ген был приобретен различными
4.6. Сравнение филогенетических методов 133
путями (рекомбинация, горизонтальный перенос генов и др.),
то результаты филогенетического анализа последовательности
этого гена будут отличаться от результатов анализа других
генов. Подходы, основанные на понятии филогенетических се-
тей, могут отобразить несоответствия между деревьями, ос-
нованными на анализе различных генов. Другими подходами
к учету и пониманию несоответствий между деревьями, по-
строенными при анализе различных генетических участков,
являются подходы, основанные на согласовании деревьев (tree
reconciliation) [Вьюгин с соавт., 2002, Вьюгин с соавт., 2005;
Morris, 1998; Martin, 2000; Arvestad et al., 2003; Mirkin et al.,
2003; Jackson, 2004; Jackson and Charleston, 2004; Beiko and
Hamilton, 2006]. Целью согласования деревьев является созда-
ние одного итогового, консенсусного, дерева (consensus tree),
которое отражает филогенетические отношения между формами
жизни на основании установленных отношений между различ-
ными их генетическими участками (в том числе и неконгруэнт-
ных отношений). Отметим, что понятие консенсусного дерева
в современной литературе неоднозначно: в ряде случаев под
консенсусным деревом понимается дерево, имеющее только ста-
тистически достоверные (например, поддержанные результатами
бутстрэп-анализа) ветви, иначе — показана политомия, мульти-
фуркация.
4.6. Сравнение филогенетических методов
Как и при сравнении эволюционных моделей (разд. 3.2.6),
при обсуждении характеристик различных филогенетических
методов прежде всего необходимо отметить, что не существу-
ет филогенетического метода, заведомо превосходящего все
Другие методы во всех случаях. При сравнении эволюцион-
ных моделей единственным ключевым критерием является во-
прос о том, насколько расчетные дистанции близки к истин-
ным эволюционным дистанциям. Безусловно, аналогичный во-
прос — насколько результаты филогенетического анализа с по-
мощью того или иного метода отражают истинные эволю-
ционные отношения между анализируемыми OTU — является
ключевым и при сравнении филогенетических методов, одна-
ко не единственным существенным вопросом. В ряде случаев,
пРежде всего при анализе большого числа OTU, ключевым
становится вопрос о требующемся для такого анализа вре-
мени.
134 Глава 4. Филогенетический анализ
Сравнению различных филогенетических методов посвяще(1
ряд работ, базирующихся либо на компьютерных симуляциях
либо на анализе случаев, когда эволюционные отношения между
OTU известны, скажем, анализе генетической информации, ц0.
лученной от инбредных линий лабораторных мышей или пасса,,
жей быстро мутирующих вирусов в клеточных культурах [Fitch
and Atchley, 1985; Atchley and Fitch, 1991; Hillis et al., 1992].
Однако интерпретация этих работ неоднозначна, а обобщения за-
труднены по ряду причин. Скажем, в неком исследовании, бази-
рующемся на известных филогенетических отношениях между
анализируемыми нуклеотидными последовательностями, было
показано, что филогенетическое дерево, построенное с помощью
метода МР, более близко к известным истинным отношени-
ям, чем дерево, построенное с помощью метода NJ. При этом
алгоритм метода МР базируется на анализе состояния нук-
леотидов в конкретных позициях и не использует (выбранные
исследователем) эволюционные модели, а алгоритм метода NJ —
анализирует нуклеотидные дистанции, рассчитанные согласно
выбранной исследователем эволюционной модели. Таким обра-
зом, возникает вопрос, свидетельствуют ли результаты этого
исследования о недостатках метода NJ по сравнению с методом
МР или же о неадекватности выбранного в исследовании метода
установления эволюционных дистанций.
Рассмотрим преимущества и недостатки описанных выше]
методов (кроме методов UPGMA и трансформированной дистан-J
ции, заведомо проигрывающих другим методам и в настоящее
время практически не используемых). Особый интерес пред-
ставляет сравнение дистанционного метода NJ и метода анализа
дискретных признаков МР, двух наиболее часто используемых
в эволюционном анализе методов.
При сравнении дистанционных методов и методов анализа
дискретных признаков вообще, большинство исследователей от-
дают предпочтение последним. Это определяется и результата-
ми исследований, в целом демонстрирующих большую надеж,
ность методов анализа дискретных признаков, и интуитивными
соображениями. Методы анализа дискретных признаков учи-
тывают состояние, распределение нуклеотидов (аминокислот)
в каждой позиции последовательностей отдельно, в то время
как дистанционные методы усредняют изменения по всем по-
зициям, сводят анализ многообразия индивидуальных измене-
ний к анализу одного параметра — эволюционных дистанШ111
между последовательностями. Однако отметим, что метод
4.6. Сравнение филогенетических методов 135
анализирует только состояние филогенетически информативных
позиций последовательностей, и не учитывает эволюционные
изменения в других позициях. В то же время изменения
в ДРУГИХ позициях также содержат филогенетически значи-
мую информацию, которая учитывается в дистанционных ме-
тодах.
Большая, в целом, надежность методов анализа дискрет-
ных признаков, безусловно, не является основанием для отказа
от использования дистанционных методов. Во многих случа-
ях возникающие в практической работе задачи эволюционного
анализа являются относительно простыми, и использование,
скажем, методов NJ и МР приводит к одинаковым результа-
там. При этом метод NJ занимает гораздо меньше компьютер-
ного времени. Кроме того, метод NJ позволяет использовать
и сравнивать различные эволюционные модели и просто и на-
дежно анализировать синонимичные и несинонимичные заме-
ны раздельно. При анализе данных ДНК-ДНК гибридизации,
рестрикционного анализа или анализа иммунной реактивности,
т. е. при отсутствии непосредственной информации о порядке
нуклеотидов (аминокислот) в анализируемых последовательно-
стях, могут использоваться только дистанционные методы, ме-
тоды анализа дискретных признаков в таких случаях неприме-
нимы.
Кроме того, проблемы, возникающие при несоблюдении мо-
дели молекулярных часов в эволюции анализируемой группы
последовательностей (разд. 5.3.4), проявляются гораздо сильнее
при использовании метода МР, чем NJ.
Возможность филогенетического метода предложить не толь-
ко одну, самую оптимальную, топологию дерева, но и альтерна-
тивные топологии (вторую оптимальную, третью и т. д.), и ста-
тистически сравнить различия между этими топологиями, без-
условно, является преимуществом метода. Такая возможность
предоставляется как методами анализа дискретных признаков,
Так и рядом дистанционных методов, например методом ME.
Идеальным является рассмотрение всех возможных топологий
Дерева для анализируемых последовательностей. Понятно, что
Такой анализ занимает значительное время и при большом
числе анализируемых последовательностей вообще практически
Неприменим. В этом плане использование методов повторных
Сборок, в частности бутстрэп-анализа, для статистической
^Ценки достоверности построенного методом NJ дерева является
встрой и надежной альтернативой.
136 Глава 4. Филогенетический анализ
4.7. Филогенетический анализ в таксономии,
фенетика и кладистика
В заключение этой главы рассмотрим роль и возможности фи.
логенетического анализа в создании эволюционной таксономии.
Обсужденные в разд. 4.1 различия между моно-, поли- и пара-
филетическими группами имеют ключевое значение для обосно-
вания и понимания принципов эволюционной систематики, клас-
сификации форм жизни. В основе любого подхода к систематике
лежит объединение форм жизни в иерархическую систему так-
сонов (ед.ч. — taxon, мн.ч. — taxa), начиная от низшего таксона,
вида (species), к высшему таксону, царству (kingdom).
Исторически систематика форм жизни в значительной степе-
ни базировалась на фенетическом подходе, фенетике (разд. 1.15).
Этот подход основан на объединении форм жизни в таксономи-
ческие группы, исходя из их форм жизни, морфологического
сходства между собой. В качестве примера применения фонети-
ческого подхода отметим отнесение к единому таксону — классу!
пресмыкающихся — современных черепах (анапсиды), ящериц
и змей (чешуйчатые лепидозавры) и крокодилов (все — диап-
сиды), а также ряд других современных и вымерших групп,
на основании ряда общих для этих групп признаков (рис. 4.17).
В то же время птицы, также имеющие диапсидный череп,
отнесены к другому классу. Класс пресмыкающихся является
парафилетической группой, одной из эволюционных ветвей, име-
ющей общее с пресмыкающимися происхождение — а именно
класс птиц («крокодилы — те же чтицы, только они не ле-
тают»).
Фенетический анализ не различает плезиоморфий (plesiomor-
phies) — признаков, унаследованных группой от общего пред-
ка, и потому филогенетически неинформативных, и апоморфий
(apomorphies) — признаков, приобретенных в результате эволю-
ции в одной или нескольких эволюционных линиях независи-
мо. Фенетический анализ не выявляет случаев конвергентной,
эволюции. Некий признак, возникший в результате конвер-
гентной эволюции различных форм жизни, при таком анализе
будет (неправомерно) расценен как объединяющий эти формы
жизни.
Принципиальным отличием кладистического подхода, кла-
дистики, является базирование таксономической системы не
на сходствах форм жизни, а на их общем эволюционном проис-
хождении. При этом базовым положением является выделение
4.7. Таксономия: фенетика и кладистика 137
Рис. 4.17. Эволюционные отношения между позвоночными. Указан
порядок дивергенций, но не масштаб эволюционных ветвей, ведущих к
современным группам позвоночных. Группа пресмыкающихся (черепа-
хи, змеи, ящерицы и крокодилы) является парафилетической группой,
поскольку не включает в себя птиц. Монофилетическая группа, включа-
ющая в себя птиц, выделена серым цветом. Примером полифилетиче-
ской группы является (гипотетическое) объединение млекопитающих
и птиц в одну таксономическую единицу
в таксоны только монофилетических групп, поскольку только
в отношении таких групп можно формулировать содержатель-
ные обобщения. Группа пресмыкающихся будет являться мо-
нофилетической, если включить в ее состав птиц — например,
6 качестве одной из подгрупп архозавров, наряду с крокодилами.
Альтернативно возможно расформирование класса пресмыкаю-
щихся и выделение эволюционных ветвей черепах, лепидо-
Знвров, крокодилов (вместе с птицами или отдельно от птиц)
в отдельные классы (см. рис. 4.17).
138 Глава 4. Филогенетический анализ
Надкласс рыб также является парафилетической группой
поскольку иначе в нее должен входить и предок наземных
животных, четвероногих, который, очевидно, рыбой не являлся;
Примеры моно-, пара- и полифилетических групп приведены
на рис. 4.17.
В идеале систематика форм жизни должна быть направлена
на выявление и отнесение к различным таксонам лишь моно-
филетических групп. Одной из ключевых задач современной
таксономии является реорганизация таксонов, создание системы
чистых клад, монофилетических групп, т. е. филогенетических
кластеров. Такие понятия, как беспозвоночные или хладнокров-
ные, основаны на остаточном принципе — отсутствии того или
иного признака у группы организмов. Содержательность оста-
точных определений крайне низка, если есть вообще. Непри-
емлемость полифилетических групп в качестве таксонов стала
понятной уже в начале XX века, проблема же парафилии была
осознана значительно позже и полностью не решена до сих
пор. До сегодняшнего времени в классификациях многих форм
жизни в качестве таксонов могут присутствовать пара- и даже!
полифилетические группы. Во многих случаях это связано не со
стремлением сохранить пара- и полифилетические таксоны, но
с отсутствием детальных данных об эволюционных отношениях
между различными формами жизни, в связи с чем монофилети-
ческие группы не выявлены. По мере получения данных о фило-
генетических отношениях между формами жизни, их классифи-
кация пересматривается, уточняется. Так, в разд. 1.15 уже рас-
сматривался пример неправомерности выдг чения группы копыт-
ных животных (надотряд Ungulata): парнокопытные и непар-
нокопытные животные не являются близкими родственниками!
не образуют монофилетической группы. По мере накопления
филогенетических данных классификации различных групп жи-
вых систем подвергаются сомнениям, их приходится неоднократ-
но проверять и уточнять, пересматривать. В качестве примера
отметим, что до недавнего времени не было никаких сомнений
касательно классификации морских свинок: они считались пред-
ставителями самого многочисленного отряда млекопитающих,
отряда грызунов (Rodentia). Однако эта классификация была
подвергнута серьезным сомнениям: проведенный эволюционный
анализ генетических последовательностей морских свинок пока-
зал, что они, возможно, являются не родственниками крыс и мьг!
шей, а значительно более древней группой, отпочковавшейся
от общего дерева еще до разделения эволюционных линий крЫс
4.7. Таксономия: фенетика и кладистика 139
0 мышей и, скажем, приматов [Graur et al., 1991; Graur et al.,
1992; Li et al., 1992; D’Erchia et al., 1996]. Последующий анализ
подтвердил правильность традиционной классификации морских
съинок как представителей отряда грызунов [Hasegawa et al.,
1992; Robinson-Rechavi et al., 2000; Lin et al., 2002a; Lin et al.,
2002b]. Вообще, классификация животных, традиционно относи-
мых к отряду грызунов, вызывает много дискуссий и сомнений.
Проблемы пара- и полифилии проявляются и в клас-
сификации вирусов. В качестве примера рассмотрим клас-
сификацию парвовирусов позвоночных — вирусов подсемейства
Parvovirinae семейства Parvoviridae. Традиционная классифи-
кация этого подсемейства базировалась на способности виру-
сов к автономной репликации или зависимости от вирусов-
помощников и спектре хозяев. Соответственно выделялись ро-
ды Dependovirus — неспособные к автономной репликации, за-
висимые от вирусов-помощников аденоассоциированные виру-
сы, ААВ, Erythrovirus — вирусы человека (В19) и приматов
и Parvovirus — все остальные автономные парвовирусы позво-
ночных. Однако филогенетический анализ полноразмерных гено-
мов вирусов этого подсемейства продемонстрировал необоснован-
ность такой классификации (рис. 4.18) [Lukashov and Goudsmit,
2001]. В частности, парвовирусы птиц, относившиеся согласно
принятой классификации к роду Parvovirus, являются близ-
кородственными ААВ, но не другим автономным вирусам. Та-
ким образом, отнесение парвовирусов птиц к роду Parvovirus
приводит к тому, что этот род становится парафилетическим.
В настоящее время классификация парвовирусов позвоночных
приведена в соответствие с эволюционными данными.
Обсужденные выше различия между фенетическим и клади-
стическим подходами принципиальны. При практическом ана-
лизе, однако, может возникать неопределенность, поскольку ряд
филогенетических методов невозможно охарактеризовать как ис-
ключительно фенетические или кладистические. Соответствен-
но филогенетические деревья могут иметь и промежуточную
’'Южду фенограммой и кладограммой топологию. Из рассмот-
ренных в этой главе методов, метод UPGMA является наибо-
лее «чистым» фенетическим методом, а метод максимальной
экономии — наиболее «чистым» кладистическим методом. Дру-
гие обсужденные выше методы сложнее классифицировать как
Фенетические или кладистические. Например, метод присоеди-
нения соседей принято относить к фенетическим методам. Тем
Не менее это утверждение не является абсолютно верным: метод
140 Глава 4. Филогенетический анализ
100
100
Традиционно:
□ Dependovirus
Q Erythrovirus
остальные - Parvovirus
100
100
PorcinePV
---AMDV
MousePV-1
MousePV-lb
MousePV-lc
Hamster PV-lc
---LuIII
MVM
KilratPV
-Hl
RatPV
CaninelPV
FelinePV
^MEV
I_RaccoonPV
AAV-5
AAV 4
AAV-2
AAV 1
AAV 6
AAV-3
100 г BarbduckPV
MuscduckPV
GoosePV
парвовирусы
грызунов,
плотоядных и
свиней
AAV н
парвовирусы
птиц
AAV ЗВ|
автономные
парвовирусы
приматов,
бурундуков и
коров
0.05
Рис. 4.18. Эволюционные отношения между парвовирусами позвоноч-
ных. Традиционная классификация парвовирусов не отражала эволю-
ционных отношений между ними. По Лукашову и Годсмиту [Lukashov
and Goudsmit, 2001]
действительно базируется на анализе сходства признаков (разли-
чия признаков, генетической дистанции между последователь-
ностями), однако не устанавливает прямой зависимости межЛУ
генетической дистанцией и эволюционными отношениями.
Глава 5
ОТДЕЛЬНЫЕ ЗАДАЧИ
ЭВОЛЮЦИОННОГО АНАЛИЗА
5.1. Рекомбинационный анализ
рекомбинация представляет собой особый вид эволюционного
изменения генетической информации, при котором считывание
дочерней последовательности происходит с двух родительских
последовательностей, матриц. В приведенном ниже примере
последовательность 2 является рекомбинантной между после-
довательностями 1 и 3: считывание начала дочерней последова-
тельности 2 (позиции 1-19) происходило с последовательности 1,
а ее конца (позиции 20-30) — с последовательности 3:
111111111122222222223
123456789012345678901234567890
1 ATAGTACTTGGCTAGAGTCAGATTAGGACT
I I I I I I I I I I I I I I I I I I I *********|*
2 ATAGTACTTGGCTAGAGTCTCTAGTCCTCA
****|************* | | | | | | | | | | | |
3 GAGTTCGACCAAAGCTAACTCTAGTCCTCA
Точкой рекомбинации (recombination point) здесь является
позиция 19. Последовательность, имеющую рекомбинантное про-
исхождение, иначе называют мозаичной последовательностью
(mosaic), или рекомбинантной формой. Отметим, что описыва-
емый пример, в котором две родительские последовательности
столь различны между собой, является заведомо искусственным,
приведенным для более ясного представления результатов ре-
комбинации. В реальных биологических процессах рекомбина-
ция требует гораздо большего сходства родительских последова-
тельностей.
Естественно, что в процессе считывания последовательно-
сти 2 с каждой из двух родительских последовательностей
^огли происходить и ошибки, в том числе замены нуклеотидов.
Кроме того, рекомбинация могла произойти и некое время тому
Чэзад, между предками последовательностей 1 и 3, после чего
э®олюция линий 1-3 происходила независимо друг от друга.
Соответственно участки рекомбинантной последовательности 2
142 Глава 5. Отдельные задачи эволюционного анализа
могут иметь отличия от соответствующих участков родитель,
ских эволюционных линий. Такой пример приведен ниже:
111111111122222222223
123456789012345678901234567890
1 ATAGTACTTGGCTAGAGTCAGATTAGGACT
111*11111*1111111|**********|*
2 ATACTACTTCGCTAGAGTGTCTAGTCCTCA
****|****|********* I I I I I I I I I I I
3 GAGTTCGACCAAAGCTAATTCTAGTCCTCA
В данном случае последовательность 2 является результатом
рекомбинации между последовательностями 1 и 3, после чего
эволюция линий 1-3 шла независимо друг от друга, и произо-
шли нуклеотидные замены в позициях 4 и 10 последовательно-'
стей 1 или 2.
Кроме рекомбинаций существует и целый ряд других био-
логических процессов, при которых дочерняя последователь-
ность, дочерний геном получает генетическую информацию не от
одного, а от нескольких родительских последовательностей.
К таким феноменам относятся реассортация, горизонтальный
перенос генов, встраивание ретропоследовательностей, включая
ретровирусы и т. д. [Koonin et al., 2001; Deininger and Batzer,
2002; Deininger et al., 2003; Koonin, 2003]. Биологические меха-
низмы этих явлений отличаются друг от друга, однако методы
идентификации таких событий практически одинаковы.
При построении филогенетического дерева (гл. 4) филогене-
тические методы не принимают во внимание возможное ре-
комбинантное происхождение анализируемых последовательно-
стей, не учитывают их возможного мозаицизма. Соответственно
использование любого филогенетического метода для анали-
за группы последовательностей 1-3 по всей их длине (пози-
ции 1-30) не позволит выявить участки последовательности 2,
в которых она родственна последовательности 1 или 3, и при-
ведет к ошибочной топологии дерева (рис. 5.1, а) и длинам его
ветвей. Таким образом, рекомбинантное происхождение (оД'
ной из) анализируемых последовательностей препятствует ре-
конструкции истинных эволюционных отношений между ними
при использовании любого филогенетического метода. Соответ-
ственно задачей исследователя является выявление возможны*
рекомбинантных форм в анализируемой группе последователь-
ностей и учет этого факта при филогенетичесом анализе. ТаК,
5.1. Рекомбинационный анализ 143
Рис. 5.1. Выявление рекомбинантного происхождения последовательно-
сти 2. Для приведенных в тексте последовательностей 1-3 построены
филогенетические деревья: на основе анализа всех позиций 1-30 (а)
и на основе раздельного анализа позиций 1-19 (б) и 20-30 (в). Отметим
изменение порядка ответвления последовательности 2
раздельный филогенетический анализ позиций 1-18 (рис. 5.1, б)
и 19-30 (рис. 5.1, в) с помощью того же филогенетического мето-
да приведет к правильной реконструкции эволюционных отно-
шений между участками последовательностей 1-3. Свидетель-
ством рекомбинантного происхождения мозаицизма последова-
тельности 2 будет являться изменение ее положения в филогене-
тических деревьях при анализе различных участков. Отметим,
что, естественно, некая рекомбинантная форма необязательно
является рекомбинантной между другими последовательностя-
ми анализируемой группы, ее родительские последовательности
Могут быть и не включены в анализ.
Для идентификации рекомбинаций предложен ряд подхо-
дов [Posada and Crandall, 2001а]. Наиболее распространенный
Метод установления рекомбинантного происхождения анализи-
руемой последовательности — метод ее сканирования с помощью
скользящих, или движущихся, окон (sliding or moving windows),
предположим, что мы анализируем последовательность 1, ко-
т°Рая, возможно, является рекомбинантной между последова-
тельностями 2 и 3. При сканировании эти три выровненные
Последовательности разбиваются на участки, скользящие окна,
^Иной, скажем, в 200 нуклеотидов и перекрывающиеся друг
144 Глава 5. Отдельные задачи эволюционного анализа
с другом на 150 нуклеотидов (шаг в 50 нуклеотидов). Таким об.
разом, сначала будет проанализирован участок 1-200 этих после,
довательностей, затем участок 51-250, затем участки 101-300
151-350, 201-400 и т. д. Для каждого такого окна, можно рас.
считать генетические дистанции между последовательностью (
и последовательностями 2 и 3, и изобразить их в виде гра.
фика. Можно и построить филогенетические деревья отдельно
для каждого окна и провести анализ этих деревьев методом
случайных выборок. Группирование участков последовательно-
сти 1 с соответствующими участками последовательности 2 или
3 можно изобразить в виде графика, отражающего значения
бутстрэп-анализа (процедура бут-сканирования, bootscan). Одной
из наиболее распространенных компьютерных программ для вы-
явления рекомбинаций является программа SimPlot (разд. 6.6).
Для демонстрации этих подходов к выявлению ре-
комбинаций, рассмотрим пример идентификации первого
установленного случая рекомбинации в семействе вирусов
Parvoviridae [Lukashov and Goudsmit, 2001].
Геном парвовирусов представлен одноцепочечной молекулой
ДНК длиной около 5000-6000 нуклеотидов и содержит две
открытые рамки считывания: orfl, кодирующую два неструк-
турных белка — NS-1 и NS-2, и orf2, кодирующую два или три
структурных белка — VP-1 — VP-3. К парвовирусам грызунов,
представителям рода Parvovirus этого семейства, относятся ви-
русы мышей — MPV-1, 1b, 1с и MVM, хомяков — HaPV, крыс —
RPV и KPV, а также вирусы неизвестных хозяев — LuIII и Hl-
Филогенетические отношения между парвовирусами грызу-
нов представлены на рис. 5.2, в качестве внешней группы ис-
пользован парвовирус кошек — FPV. Анализ полноразмерного
генома показывает, что вирусы мышей MPV группируются вме-
сте с вирусом хомяков HaPV и отдельно от другого вируса
мышей, MVM (рис. 5.2, а). Эволюционная линия вируса Lull!
отпочковывается от дерева между линиями MPV-HaPV. Тако-
ва же и топология дерева, построенного на основании анализа
последовательностей orf2 этих вирусов (рис. 5.2, в).
Однако филогенетический анализ последовательностей orfl
этих вирусов приводит к дереву с другой топологией (рис. 5.2, б)-
При этом анализе, вирусы MVP и HaPV группируются вместе
с вирусами MVM, а линия вируса LuIII является внешне15
по отношению к этой группе. Эти результаты дают основание
предположить, что в эволюционной истории парвовирусов грЫ'
зунов произошла рекомбинация. Поскольку филогенетически6
5.1. Рекомбинационный анализ 145
0.05
MVM J02275
----LuIII М81888
HaPV U34255
MPV-lc U34254
MPV-lbU34253
MPV-1 U12469
- KPV U79033
— H-1PVX01457
RPV-1 AF036710
------FPV M38246
95
100Г
77j[
loo Г
MPV-1 U12469
MPV-lb U34253
- MPV-lc U34254
- MVM J02275
---HaPV U34255
100
100
----------LuIII M81888 <
r KPV U79033
WCp— Hl X01457
RPV-1 AF036710
FPV M38246
6
I-----------1
0.05
I-------1
0.05
рис. 5.2. Идентификации первого установленного случая рекомбинации
8 семействе вирусов Parvoviridae. При анализе полноразмерного генома
(о), orfl (б) и orf2 (в) этих вирусов выявляется различная топология
Деревьев. Для (б) и (в) различия в топологии выделены. По Лукашову
11 Годсмиту [Lukashov and Goudsm.it, 2001]
146 Глава 5. Отдельные задачи эволюционного анализа
отношения, т. е. порядок дивергенций, между всеми вирусами
кроме группы MPV-HaPV, одинаковы в обоих участках генома
то наиболее вероятным сценарием является рекомбинантное
происхождение группы вирусов MPV-HaPV, при котором их
orfl происходит от (предка) MVM, a orf2 — от (предка) Lull!
Однако на основании имеющихся результатов нельзя исключить
и рекомбинантного происхождения вируса MVM, когда его orfl
происходит от (предка) группы MPV-HaPV, a orf2 — от неиз-
вестного вируса.
Вообще, установление того, какая именно из анализируе-
мых последовательностей имеет рекомбинантное происхожде-
ние, возможно только при достоверно известных обоих ее пред-
ках. Рассмотрим пример с тремя последовательностями:
111111111122222222223
123456789012345678901234567890
1 ATAGTACTTGGCTAGAGTCAGATTAGGACT
111*11111*11111111 **********|*
2 ATACTACTTCGCTAGAGTGTCTAGTCCTCA
I I I I1*1 I I I I I I I I I I I I I I I I I I 1*1 I I
3 ATACTCCTTCGCTAGAGTGTCTAGTCATCA
Здесь можно предполагать рекомбинантное происхождение
либо последовательности 1 (рекомбинация между группой после-
довательностей 2-3 и неизвестной последовательностью), либо
группы последовательностей 2-3 (рекомбинация между после-
довательностью 1 и неизвестной последовательностью). Отме-
тим также, что для установления рекомбинантного происхож-
дения некой последовательности участки этой последовательно-
сти, имеющие различное эволюционное происхождение, должны
быть достаточно протяженными, а различие между родительски-
ми последовательностями — достаточно большим. Рассмотрим
следующие случаи:
111111111122222222223
123456789012345678901234567890
1 ATAGTACTTGGCTAGAGTCAGATTAGGACT
111*11111*1111111111111Н11*1*
2 ATACTACTTCGCTAGAGTCAGATTAGGTCA
****|*********************||||
3 GAGTTCGACCAAAGCTAATTCTAGTCGTCA
5.1. Рекомбинационный анализ 147
111111111122222222223
123456789012345678901234567890
1 ATAGTACTTCGCTAGAGTCAGATTAGGACT
I I I I I I I I I I I I I I I I I I I I I1*1 I I 1*1*
2 ATAGTACTTCGCTAGAGTCAGAGTAGGTCA
I I1*1 I I I1*1 I I I I I I I I I I I I I I I I I I I
3 ATACTACTTAGCTAGAGTCAGAGTAGGTCA
В обоих этих случаях можно предполагать, что последова-
тельность 2 является результатом рекомбинации между после-
довательностями 1 и 3. Однако в обоих случаях статистическое
подтверждение этого предположения будет невозможно: в пер-
вом случае из-за недостаточной длины участка последовательно-
сти 2, возможно, происходящей от последовательности 3, а во
втором случае — из-за недостаточной генетической дистанции
между возможными родительскими последовательностями.
Возвращаясь к примеру рекомбинации среди парвовирусов
грызунов, рассмотрим метод сканирования последовательностей
для выявления рекомбинаций. На рис. 5.3 представлены резуль-
таты сканирования генома парвовирусов грызунов с помощью
программы Simplot (разд. 6.6). Тестируемыми (query) в этом ана-
лизе являются последовательности геномов вирусов MPV-HaPV
(они рассматриваются как единая группа последовательностей),
а их сравнение проводится с последовательностями геномов ви-
русов MVM, LuIII и использованной в качестве внешней группы
последовательностью генома вируса Н1.
Все эти последовательности поделены на перекрывающиеся
с шагом в 50 нуклеотидов скользящие окна (рис. 5.3, а, б —
длина окна 500 нуклеотидов, рис. 5.3, в—250 нуклеотидов). На
Рис. 5.3, а, сравнение тестируемой группы с другими последо-
вательностями проведено путем сравнения их генетического
сходства. На рис. 5.3, б, в, для каждого окна построены филогене-
тические деревья, а изображенные графики отражают значения
бутстрэп-анализа для группирования тестируемой группы с каж-
дой из других последовательностей.
Как видно, на участке 1-2500 группа MPV-HaPV имеет
высокий процент генетического сходства с последовательностью
MVM и группируется с этой последовательностью при фило-
гснетическом анализе. Однако при анализе участка 3000-4500,
148 Глава 5. Отдельные задачи эволюционного анализа
SimPlot - Query: MPV-HaPV
a Similarity
...................................................—
50 1.000 1.500 2.000 2.500 3.000 3.500 4.000 4.500 5.000
Position (bp)
Window: 500 bp, Step: 50 bp, GapStrip: On, J-C Correction: On
BootScan - Query: MPV-HaPV
6 % of Permuted Trees
Position (bp)
Window: 500 bp. Step: 50 bp, GapStrip: On, Reps: 100, Kimura, T/t: 2.0, NEIGHBOR
BootScan - Query: MPV-HaPV
в % of Permuted Trees
....MVM
....LuIII
----Hl
Position (bp)
Window: 500 bp, Step: 50 bp, GapStrip: On, Reps: 100, Kimura, T/t: 2.0, NEIGHBOR
5.1. Рекомбинационный анализ 149
группа MPV-HaPV более сходна с последовательностью LuIII
и группируется с ней в филогенетическом дереве. Последующий
участок группы MPV-HaPV (после 4500 нуклеотидов) снова
ймеет большее сходство и группируется с последовательностью
MVM.
Отметим, что при уменьшении длины окон с 500 до 250
(рис. 5.3, б, в) значения бутстрэп-анализа при группировании
участков MPV-HaPV с MVM и LuIII снижаются. Более того,
при этом обнаруживается участок 700-900, в котором последова-
тельности MPV-HaPV равновероятно группируются с последо-
вательностями MVM и Н1. Однако недостаточная длина участ-
ка и невысокие значения бутстрэп-анализа не дают оснований
предполагать, что этот участок группы MPV-HaPV является
результатом рекомбинации с (предком) Н1. Подобный результат
может являться и следствием малой генетической дистанции
между всеми анализируемыми последовательностями на этом
участке (т. е. этот участок — консервативен).
Описаны и более сложные случаи рекомбинации, при кото-
рых мозаичные последовательности имеют не одну-две, а больше
точек рекомбинации и более двух родительских последователь-
ностей.
Рис. 5.3. Анализ геномов парвовирусов грызунов методом сканирова-
ния последовательностей для выявления рекомбинации. Тестирование
сгруппированных геномов вирусов MPV-HaPV проводится путем их
сравнения с последовательностями геномов вирусов MVM, LuIII и ис-
пользованной в качестве внешней группы последовательностью генома
вируса Ш. Сравнение тестируемой группы и с другими последова-
тельностями проведено путем сравнения их генетического сходства (а)
Или с помощью филогенетического анализа (б, в). Горизонтальная ось
°тражает позиции генома, вертикальная — генетическое сходство (а)
Или значения бутстрэп-анализа (б, в). Во всех случаях анализ проведен
Для перекрывающихся с шагом в 50 нуклеотидов окон, длина которых
составляла 500 (а, б) или 250 (в) нуклеотидов. Отметим снижение
значений бутстрэп-анализа при уменьшении длины окон с 500 до
250. Анализ проведен с помощью программы SimPlot. По Лукашову
11 Годсмиту [Lukashov and Goudsmit, 2001]
150 Глава 5. Отдельные задачи эволюционного анализа
5.2. Анализ нуклеотидного и аминокислотного
состава и использования кодонов
5.2.1. Смещения нуклеотидного состава
Ранее в этой книге (разд. 1.7, 3.2.1, 3.2.3, 3.2.6 и др.) мы
неоднократно отмечали, что процентное содержание каждого
из четырех нуклеотидов в некой последовательности (ее нуклео-
тидный состав, нуклеотидная композиция) может значительно
отличаться от 25%. Эти смещения нуклеотидного состава, пред-
почтение того или иного нуклеотида за счет другого необходимо,
в частности, принимать во внимание при филогенетическом
анализе нуклеотидных последовательностей. Так, если в неких
последовательностях имеются смещения нуклеотидного состава
(скажем, преобладание А за счет Т), то этот факт следует
учитывать при выборе эволюционной модели (гл. 3), оптимально
описывающей эволюцию этих последовательностей.
Понятно, что установление нуклеотидного состава некой
последовательности или группы последовательностей является
тривиальной задачей: нужно просто подсчитать процентное со-
держание каждого из четырех нуклеотидов. Далее, с помощью
стандартного статистического аппарата можно установить, до-
стоверно ли отличается содержание каждого нуклеотида от ожи-
даемых 25%, или достоверно ли отличается содержание каждого
нуклеотида в некой последовательности (группе последователь-
ностей) от другой последовательности (группы последовательно-
стей).
Можно учитывать и содержание каждого из четырех нук-
леотидов в отдельности, и суммарное содержание нуклеоти-
дов А и Т (образующих слабую связь, А+Т-содержание, или
АТ-содержание) в сравнении с суммарным содержанием нуклео-
тидов G и С (образующих сильную связь, G+C-содержание, или
GC-содержание). Для кодирующих последовательностей можно
учитывать и содержание различных нуклеотидов в каждой
из трех позиций кодонов — например, GC-содержание в тре-
тьей позиции кодона, ССЗ-содержание. Поскольку большинство
замен в третьей позиции кодона являются синонимичными
(разд. 1.6.2), то они не будут подвергаться влиянию факторов
естественного отбора, действующих на уровне белка. Можно
подойти и более строго, рассчитав GC-содержание в третьей
синонимичной позиции (ССЗз-содержание), для чего из расчетов
исключают кодоны метионина и триптофана. Поскольку эти две
5.2. Анализ нуклеотидного и аминокислотного состава 151
аминокислоты кодируются только одним кодоном (разд. 1.6.2,
табл. 1.4), то любая замена даже в третьей позиции кодонов
метионина и триптофана будет несинонимичной. Понятно, что
все эти расчеты процентного содержания нуклеотидов являются
тривиальной задачей.
Аналогично не составляет труда подсчитать и процентное
содержание различных аминокислот в белке, его аминокислот-
ный состав. Для белка достаточно понятно, что процентное
содержание аминокислот различных классов (разд. 1.3) будет
связано с функциональными характеристиками этого белка. Так,
понятно, что в трансмембранном домене некоего белка будут
доминировать гидрофобные аминокислоты, в каталитическом
центре фермента будут представлены полярные аминокисло-
ты и т. д.
Однако существенным вопросом является установление при-
чин, эволюционных процессов, ведущих к смещениям нуклео-
тидного состава. В частности, GC-содержание в геномах бакте-
рий варьирует в широких пределах: от 25 до 75% [Sueoka et al.,
1959; Sueoka, 1964]. При этом более родственные между собой
бактерии имеют и сходное GC-содержание [Muto et al., 1986].
При анализе нуклеотидного состава следует учитывать целый
ряд факторов, обсуждаемых далее в этом разделе.
Прежде всего, как обсуждалось ранее, любая наблюдаемая
нуклеотидная замена является результатом, во-первых, ошиб-
ки при репликации генетического материала (разд. 1.5) и, во-
вторых, закрепления этой замены (разд. 1.12). Таким образом,
при анализе наблюдаемого смещения нуклеотидного состава
в некой последовательности, скажем, преобладания А за счет G,
необходимо учитывать различные факторы.
Во-первых, возможно, что при репликации этой последо-
вательности преимущественно происходят ошибки, при кото-
рых G меняется на A (G—>А гипермутация, hypermutation,
мутационное смещение, mutational bias, мутационное давление,
mutational pressure). Причинами этого явления могут являться
особенности ферментов, участвующих в репликации и проверке
ошибок репликации: скажем, некая полимераза преимуществен-
но меняет G на А, или механизм проверки ошибок при репли-
кации (proofreading activity) неэффективно исправляет замены
G на А. Кроме того, необходимо учитывать внутриклеточное
содержание, пул различных нуклеотидов (их трифосфатов): ес-
ли, скажем, в клетке мало трифосфата G и много трифосфата
А, то можно ожидать, что при необходимости присоединить
152 Глава 5. Отдельные задачи эволюционного анализа
G в процессе репликации генома будет с некой (относительно
большой) вероятностью ошибочно присоединен А.
Во-вторых, возможно, что различные ошибки равновероятны
но последовательности с большим содержанием А и меньшим
содержанием G (т.е. замены G на А) имеют селективное преиму.
щество и предпочтительно отбираются под влиянием факторов
естественного отбора.
Таким образом, единого универсального механизма, который
объяснял бы наблюдаемые в природе смещения нуклеотидного
состава различных последовательностей, не существует. В зна-
чительной степени дискуссия о причинах смещений нуклео-
тидного состава является составной частью общей дискуссии
между неодарвинистами и сторонниками нейтральной теории
эволюции (разд. 1.14): определяются ли эти причины факторами
естественного отбора или же предпочтительными типами замен?
Как отмечалось выше, GC-содержание у различных бактерий
значительно различается. Напомним (разд. 1.7), что повышение
GC-содержания приводит к термодинамически более стабильной
структуре ДНК. Для геномов ряда термофильных бактерий,
живущих при очень высоких температурах, показано высо-
кое GC-содержание [Argos et al., 1979; Kagawa et al., 1984;
Nishiyama et al., 1986; Kushiro et al., 1987]. Это проявляется
и в большом процентном содержании в белках этих бактерий
аминокислот, кодоны которых имеют большое GC-содержание
(аланин и аргинин) при малом содержании аминокислот, ко-
доны которых имеют большое АТ-содержание (серин и ли-
зин), и в преимущественном содержании G и С в третьей
позиции кодонов. Так, в геноме бактерии Thermus aquaticus,
полимераза которой используется в ПЦР (Tag-полимераза), GC-
содержание достигает 67%, а ССЗ-содержание — 90% [Ono et al.,
1990; Kaplan and Steitz, 1999]. Безусловно, этот факт может
быть интерпретирован как результат влияния естественного
отбора, направленного на повышение приспособленности этих
бактерий к высокой температуре. Однако дискуссия о воз-
можном влиянии других эволюционных механизмов, включая
мутационное давление, ведущих к столь большим различиям
в GC-содержании у бактерий, не закончена [Sueoka, 1964; Muto
and Osawa, 1987].
В этой дискуссии аргументы сторонникам идеи мутацион-
ного давления предоставляют, в частности, исследования ви-
русов. Для вирусных геномов вообще также характерны зна-
чительные вариации в GC-содержании. При этом в отличие
5.2. Анализ нуклеотидного и аминокислотного состава 153
от бактерий значительные различия в GC-содержании мо-
гут наблюдаться даже у близкородственных вирусов в пре-
делах одного семейства. Так, для ДНК-содержащих вирусов,
GC-содержание у иридовирусов (семейство I ridoviridae) варьи-
рует от 27 до 55% [Tsai et al., 2007], у поксвирусов (семей-
ство Poxviridae)— от 26 до 64% [Shackelton et al., 2006]. При
учете только третьих синонимичных позиций геномов поксви-
русов, выявляются еще большие различия: ССЗз-содержание
в этом семействе варьирует от 15 до 82% [Shackelton et al.,
2006]. Значительные вариации наблюдаются и в семействах
рНК-содержащих вирусов: так, у астровирусов (Astroviridae)
ОСЗ-содержание в различных открытых рамках считывания
варьирует от 36 до 56%, различия в ОСЗз-содержании — еще
больше [van Hemert et al., 2007].
Значительные смещения нуклеотидного состава выявлены
и среди ретровирусов (семейство Retroviridae) [Berkhout and
van Hemert, 1994; Bronson and Anderson, 1994; van Hemert
and Berkhout, 1995; Zsiros et al., 1999; Berkhout et al., 2002].
Так, GC-содержание в геномах ретвовирусов варьирует от 38
до 54%, а, скажем, содержание нуклеотида А — от 24% (ви-
рус саркомы Рауса) до почти 40% (ВИЧ-1). Для ВИЧ-1 экс-
периментально показано, что при его репликации, на этапе
обратной транскрипции его генома (вирусная РНК —> прови-
русная ДНК), обратная транскриптаза преимущественно делает
ошибки, заменяя G на Т. Соответственно в итоговом дочернем
вирусном геноме (после этапа провирусная ДНК —» вирусная
РНК), будет наблюдаться повышенное число замен G на А:
G—»А гипермутация [Vartanian et al., 1991; Vartanian et al.,
1994; Berkhout et al., 2001]. Таким образом, предпочтительное
использование нуклеотида А в геноме ВИЧ-1 можно объяснить
не только действием естественного отбора, но и особенностями
Репликации вируса, что является аргументом в поддержку тео-
рии нейтральной эволюции.
По сравнению с вирусами и бактериями GC-содержание
в геномах высших организмов гораздо более единообразно.
Так, например, GC-содержание в геномах позвоночных (подтип
Vertebrata) варьирует в узких пределах от 35 до 45% [Sueoka,
1964], что меньше вариаций в пределах одного семейства виру-
сов. Для высших организмов выявлена значительная неравно-
мерность GC-содержания в различных участках генома. В гено-
мах высших организмов содержатся продолжительные (длиной
в десятки и сотни тысяч нуклеотидов) участки, характеризу-
154 Глава 5. Отдельные задачи эволюционного анализа
ющиеся гораздо более высоким и гораздо более низким, по срав-
нению со средним, GC-содержанием — тяжелые и легкие изо-
хоры соответственно (heavy and light isochors) [Bernardi et al
1985; Bernardi, 1993; Bernardi, 2000a; Bernardi, 2000b]. Содержа-
ние генов гораздо (до 20 раз) выше в тяжелых изохорах. Таким
образом, GC-содержание в генах высших организмов значитель-
но выше GC-содержания в геномах высших организмов. Для
объяснения этого феномена предложены гипотезы, основанные
на идеях как естественного отбора, так и мутационного давле-
ния.
Особый интерес представляет анализ не только суммарного
содержания G и С (и А и Т), но и сравнительное содержание
G относительно С (и А относительно Т). Содержание G относи-
тельно С называют GC-смещением (GC-skew) и рассчитывают по
формуле (G—C)/(G+C). АТ-смещение рассчитывают аналогично:
(А — Т)/(А + Т).
Для геномов дцДНК-содержащих вирусов и бактерий по-
казано относительно низкое содержание С по сравнению с G
в лидирующей цепочке, и относительно высокое содержание
С — в запаздывающей цепочке [Lobry, 1996; Grigoriev, 1999;
Frank and Lobry, 1999; Tillier and Collins, 2000; Sernova and
Gelfand, 2008]. Причины этого явления, называемого асиммет-
рией, продолжают обсуждаться, а само явление используется
для установления точек инициации репликации.
До сих пор мы говорили о содержании отдельных нуклео-
тидов — 35% А, 30% Т, 20% G, 15% С — или сумм содержания
нуклеотидов, GC-содержание — 35%. Однако можно учитывать
и содержание различных динуклеоп дов: как часто в анализи-
руемой последовательности Т находится, скажем, сразу после
А, и как часто А находится сразу после Т. Эти динуклеотиды
обозначают АрТ (А-фосфат-Т) и ТрА соответственно, чтобы
содержание, частоту встречаемости динуклеотидов не путать
с АТ-содержанием, суммой содержания А и Т.
Понятно, что, зная частоту встречаемости каждого нук-
леотида в отдельности, можно легко (перемножением частот
встречаемости) рассчитать ожидаемую частоту встречаемости
любого динуклеотида. При этом, если нет эволюционных огра-
ничений, то, независимо от АТ-содержания, процент динуклео-
тидов АрТ и ТрА в анализируемой последовательности дол-
жен быть одинаковым: большим или меньшим, в зависимости
от АТ-содержания, но одинаковым. Однако существуют эволю-
ционные факторы, действующие и на уровне динуклеотидов.
5.2. Анализ нуклеотидного и аминокислотного состава 155
Особое внимание уделяется анализу частоты встречаемо-
сти динуклеотида CpG, по сравнению с частотой встречаемо-
сти динуклеотида GpC. CpG — наиболее редкий динуклеотид
в геномах практически всех исследованных форм жизни (кро-
ле ДНК-содержащих вирусов с большими геномами) [Shpaer
and Mullins, 1990; Burge et al., 1992; Karlin et al., 1994;
De Amicis and Marchetti, 2000; Jabbari and Bernardi, 2004;
Shackelton et al., 2006]. Так, его содержание в геномах позво-
ночных составляет порядка 20% от ожидаемого (рассчитанного
на основе GC-содержания).
Общепринятое объяснение столь низкой встречаемости ди-
нуклеотида CpG связано с тем фактом, что в геномах мно-
гих позвоночных (и эукариот вообще, в отличие от бак-
териальных геномов) до 90% всех цитозинов в динуклео-
тиде CpG метилированы клеточными метилтрансферазами,
а неспаренный метилированный цитозин (5-метилцитозин) ча-
сто подвергается деаминированию и превращается в тими-
дин [Scarano et al., 1967; Cooper, 1983; Beutler et al., 1989;
Kress et al., 2001; Jabbari and Bernardi, 2004]. В данном
случае мы имеем дело с нуклеотидной заменой, являющей-
ся следствием не ошибки при репликации генома, а постре-
пликационной модификации генома и последующих биохими-
ческих процессов. Метилирование цитозина является одним
из ключевых механизмов регуляции экспрессии генов как пу-
тем прямого предотвращения связывания с факторами тран-
скрипции, так и изменением структуры хроматина и взаи-
модействий с гистонами. Метилирование цитозина в промо-
торах может приводить к сайленсингу (silencing) генов. Для
ряда онкогенов показана связь гипометилирования цитозина
в динуклеотидах CpG с повышенной экспрессией этих онко-
генов [Jones et al., 1992b; Jones and Laird, 1999]. У ретро-
вирусов метилирование провирусной ДНК снижает экспрес-
сию генов. Активация ретровирусов может быть вызвана до-
бавлением 5-азацитидина, снижающего уровень метилирова-
ния [Hoffmann et al., 1982].
Хотя CpG является наименее частым динуклеотидом, в гено-
мах встречаются участки с относительно более высоким (хотя
все равно ниже ожидаемого) содержанием CpG — CpG-островки
(CpG-islands) [Cooper et al.,# 1983; Bird, 1986; Cross and Bird,
1995; Gardiner, 1995]. В геномах млекопитающих такие островки
встречаются в начале генов и этот феномен используется, в част-
ности, при поиске новых генов. Таким образом, динуклеотиды
156 Глава 5. Отдельные задачи эволюционного анализа
CpG могут подвергаться влиянию различных эволюционных
факторов в разных участках генома.
Ряд наблюдений, однако, указывает на то, что кроме ме.
тилирования и деаминирования, существуют и другие факто-
ры, ведущие к низкой встречаемости CpG. Так, ДНК мито.
хондрий и хлоропластов не метилирована, однако и в ней
CpG-содержание ниже ожидаемого [Boudraa and Perrin, 1987;
Cordon et al., 1994; De Amicis and Marchetti, 2000]. Кроме
того, метилирование-деаминирование CpG должно приводить
к повышению содержания TpG, чего часто не наблюдается.
Кроме CpG, во многих геномах наблюдается пониженное (по
сравнению с ожидаемым расчетным) содержание динуклеотида,
ТрА. Этот феномен может быть связан с целым рядом фак-
торов: тем, что два из трех стоп-кодонов начинаются с ТА
(см. табл. 1.2), чувствительностью этого динуклеотида в РНК
(т. е. динуклеотида UpA) к нарезанию, нестабильностью струщ
туры спирали ДНК с большим содержанием ТрА и др. [Boudraa
and Perrin, 1987; De Amicis and Marchetti, 2000; Jabbari and
Bernardi, 2004].
В кодирующих последовательностях, действие эволюционных
факторов, связанных со смещениями нуклеотидного состава!
в общем случае гораздо сильнее проявляется на уровне синони-
мичных замен. Это понятно, поскольку несинонимичные замены
подвергаются влиянию дополнительных, как правило, гораздо
более сильных эволюционных факторов, действующих на уровне
белка.
5.2.2. Различия в использовании кодонов
Большинство аминокислот может кодироваться более чем одним
(до шести) кодонов, и, как уже отмечалось ранее (разд. 1.7),
выбор синонимичных кодонов в составе кодирующей нуклео!
тидной последовательности — использование кодонов — в общем
случае не является случайным [Grantham et al., 1980]. Говорят
также о предпочтениях кодонов (codon preference) или сме-
щениях в использовании кодонов (codon usage bias). Понятно^
что использование кодонов является эволюционным феноменом,
по определению проявляющимся исключительно на уровне си-
нонимичных замен.
При описании использования кодонов следует в первую оче-
редь учитывать два фактора.
5.2. Анализ нуклеотидного и аминокислотного состава 157
Во-первых, использование кодонов в последовательности мо-
jgeT в значительной степени определяться смещением в ее нук-
леотидном составе (независимо от причин этого смещения). Так,
при предпочтении в некой кодирующей последовательности, ска-
жем, нуклеотида А за счет Т, это предпочтение, в общем случае,
будет особенно выражено в синонимичных позициях кодонов
(для сохранения аминокислот). Соответственно предпочтение А
за счет Т приведет к доминированию в этой последовательности
тех кодонов, в синонимичных позициях которых находится
нуклеотид А. Скажем, аланин будет чаще кодироваться кодоном
GCA, чем кодоном GCT.
Во-вторых, предпочтение одного синонимичного кодона перед
другим может быть связано с различиями во внутриклеточном
содержании тРНК, специфичных для этих кодонов. Если, ска-
жем, внутриклеточное содержание тРНК, специфичной для ко-
дона GCA, выше, чем тРНК, специфичной для синонимично-
го кодона GCT, то трансляция мРНК с большим содержани-
ем GCA будет проходить эффективнее (быстрее), что может
иметь селективное преимущество. В таких случаях говорят
о связи использования кодонов с трансляционным смещением
(translation bias), оптимизации использования кодонов. Транс-
ляционное смещение в значительной степени проявляется у
быстрорастущих микроорганизмов, включая, например, кишеч-
ную полочку Escherichia coli [Ikemura, 1981; Ikemura and
Ozeki, 1983] и дрожжи Saccharomyces cerevisiae [Bennetzen and
Hall, 1982; Sharp and Cowe, 1991]. Так, в последовательностях
Двух проанализированных генов S. cerevisiae для кодирования
98% из 1004 аминокислот используются только 25 из 61 суще-
ствующего смыслового кодона, что коррелирует с повышенным
внутриклеточным содержанием соответствующих тРНК. Более
того, в генах с высоким уровнем экспрессии смещения в ис-
пользовании кодонов могут быть выше, чем у генов с низким
Уровнем экспрессии. Таким образом, уровень экспрессии генов
Может регулироваться и с помощью использования кодонов.
Отметим, что два вышеописанных эволюционных фактора,
влияющие на использование кодонов, не являются, в общем
случае, взаимоисключающими: при эволюции какой-либо после-
довательности, они могут проявляться и сочетанно [Sharp et al.,
1993; Stenico et al., 1994]. Кроме того, отметим, что у клеточ-
ных форм жизни может иметь место как адаптация использо-
вания кодонов к внутриклеточному содержанию специфичных
Для этих кодонов тРНК, так и обратный процесс: адаптация
158 Глава 5. Отдельные задачи эволюционного анализа
внутриклеточного содержания различных синонимичных tPjjj^
к использованию кодонов. Соответственно вопрос о первичности-
вторичности этих эволюционных процессов остается в знд.
чительной степени открытым. В этом плане особый интерес
представляют вирусы, использующие при своей репликации
существующий в клетке пул тРНК. Адаптация использования
кодонов к внутриклеточному пулу тРНК показана, в частности,
для фагов [Sharp et al., 1985]. Особенно интересны данные для
фага Т4, геном которого кодирует восемь собственных тРНК.
Для этого фага показаны различия в использовании кодонов
в ранних и поздних генах: использование кодонов в ранних ге-
нах адаптировано к внутриклеточному пулу тРНК, в поздних —
к тРНК, кодируемым геномом фага [Cowe and Sharp, 1991].
Для анализа использования кодонов предложен ряд мате-
матических подходов, включая изучение частоты оптимальных
кодонов (.frequency of optimal codons, Fop) [Ikemura, 1985],
относительного использования синонимичных кодонов (relative
.synonymous codon i/sage, RSCU) [Sharp et al., 1986], индекса
адаптации кодонов (codon adaptation zndex, CAI) [Sharp and Li,
1987] и др. [Cameron and Aguade, 1998].
В настоящее время одним из наиболее распространенных
подходов к анализу использования кодонов является под-
ход, основанный на понятии эффективного числа кодонов
(effective number of codons, ENC) [Wright, 1990], позволяющий
визуализировать отношения между нуклеотидным составом
(GC-содержанием в третьих позициях кодонов, ОСЗ-содержание)
и использованием кодонов.
Рассмотрим принцип этого подхода, опуская полные матема-
тические выкладки (хотя и достаточно простые). Пусть некая
аминокислота кодируется четырьмя кодонами. В конкретной
анализируемой последовательности каждый из этих кодонов бу-
дет встречаться с некой вероятностью: pi,... ,pi соответственно-
Если все эти вероятности равны, т. е. каждый из четырех воз-
можных кодонов использован одинаково часто, то значение ENC
для этой аминокислоты будет приближаться к четырем (по мер6
увеличения длины анализируемой последовательности). Если
использован только один из возможных кодонов, то значение
ENC для этой аминокислоты будет приближаться к единице-
При промежуточных значениях вероятностей, значение ENC
будет варьировать от 1 до 4. Сумма значений ENC для все*
аминокислот и будет составлять ENC для данной последователь'
ности. Отметим, что в отличие от филогенетического анализа»
5.2. Анализ нуклеотидного и аминокислотного состава 159
который в ряде случаев может быть достаточно эффективно
и достоверно проведен и при использовании относительно корот-
ких (100-200 нуклеотидов) генетических последовательностей,
достоверные результаты анализа использования кодонов могут
быть получены только при использовании достаточно длинных
(сотни-тысячи аминокислот) последовательностей.
Понятно, что в любой кодирующей последовательности чис-
ло использованных кодонов может, в общем случае, варьиро-
вать от 20 (каждая аминокислота кодируется только одним
кодоном) до 61 (использованы все смысловые кодоны). Одна-
ко в конкретной кодирующей последовательности максимально
возможное число использованных кодонов будет определяться
ОСЗ-содержанием. Так, максимальное число кодонов, 61, мо-
жет быть использовано только в случае отсутствия смещений
в нуклеотидном составе. Однако в случаях абсолютно низкого
(0%) и абсолютно высокого (100%) ОСЗ-содержания максимально
могут быть использованы не 61 кодон, а только 30 (заканчива-
ющиеся на А или Т) или 31 (заканчивающиеся на G или С)
соответственно. Понятно, что и для любого промежуточного
значения ОСЗ-содержания (10, 20% и т. д.) существует некое
ожидаемое значение ENC (рис. 5.4). Если значение ENC близко к
тому значению, которое определяется ОСЗ-содержанием, то нет
оснований для утверждений о наличии трансляционного смеще-
ния. Если значения ENC много меньше максимально возможного
для данного ОСЗв-содержания, то вероятно наличие трансляци-
онного смещения (математический аппарат для статистическо-
го выявления трансляционного смещения в настоящее время
не разработан). Отметим также, что вместо ОСЗ-содержания
можно использовать ОСЗз-содержание, т. е. исключить кодоны
метионина и триптофана, кодирующиеся только одним кодоном,
из анализа. Пример такого анализа приведен на рис. 5.4.
Отметим, что одинаковые для двух последовательностей зна-
чения ENC при одинаковом же ОСЗ-содержании, в общем случае
Не означают, что в этих двух последовательностях используются
°Дни и те же кодоны. Так, две расположенные рядом на рис. 5.4
Последовательности используют одно и то же эффективное число
Иодонов — 35, однако эти кодоны могут быть как одними и теми
35{е, так и различными.
Разработаны и методы преобразования данных по исполь-
а°ванию кодонов в деревья [Long and. Gillespie, 1991]. Термин
'Филогенетические» деревья в данном случае не следует исполь-
3°вать (или следует использовать с осторожностью): подобный
160 Глава 5. Отдельные задачи эволюционного анализа
Рис. 5.4. Анализ ENC в зависимости от ОСЗз-содержания. Кривая линия
показывает значение ENC, ожидаемое в случае, если использование ко-
донов определяется исключительно ОСЗз-содержанием. Знаки (о) отра-
жают ситуации, когда значения ENC близки к максимально возможным
для данного ОСЗз-содержания, т. е. нет оснований для утверждения о
наличии трансляционного смещения. Знаки (•) отражают ситуации, ко-
гда значения ENC много меньше максимально возможного для данного
ОСЗз-содержания, т. е. возможно наличие трансляционного смещения
анализ не направлен на установление филогенетических отно-
шений между последовательностями.
5.2.3. Анализ нуклеотидного состава и использования кодонов
в филогенетических исследованиях
Для демонстрации того, как нуклеотидный состав и использова-
ние кодонов могут быть учтены в филогенетических исследова-
ниях, рассмотрим эволюционные отношения между астровиру-
сами, представителями семейства Astrovlridae.
Анализ несинонимичных замен выявил порядок эволюцион-
ных событий в истории астровирусов (на рис. 5.5, а представлены
результаты анализа участка orf2 геномов астровирусов, резуль-
таты анализа остальных открытых рамок считывания геномов
астровирусов, orfla и orflb, были аналогичными) [Lukashov and
Goudsmit, 2002а]. Наиболее древним эволюционным событием
в этом семействе вирусов являлось разделение эволюционных
линий вирусов птиц и млекопитающих. В последующем, среД11
астровирусов млекопитающих отделилась линия вируса овей
OAstV, затем — линии вируса свиней PAstV и вируса когне1<
5.2. Анализ нуклеотидного и аминокислотного состава 161
FAstV, и, наконец, произошла диверсификация между эволюци-
онными линиями различных серотипов вируса человека HAstV.
Структура филогенетического дерева астровирусов, наблюда-
емая при анализе несинонимичных замен, терялась при анализе
синонимичных замен (рис. 5.5,6). В этом дереве все виды виру-
сов и серотипы HAstV, кроме HAstV-4 и 8, были равноудалены
друг от друга: их эволюционные линии ответвлялись практи-
чески в одной точке. Эта потеря структуры дерева при анали-
зе синонимичных замен может быть объяснена как следствие
эффекта насыщения синонимичных замен (разд. 3.2.6): действи-
тельно, синонимичные дистанции между анализируемыми по-
следовательностями составляли 0,7 и более, что препятствует
надежной реконструкции эволюционных отношений на основе
анализа синонимичных замен [Lukashov and Goudsmit, 2002а].
Последующий анализ нуклеотидного состава геномов аст-
ровирусов выявил значительные, одинаковые для всех трех
открытых рамках считывания, различия в нуклеотидном со-
ставе между астровирусами млекопитающих (в этой работе
дополнительно был проанализирован геном астровирусов норок,
MAstV) [пап Hemert et al., 2007]. При этом геномы эволюци-
онно удаленных друг от друга астровирусов человека и птиц
имели сходный нуклеотидный состав, а геномы относительно
близких к астровирусам человека вирусов овец и норок имели
резкие отличия в нуклеотидном составе. В частности, содержа-
ние нуклеотида А в третьей позиции кодонов открытых рамок
считывания геномов астровирусов человека и птиц составляло
порядка 25%, т. е. значения, ожидаемого при отсутствии предпо-
чтения нуклеотида А: так, для orf2 оно варьировало в пределах
от 24,1 до 27,6% (в среднем — 26,4%) (рис. 5.6). В то же время
в третьей позиции кодонов открытых рамок считывания гено-
мов астровирусов овец и норок наблюдалось резко сниженное
содержание нуклеотида А: так, для orf2 оно составляло 17,5
и 18,2%, соответственно. Для астровирусов кошек и свиней эти
значения составляли 21,0 и 21,8% соответственно. Аналогичное
снижение содержания нуклеотида А наблюдалось и в первой
позиции кодонов открытых рамок считывания астровирусов овец
и норок (и, в меньшей степени — астровирусов кошек и свиней),
По сравнению с астровирусами человека и птиц. Пониженное со-
держание нуклеотида А в третьей позиции кодонов астровирусов
°вец и норок сопровождалось повышенным ССЗв-содержанием,
Наблюдавшимся также и для астровирусов кошек и свиней
<см. рис. 5.6).
'! 223«
162 Глава 5. Отдельные задачи эволюционного анализа
HAstV 4 АВ025809
HAstV 4 АВ025807
HAstV 4 АВ025812
HAstV 4 ABO25808
a
87
100
100
& 100
100
94
4)
99
100
3
2
HAstV 4 AB025811
" HAstV 4 AB025806
HAstV 4 консенсус
HAstV 4 AB025805
HAstV 4 AB025804
J HAstV 4 AB025810
Г1- HAstV 4 AB025801
j— HAstV 4 Z33883
I HAstV 4 AB025803
HAstV 4 AB025802
___I— HAstV 8 Z66541
100 H HAstV 8 консенсус
1001 HAstV 8 AF260508
I HAstV 2 L06802
100 HAstV 2 консенсус
HAstV 2 LI3745
100j HAstV 3 консенсус
- 100 Г HAstV 3 AF141381
L HAstV 3 AF117209
_______[ HAstV 7 консенсус
*0°*HAstV 7 Y08632
p HAstV 1 L23513
1001 HAstV 1 консенсус
b HAstV 1 Z25771
100 HAstV 1 S68561
HAstV 5 консенсус
HAstV 5 AB037274
HAstV 5 AB037273
HAstV 5 U15136
5 HAstV 6 Z46658
HAstV 6 AB031031
HAstV 6 AB031030
HAstV 6 консенсус
HAstV 6 AB013618
-----1 FAstV AF056197 j
100
74
100
100
94
1001
100
100 Г
100
PZ.lV дво е -
OAstV Y159371
TAstV 2 AF20666j
100
100
100
TAstV 1 Y15936]
X ANV 2 AB0468641
1 ANVAB033998I
0.05
5.2. Анализ нуклеотидного и аминокислотного состава 163
б
100
100
HAstV 6 консенсус
HAstV 6 АВ013618
HAstV 6 АВ031031
HAstV 6 АВОЗЮЗО
HAstV 6 Z46658
I----HAstV 1 L23513
T00|| HAstV 1 консенсус
871__iHAstV 1 Z25771
1001 HAstV 1 S68561
[HAstV 2 консенсус
HAstV 2 LI 3745
- HAstV 2 L06802
___IPlb HAetV 7 консенсус
' HAstV 7 Y08632
100
J---HAstV
1___Г HAstV
1001- HAstV
3 AF117209
3 консенсус
3 AF141381
100
1001
-----HAstV 5 U15136
IHAstV 5 консенсус
HAstV 5 AB037274
— HAstV 5 AB037273
HAstV 8 консенсус
L HAstV 8 AF260508
----HAstV 8 Z66541
100
100 [HAstV 4 AB025803
1 HAstV 4 AB025802
___HAstV 4 Z33883
___Г HAstV 4 AB025810
1001-HAstV 4 AB025801
- HAstV 4 AB025804
—100 HAstV 4 AB025805
HAstV 4 консенсус
70 I HAstV 4 AB025811
1 HAstV 4 AB025806
HAstV 4 AB025809
81 HAstV 4 AB025807
77 HAstV 4 AB025812
HAstV 4 AB025808
—| r —gj
~l TAst V 2 AF206663]
OAstV Y15937
100
70
ANV 2 AB046864]
ANV AB033998|
TAstV 1 Y15936
0.1
Рис. 5.5. Филогенетические отношения между астровирусами, установленные
при анализе последовательностей orf2. (а) — анализ несинонимичных замен,
(б) — анализ синонимичных замен. HAstV-l-HAstV-8 — астровирусы человека
серотипов 1-8 (не выделены), FAstV — астровирус кошек, PAstV — астровирус
свиней, OAstV — астровирус овец (выделены различными оттенками серого
Ивета), TAstV-1 и 2— астровирусы индюков типов 1 и 2, ANV - вируса нефрита
птиц типов 1 и 2 (выделены рамками). Природным хозяином вируса нефрита
птиц являются куры. Номера индивидуальных последовательностей в GenBank
Указаны, для HAstV использованы также консенсусные последовательности
серотипов. Для (а), указан порядок событий в эволюционной истории астровиру-
сов: от разделения эволюционных линий астровирусов птиц и млекопитающих
(1) до начала диверсификации среди астровирусов человека (5). При анализе
Синонимичных замен структура филогенетического дерева теряется
164 Глава 5. Отдельные задачи эволюционного анализа
позиции кодонов:
первая
вторая
третья
OAstV
MAstV
PAstV
FAstV
HAstV-lL
HAstV-1Z
HAstV-2
HAstV-3
HAstV-4
HAstV-5
HAstV-6
HAstV-7
HAstV-8
TAstV-1
TAstV-2
ANV-1
ANV-2
> о
X<
О
о
Д о
(3
А
С i
L 1
ТС AG
X
X
X
X
G
•>
<
□
OO
> о
•>
Т АС
xi
Zx
>с
X
X 3
XI
X I 1
> о
60
□
<P
ДО
Д
О
о
о
<>
о
I
ti
: Ф
<>
О
д >
GCA Т
x
х
<
5
<
>
<
3
О
О
I Д
С к
( i
I >
□
X
X
□
[
> о
□
□
X
14 19 24 29 34 39
14 19 24 29 34 39
14 19 24 29 34 39
ДА ОТ XG ОС
Рис. 5.6. Нуклеотидный состав orf2 астровирусов. MAstv — астровирус
норок, HAstV-1L и Z—два варианта астровирусов человека сероти-
па 1, HAstV-1, обозначения остальных вирусов приведены в подпи-
си к рис. 5.5. Горизонтальная ось отображает процентное содержание
нуклеотидов, вертикальные линии показывают среднее содержание
каждого нуклеотида в геномах всех астровирусов. Отмечены неко-
торые из случаев смещений нуклеотидного состава: повышенное со-
держание G за счет А в геномах астровирусов овец, норок, свиней
и кошек в первой позиции кодонов, пониженное содержание А в
геномах этих же астровирусов (в особенности у вирусов овец и норок)
по сравнению с остальными астровирусами. По ван Хемерту с соавт.
[van Hemert et al., 2007]
Таким образом, близкородственные отношения между астро-
вирусами млекопитающих не сопровождаются сходством нук-
леотидного состава их геномов. Более того, астровирусы че-
ловека, эволюционно далекие от астровирусов птиц, близки
по нуклеотидному составу геномов к астровирусам птиц, по не к
астровирусам других млекопитающих. Отметим, что различия
в нуклеотидном составе между астровирусами человека и других
млекопитающих проявляются в первой и третьей позициях
5.2. Анализ нуклеотидного и аминокислотного состава 165
БСЗв-содержание
Рис. 5.7. Анализ эффективного числа кодонов (ENC) у астровирусов
и их хозяев. Три верхние панели показывают данные для трех от-
крытых рамок считывания геномов астровирусов. Близкие друг другу
по использованию кодонов астровирусы человека и птиц обозначены
светлыми символами: восемь серотипов астровирусов человека (Д),
Два типа вирусов индюков (о) и два типа вирусов нефрита птиц (о).
Остальные астровирусы обозначены темными символами: астровирусы
овец (•), норок (), кошек (А) и свиней (♦). Для ряда астровирусов
нуклеотидные последовательности orfl к настоящему времени не опре-
делены. Данные для хозяев астровирусов приведены на нижней панели,
хозяева обозначены в соответствии с инфицирующими их вирусами.
По ван Хемерту с соавт. [van Hemert et al., 2007]
166 Глава 5. Отдельные задачи эволюционного анализа
кодонов, в которых, в отличие от второй позиции, могут иметь
место синонимичные замены (разд. 1.6.2).
Аналогичные результаты были получены и для использо-
вания кодонов в геномах астровирусов. При анализе эффек-
тивного числа кодонов астровирусы человека группировались
вместе с астровирусами птиц и отдельно от астровирусов других
млекопитающих (рис. 5.7). При этом для всех астровирусов зна-
чения ENC были близки к максимуму, ожидаемому при данном
нуклеотидном составе, т. е. не наблюдалось (резкого) трансляци-
онного смещения. Аминокислотные дистанции между последо-
вательностями астровирусов человека и других млекопитающих
были значительно ниже (соответственно — их аминокислотный
состав был сходен), чем между астровирусами человека и птиц
(рис. 5.8, а) — однако при анализе отношений между астровиру-
сами на основании использования кодонов астровирусы человека
и птиц группировались вместе (рис. 5.8, б).
В результате невозможность установить близкородствен-
ные отношения между астровирусами млекопитающих (потеря
структуры дерева) при анализе синонимичных замен (рис. 5.5, б)
может быть (дополнительно к эффекту насыщения замен) свя-
зана и с различиями в нуклеотидном составе и использова-
нии кодонов. Резкие отличия нуклеотидного состава и исполь-
зования кодонов в геномах астровирусов овец, норок, свиней
и кошек от этих характеристик геномов астровирусов человека
и птиц приводят к тому, что при анализе синонимичных замен
вирусы человека находятся ближе к эволюционно удаленным
от них вирусам птиц, чем к вирусам других млекопитающих
(рис. 5.8, а и б).
Филогенетические отношения между астровирусами млеко-
питающих не соответствовали отношениям между их хозяе-
вами (рис. 5.8,в). В эволюции млекопитающих отделение ли-
нии человека явилось первым из рассматриваемых событий,
с последующим отделением линии кошки, а разделение меж-
ду линиями свиньи и овцы являлось наиболее поздним со-
бытием [Kumar and. Hedges, 1998]. В эволюции астровиру-
сов порядок событий был прямо противоположным (рис. 5.5,о
и 5.8, а): первым произошло отделение линии астровирусов овеН»
а наиболее поздним событием являлось разделение между ли-
ниями астровирусов кошек и человека. Такие неконгруэнтные
отношения между астровирусами и их хозяевами предпола-
гают по меньшей мере два межвидовых переноса в истории
семейства Astroviridae. Наиболее вероятным сценарием явлЯ'
5.2. Анализ нуклеотидного и аминокислотного состава 167
0.5
TAstV-2
TAstV-1
HAatV-2
HAstV-4
HAstV d
HAdtV 8
HAetVlL
tV-lZ________
0ЛИ '
птицы (Aues)
человек (Omnivora)
кошка
норка
овца
свинья
Carnivora
Herbifora
310 II 75
50 млн лет тому назад, приблизительно
курица
— индюк
*— ---человек
-------- ----------- норка
----- । кошка
0,0005 -------j_____________________। свинья
I------------ овца
Рис. 5.8. Филогенетические отношения между астровирусами, их хо-
зяевами, и корреляция использования кодонов астровирусами и их
хозяевами, (а) — филогенетические отношения между астровирусами
На основе анализа аминокислотных дистанций в orf2; (б) — отношения
Между астровирусами на основе анализа использования кодонов; (в) —
Филогенетические отношения между хозяевами астровирусов и вре-
мя их дивергенции; (г) — отношения между хозяевами астровирусов
На основе анализа использования кодонов. Эволюционно удаленные
ДРУг от друга астровирусы человека и птиц (а) близки при анализе
Использования кодонов (б), группы выделены. По ван Хемерту с со-
авт. [van Hemert et al., 2007]
168 Глава 5. Отдельные задачи эволюционного анализа
ется передача PAstV-подобного вируса кошкам и затем—чед0.
веку.
Анализ нуклеотидного состава и использования кодонов
(рис. 5.7 и 5.7, г) в геномах хозяев астровирусов показал сходство
этих характеристик у человека и птиц. Таким образом, сходный
нуклеотидный состав и использование кодонов в геномах аст-
ровирусов человека и птиц может быть результатом адаптации
этих астровирусов к их хозяевам.
5.3. Анализ молекулярных часов
5.3.1. Вопросы, связанные с концепцией молекулярных часов
На протяжении этой книги мы неоднократно отмечали важ-
ность концепции молекулярных часов (разд. 1.13) при изучении
различных вопросов молекулярной эволюции. Действительно,
если скорость эволюции каких-либо макромолекул постоянна
во времени и одинакова во всех нисходящих эволюционных
линиях, то эволюционные события в изучаемой группе форм
жизни могут быть достаточно легко установлены и датированы.
Реконструкция филогенетических отношений между формами
жизни является гораздо более простой задачей в случае суще-
ствования молекулярных часов в их эволюции.
До настоящего времени, однако, концепция молекулярных
часов продолжает оставаться предметом дебатов.
Основанные на экспериментальных данных мнения касатель-
но существования молекулярных часов варьируют от полного от-
рицания даже приблизительных молекулярных часов [Goodman,
1981а; Goodman, 1981b; Czelusniak et al., 1982] до пред-
положения о существовании универсальных для всех форм
жизни молекулярных часов в эволюции синонимичных пози-
ций [Ochman and Wilson, 1987]. Большинством исследователей,
тем не менее, сама возможность существования молекуляр-
ных часов принципиальным сомнениям не подвергается, как
не разделяется и идея о существовании универсальных часов.
Так, показана более высокая скорость эволюции у эукариот,
по сравнению с прокариотами [Hedges et al., 2001]. Дискус-i
сии идут о том, применимы ли принципы молекулярных ча-
сов в том или ином конкретном случае, и, если да — то на-
сколько.
Рассмотрим вопросы, связанные с анализом молекулярных
часов в эволюции форм жизни. К числу таких вопросов от-
5.3. Анализ молекулярных часов 169
лосятся проверка наличия молекулярных часов в эволюции
анализируемой группы, калибровка этих часов и использование
этих данных для датирования эволюционных событий, а также
вопросы, связанные с проблемами реконструкции филогенетиче-
ских отношений при отсутствии молекулярных часов в эволюции
анализируемой группы.
5.3.2. Установление и калибровка молекулярных часов —
тест относительных скоростей эволюции
Рассмотрим эволюцию двух последовательностей А и В, проис-
ходящих от общего предка О (рис. 5.9). Если скорость эволюции
линий А и В одинакова, то эволюционная дистанция последова-
тельностей А и В от общего предка также будет одинаковой (не
будет статистически достоверного различия между ними). Таким
образом, если общий предок А и В известен, то статистические
методы позволяют установить, одинакова ли скорость эволюции
линий А и В.
Однако в общем случае последовательность общего пред-
ка О — неизвестна. В таких случаях для установления нали-
чия молекулярных часов в эволюции линий А и В можно
использовать тест относительных скоростей эволюции, RRT
(relative-rate test), основанный на использовании внешней груп-
пы (разд. 4.1) [Sarich and Wilson, 1973; Wu and Li, 1985; Li and
Tanimura, 1987; Li and Bousquet, 1992].
При использовании такой внешней группы последовательно-
сти С в анализе (рис. 5.9) эволюционные дистанции Dij меж-
ду последовательностями могут быть выражены следующим
А ВС
рис. 5.9. Тест относительных скоростей эволюции для последователь-
ностей А и В, имевших общего предка О, с использованием последова-
тельности С в качестве внешней группы
170 Глава 5. Отдельные задачи эволюционного анализа
образом:
Dac = Dq а + Doc,
Dec = Dob + Doc-
D$b = Do a + Dob-
Поскольку эволюционные дистанции между последовательно-
стями А, В и С могут быть определены с использованием той
или иной эволюционной модели (гл. 3), можно найти дистанции
между последовательностями А и В и их (неизвестным) общим
предком О:
„ Dac + Dab — Dbc
d°a =---------2-------
И
„ Dbc + Dab ~ Due
d°b =---------2-------•
Можно найти и эволюционную дистанцию между последова-
тельностью С и общим предком последовательностей А и В:
n Dac + Dbc — Dab
Doc =---------2-------•
Отметим, что скорость эволюции внешней группы, линии С,
в данном случае не важна: тест направлен на выяснение во-
проса, одинакова ли скорость эволюции линий А и В, скорость
эволюции линии С может и отличаться.
Обозначим разность между Dac и Dbc за d. В таком случае
установление одинаковости или неодинаковости скоростей эво-
люции в линиях А и В сводится к простому статистическому
вопросу: отличается ли достоверно d от 0 или нет. Так, если
абсолютное значение d превышает две стандартные ошибки,
то оно может считаться достоверным на уровне 95%. Диспер-
сия d рассчитывается согласно формуле
1(d) = V(Dac) + V(DBC) - 2V(Doc),
а формулы для расчета дисперсии при установлении эволюцион-
ных дистанций между двумя последовательностями приведены
при описании различных эволюционных моделей в гл.З.
Рассуждения и математические выкладки при анализе более
чем двух последовательностей аналогичны [Li and Bousquet,
1992].
До сих пор мы исходили из того, что с момента дивергенции
двух анализируемых последовательностей от их общего предка
5.3. Анализ молекулярных часов 171
прошло одинаковое время, т. е. эти линии разделились, скажем,
миллион лет тому назад, а представляющие эти линии после-
довательности были получены сегодня. Понятно, что под «се-
годня» понимается незначительный в эволюционном масштабе
промежуток времени. Скажем, при анализе молекулярных часов
в эволюции группы высших приматов, диверсификации между
эволюционными линиями в которой происходили миллионы лет
тому назад, возможный временной интервал между определе-
нием нуклеотидных последовательностей различных высших
приматов в несколько десятков лет (скажем, последователь-
ность некоего гена была определена для шимпанзе в 1980 г.,
а для орангутана — в 2000 г.) является несущественным. В таком
случае от эволюционных дистанций между двумя последова-
тельностями можно прямо перейти к скорости эволюции этих
линий, деля дистанции на одно и то же время независимой
эволюции этих линий. Однако для недавно (скажем, десятилетия
назад) разделившихся линий с высокой скоростью эволюции
разница в несколько лет между определением (временем су-
ществования) двух последовательностей является весьма суще-
ственной. Такие случаи возникают при изучении вирусов с вы-
сокой скоростью эволюции — например, ВИЧ-1 —и обсуждены
в разд. 5.3.7.
Таким образом, даже при отсутствии данных о последо-
вательности общего предка анализируемой группы тест от-
носительных скоростей эволюции позволяет установить, соот-
ветствует ли эволюция анализируемого генетического участка
этой группы модели молекулярных часов. Отметим, что сам
по себе тест относительных скоростей эволюции не измеряет
скорость эволюции анализируемых линий, число эволюционных
изменений за единицу времени: становится известно только
то, что скорость эволюции анализируемых линий — одинако-
ва, или же — нет. Определение собственно скорости эволюции
называют калибровкой молекулярных часов. Такая калибровка
может быть проведена, например, с использованием палеонто-
логических данных о времени существования общего предка
анализируемых линий. Примеры определения наличия и калиб-
ровки молекулярных часов рассмотрены в разд. 5.3.5-5.3.7.
Кроме того, отметим, что современные математические ме-
тоды (используемые, в частности, в описываемой в разд. 6.5
Программе PAML) позволяют и рассчитать генетическую после-
довательность наиболее вероятного последнего общего предка
анализируемых последовательностей.
172 Глава 5. Отдельные задачи эволюционного анализа
5.3.3. Другие подходы к установлению молекулярных часов
Понятно, что любой подход к решению вопроса о том, соответ-
ствует ли эволюция двух или более последовательностей модели
молекулярных часов, в своей основе сводится к установлению
того, существуют ли статистически достоверные различия в ско-
ростях эволюции этих последовательностей. Если статистически
достоверных различий нет, то модель молекулярных часов мо-
жет быть принята.
При простейшем подходе с использованием описанного
в предыдущем разделе теста относительных скоростей эволюции
вопрос установления молекулярных часов сводится к задаче
о том, имеются ли статистически достоверные различия между
длинами ветвей анализируемых последовательностей. Однако
разработан и ряд других, более сложных, статистических под-
ходов.
Одним из наиболее распространенных из них является под-
ход, основанный на тесте отношения правдоподобий (likelihood
zatio Zest, LRT) [Felsenstein, 1988]. Принцип этого подхода
заключается в том, что метод максимального правдоподобия
(разд. 4.3.3) позволяет устанавливать длины ветвей филогенети-
ческого дерева, предполагая как соответствие, так и несоответ-
ствие эволюции анализируемой группы последовательностей мо-
дели молекулярных часов. При несоответствии эволюции модели
молекулярных часов каждая эволюционная линия будет иметь
собственную скорость эволюции (модель свободных скоростей,
free-rates model), и длины всех 2п — 3 ветвей дерева без корня
должны быть установлены. При соответствии же эволюции
модели молекулярных часов требуется установить длины только
п — 1 ветвей дерева с корнем, поскольку в таком случае пред-
полагается, что длины ветвей любых двух последовательностей,
имеющих общего предка, будут одинаковыми. Таким образом,
можно определить правдоподобие обоих случаев, предполагая
(вынуждая) и не предполагая, что эволюция анализируемой
группы последовательностей соответствует модели молекуляр-
ных часов, а затем статистически сравнить результаты обоих
предположений.
Предложены и другие подходы к установлению молекуляр'
ных часов, в том числе — основанные на анализе распределения
замен и сравнении правдоподобий предположений о соблюдения
и несоблюдении молекулярных часов [Langley and Fitch, 19741
Fitch and Langley, 1976; Gillespie and Langley, 1979; Wu and 1^’
5.3. Анализ молекулярных часов 173
1985; Gillespie, 1986а; Gillespie, 1986b; Gillespie, 1986с; Muse and
\\feir, 1992].
5.3.4. Проблемы филогенетического анализа, связанные
с несоблюдением модели молекулярных часов
Хотя реконструкция филогенетических отношений упрощается
в случае существования молекулярных часов в эволюции ана-
лизируемой группы последовательностей, современные филоге-
нетические методы (в отличие от метода UPGMA) не требуют
соблюдения модели молекулярных часов для реконструкции
эволюционных отношений (гл. 4). Это, тем не менее, не означает,
что несоблюдение молекулярных часов не приводит к проблемам
при реконструкции филогенетических отношений.
Так, одним из желательных свойств любого статистического
метода является его состоятельность (consistency) — приближе-
ние результатов расчетов к истинным по мере повторения экспе-
риментов. Скажем, вряд ли стоит ожидать, что после двенадцати
бросков шестигранной игральной кости каждый из возможных
исходов будет наблюдаться именно два раза — однако по мере по-
вторения бросков (до бесконечности) наблюдаемая вероятность
каждого из шести возможных исходов будет стремиться к ожи-
даемой вероятности, к 1/6. В филогенетическом анализе под
повтором бросков (т. е. увеличением размера экспериментальной
выборки) следует понимать увеличение длины анализируемого
генетического участка. Чем длиннее последовательность, чем
больше позиций проанализировано, тем более надежными (близ-
кими к истинным) должны быть результаты реконструкции
эволюционных отношений.
Показано, что при несоблюдении модели молекулярных ча-
сов ряд филогенетических методов, метод максимальной эко-
номии — в особенности, могут становиться несостоятельны-
ми [Cavender, 1978; Felsenstein, 1978; Schulmeister, 2004]. Си-
туация, в которой возникает несостоятельность, называется
зоной Фельзенштейна (Felsenstein zone) и связана с феноме-
ном, известным как притяжение длинных ветвей (long-branch
attraction) [Felsenstein, 2004; Bergsten, 2005]. Суть этого фено-
мена состоит в том, что в независимых эволюционных лини-
ях с высокой скоростью эволюции могут вероятно произойти
одинаковые мутации, которые будут (неправильно) расценены
Яак свидетельствующие о близком эволюционном родстве этих
янний, в действительности далеких друг от друга.
174 Глава 5. Отдельные задачи эволюционного анализа
5.3.5. Анализ молекулярных часов
в эволюции высших организмов
На протяжении десятилетий концепция молекулярных часов
интенсивно тестировалась прежде всего на основании анализа
прямых (определение последовательностей) и непрямых (в част-
ности, результатов ДНК-ДНК гибридизации и серореактивно-
сти) генетических данных высших организмов. Выбор этих
объектов исследований в значительной степени определялся
тем, что для ряда высших организмов имеются относительно
полные палеонтологические данные, позволяющие независимо
определять порядок эволюционных событий и датировать их
с известной степенью точности.
Так, анализ генетических последовательностей мышей, крыс
и хомяков с использованием последовательностей человека в ка-
честве внешней группы показал соответствие эволюции этой
группы грызунов модели молекулярных часов (по крайней мере
для синонимичных замен) [O’hUigin and Li, 1992]. Синонимич-
ные дистанции между последовательностями наиболее родствен-
ных между собой грызунов, мышей и крыс, составляли 18%.
Синонимичные дистанции между ними и последовательностя-
ми менее родственного им хомяка были одинаковы (не было
статистически достоверных отличий): 30 и 31% для последова-
тельностей мышей и крыс соответственно. Далее, синонимичные
дистанции между последовательностями всех трех грызунов
и последовательностями человека также были одинаковы: 53, 52
и 52% для последовательностей мышей, крыс и хомяков соот-
ветственно. Поскольку установлено соответствие эволюции этих
грызунов модели молекулярных часов, то можно определить, что
дивергенция между эволюционной линией хомяка и кластером
мышей и крыс произошла в 31/18 = 1,7 раз ранее дивергенции
между линиями мышей и крыс.
Другие исследования показали более высокую скорость эво-
люции грызунов по сравнению с приматами [Wu and Li, 1985;
Gu and Li, 1992], а также замедление скорости эволюции
в линиях человека и человекообразных обезьян, по сравнению
с более низкими обезьянами (мартышковыми и обезьянами
Нового света), приблизительно в 1,3 раза [/foop et al., 1986;
Li and Tanimura, 1987; Li et al., 1987; Seino et al., 1992;
Ellsworth et al., 1993]. Однако практически каждая публикация
касательно молекулярных часов вызывает дискуссии и последУ'
ющие публикации, оспаривающие и использованную методоло-
5.3. Анализ молекулярных часов 175
гию, и выбор той или иной внешней группы. В качестве примера
см. дискуссию [Hedges and Kumar, 2003; Hedges and Kumar,
2004; Graur and Martin, 2004; Glazko et al., 2005].
Особым направлением дискуссии является фундаментальный
вопрос о том, насколько вообще в качестве меры эволюционного
времени следует использовать физические единицы времени (го-
ды, миллионы лет). Для эволюционного процесса важно не фи-
зическое время — прошел миллион лет, — а число сменившихся
за это время поколений (разд. 1.12). Пусть вероятность мута-
ции и ее последующего закрепления в популяции одинакова,
скажем, у человека и мыши и составляет 0,1. В таком случае
следует по определению говорить о наличии молекулярных
часов в эволюции человека и мышей. Однако среднее время
воспроизводства потомства (репликации) у человека составляет
порядка двадцати лет (в среднем за всю историю существова-
ния человека, в настоящее время — больше), а среднее время
воспроизводства потомства у мыши — порядка шести месяцев.
Естественно, что, скажем, за десять лет в человеческой попу-
ляции накопится 0,05 мутаций, а в популяции мышей — 2,0.
Таким образом, результаты анализа генетической информации
без учета времени воспроизводства потомства приведут к вы-
воду о более высокой скорости эволюции мышей, а результаты
анализа с учетом времени воспроизводства потомства приведут
к выводу об одинаковой скорости эволюции мышей и человека.
Рассуждая еще строже, следует учитывать не только время
воспроизводства потомства, но и число репликаций (предков)
половых клеток за это время [Laird et al., 1969; Kohne, 1970].
Мутация может (равновероятно) произойти при каждом делении
(предков) половых клеток. Любая половая клетка половозре-
лого организма является результатом некоего числа делений
зиготы. При этом для образования сперматозоида и яйцеклетки
требуется разное число делений. Установлено, что у мышей
Для образования зрелой яйцеклетки требуется в среднем 27,
а зрелого сперматозоида — 29 делений зиготы [Ы, 1997]. У че-
ловека для образования зрелой яйцеклетки требуется в среднем
33, а зрелого сперматозоида — 40 (эти данные не являются на-
дежными) делений зиготы. При каждом из этих делений могут
произойти ошибки считывания, возникнуть мутации, которые
будут переданы потомкам.
Учитывая вышеизложенные рассуждения, интерпретацию
Данных о меньшей скорости эволюции человека по сравнению
с эволюцией более низких приматов или грызунов можно и из-
176 Глава 5. Отдельные задачи эволюционного анализа
менить на вывод о более высокой скорости эволюции чед0
века.
Отметим также, что генетические рекомбинации (разд.5д)
могут приводить к (неправильному) выводу о несоблюдении мо-
дели молекулярных часов, соответственно рекомбинации долж-
ны быть выявлены до анализа молекулярных часов.
5.3.6. Анализ молекулярных часов в эволюции вирусов
Для вирусов среднее время воспроизводства потомства гораздо
меньше, чем для высших организмов, и измеряется не годами
и десятилетиями, но днями. Так, для ВИЧ-1 это время состав-
ляет порядка двух дней [Но et al., 1995; Wei et al., 1995].
Кроме того, для вирусов вообще, РНК-содержащих вирусов —
в особенности, характерна гораздо более высокая вероятность
мутаций, ошибок при репликации геномов. Так, вероятность
нуклеотидных замен в расчете на один цикл репликации ВИЧ-1
составляет порядка К)”1 [Preston et al., 1988; Bebenek et al., 1989;
Roberts et al., 1989]. Для человека, эта вероятность составляет
порядка IO-8 [Drake et al., 1998].
Быстрая смена поколений и гораздо более высокая частота
мутаций делают вирусы привлекательным объектом эволюцион-
ных исследований: для многих вирусов эволюционные процессы
можно наблюдать практически в режиме реального времени.
Кроме того, геномы вирусов имеют небольшой размер (часто по-
рядка 5000-10000 нуклеотидов), что дает возможность изучать
эволюцию полноразмерного генома.
Концепция молекулярных часов использовалась для опреде-
ления дат диверсификации как ДНК-содержащих вирусов — се-
мейства гепаднавирусов [Orito et al., 1989], вирусов герпеса мле-
копитающих [McGeoch et al., 1995] и др., так и РНК-содержащих
вирусов — коронавирусов [Sanchez et al., 1992; Pyre et al.,
2006], флавивирусов [de A.Zanotto et al., 1996], пикорнави-
русов [Villaverde et al., 1991] и ретровирусов [Querat et al-,
1990], включая ВИЧ-1 и вирус иммунодефицита обезьян (.simian
immunodeficiency rirus, SIV) [Li et al., 1988; Sharp and Li, 1988;
Yokoyama et al., 1988; Eigen and Nieselt-Struwe, 1990; Gojobori
and Moriyama, 1990; Gojobori et al., 1990]. В этих исследова-
ниях, однако, существование молекулярных часов в эволюции
анализируемых групп вирусов подразумевалось, но не проверя-
лось, т. е. молекулярные часы калибровались без доказательства
того, что они существуют.
5.3. Анализ молекулярных часов 177
Учитывая высокую скорость эволюции вирусов, вирусные
последовательности, полученные годы-десятилетия тому назад,
являются по сути палеонтологическими данными, что дает
возможность прямого анализа молекулярных часов, сравнения
последовательностей вирусов, существующих сегодня и ранее.
Такие исследования были проведены для вируса гриппа А
с использованием генетических данных, полученных от ва-
риантов вируса, циркулировавших в человеческой популяции
на протяжении десятилетий (с 1918 года, эпидемия испан-
ки) [Buonagurio et al., 1986; Fitch et al., 1991; Taubenberger et al.,
2005; Tumpey et al., 2005]. Было показано, что эволюция виру-
са гриппа А соответствует модели молекулярных часов. Одна-
ко аналогичные исследования вируса везикулярного стоматита
показали несоответствие его эволюции модели молекулярных
часов [Gillespie, 1993; Nichol et al., 1993]. При этом филогене-
тические деревья как вирусов гриппа, так и вирусов везику-
лярного стоматита имеют кактусообразную топологию (разд. 4.1),
отличающуюся от звездообразной топологии филогенетического
дерева ВИЧ-1 (рис. 5.10). Отмечено, что звездообразная фило-
гения, при которой все анализируемые эволюционные линии
отпзчковались практически одномоментно, является идеальной
ситуацией для анализа молекулярных часов [Kimura, 1983].
5.3.7. Молекулярные часы в эволюции ВИЧ-1
и их использование для датирования
начала эпидемий и эволюционных событий
Среди вариантов ВИЧ-1 выделяют три группы вирусов, три
филогенетических кластера: наиболее многочисленную (> 99%
всех инфекций ВИЧ-1 в мире) главную группу М (z/iain), а также
группы О (outlier, обособленная группа) и N (поп-М-поп-О, груп-
па не-М-не-О) (см. рис. 5.10). В группе М выделяют филогене-
тические кластеры — генетические субтипы ВИЧ-1 A-К (кроме
субтипа Е, для которого полноразмерный геном пока не обна-
ружен). Географически наиболее распространенным субтипом
ВИЧ-1 является субтип В [Myers et al., 1995]. Варианты этого
субтипа вызвали эпидемии во многих развитых странах мира,
включая страны Северной Америки и Западной Европы, что
связано с эффектом основателя (разд. 1.12): попавший в некую
человеческую популяцию, определенную эпидемиологическую
сеть, специфический вариант вируса быстро распространяется
12— 2238
178 Глава 5. Отдельные задачи эволюционного анализа
Рис. 5.10. Эволюционные отношения между вариантами ВИЧ-1. Группа М
выделена, группы О и N обозначены соответствующими буквенными индексами,
как и вирусы иммунодефицита шимпанзе (индекс CPZ). Субтипы и суб-субтипы
группы М обозначены соответствующими индексами. Неклассифицируемые ва-
рианты ВИЧ-1 обозначены индексом U. Рекомбинантные формы ВИЧ-1 в ана-
лиз не включены. Эволюционные ветви вирусов иммунодефицита шимпанзе
показаны пунктирными линиями. Общий предок группы М показан стрелкой.
Для одного из субтипов группы М, субтипа В, выделены общий предок этого
субтипа (•) и эволюционные линии индивидуальных вариантов ВИЧ-1 этого
субтипа. Общий узел субтипов В и D отмечен знаком (о)
5.3. Анализ молекулярных часов 179
в ней. Более подробно вопросы генетической классификации
и молекулярной эпидемиологии ВИЧ-1 рассмотрены в разд. 5.5.3.
Филогенетическое дерево группы М ВИЧ-1 имеет единый
филогенетический узел, достаточно продолжительные общие
для каждого субтипа внутренние ветви, и, наконец, индивиду-
альные ветви отдельных вариантов вирусов (см. рис. 5.10). Таким
образом, это дерево имеет двойную звездообразную топологию:
для всех субтипов вместе и для каждого субтипа в отдельности.
Соответственно основными эволюционными событиями в группе
М ВИЧ-1 является ее образование, разделение между линиями
субтипов и диверсификации внутри каждого субтипа.
Анализируя многолетние генетические данные о вариантах
ВИЧ-1, циркулирующих в той или иной человеческой популя-
ции, можно проверить, возрастает ли генетическая гетероген-
ность этих вариантов линейно, т. е. установить наличие молеку-
лярных часов в эволюции ВИЧ-1, и откалибровать их.
Рассмотрим модель эволюции ВИЧ-1 в ходе некой эпиде-
мии [Lukashov and Goudsmit, 1998; Lukashov and Goudsmit,
2002b]. Концепция молекулярных часов постулирует постепенное
накопление нуклеотидных замен в индивидуальных геномах
ВИЧ-1 и линейное возрастание (средних) эволюционных дистан-
ций между индивидуальными последовательностями и их общим
предком в ходе эпидемии. Таким образом, если молекулярные
часы действуют в эволюции ВИЧ-1, то эволюционные дистанции
между вариантами ВИЧ-1 и их общим предком в ходе эпидемии
могут быть описаны формулой
DT = г(Т - То),
где Dr — средняя эволюционная дистанция вирусной популяции
от ее последнего общего предка (филогенетического узла), г —
средняя скорость эволюции, То — дата начала диверсификации
в вирусной популяции и Т — дата получения последовательно-
стей.
Анализ эволюционных дистанций между вирусами в какой-
либо популяции и их общим предком в зависимости от времени
дает возможность проверить, соответствует ли эволюция данной
вирусной популяции этой формуле, и, если это так, то установить
дату начала диверсификации этой вирусной популяции, дату
То: Т = То при DT = 0. Если эпидемия ВИЧ-1 в какой-либо
человеческой популяции началась после занесения и распростра-
нения одного варианта ВИЧ-1 (т. е. начальная гетерогенность
этой вирусной популяции, Dt, равна 0), то дата начала эпидемии
180 Глава 5. Отдельные задачи эволюционного анализа
может быть установлена. Если условие начальной гомогенности
вирусной популяции выполняется, то дата начала эпидемии
соответствует времени существования последнего общего предка
данной вирусной популяции. Если это условие не выполняется
и данная эпидемия является результатом нескольких независи-
мых заносов вирусов, то дата их последнего общего предка будет
предшествовать дате начала эпидемии. Таким образом, в общем
случае установленная предлагаемым методом дата должна учи-
тываться как наиболее ранняя возможная дата начала эпидемии.
Даты начала многих вспышек эпидемии ВИЧ-1 установлены
в результате ретроспективных серологических и/или эпиде-
миологических исследований. Следовательно, существует воз-
можность сравнить результаты этих исследований с данными
молекулярного анализа и определить достоверность различных
эволюционных моделей и филогенетических методов в описании
эволюции ВИЧ-1.
Анализ генетических последовательностей вариантов ВИЧ-1,
циркулирующих в ряде эпидемий, с использованием вышеопи-
санной модели установил наличие молекулярных часов в эво-
люции ВИЧ-1, по крайней мере в пределах эволюции субти-
па [Lukashov and Goudsmit, 1998; Lukashov and Goudsmit,
2002b]. Во всех изученных человеческих популяциях (эпиде-
миологических сетях, разд. 5.5.1 и 5.5.3), включая эпидемии
ВИЧ-1 субтипа В в США и среди мужчин с гомо- и бисексуаль-
ной ориентацией и внутривенных наркоманов в Нидерландах,
дистанции между индивидуальными вариантами ВИЧ-1 и их
общим предком (в качестве которого был использован консенсус
конкретной эпидемии) линейно возрастали за время наблюде-
ний [Lukashov and Goudsmit, 2002b]. Аналогичные результаты
были получены и для других субтипов ВИЧ-1 — в частности,
для субтипа С [Abebe et al., 2001а; АЪеЪе et al., 2001b]. Данные
для эпидемии ВИЧ-1 в США представлены на рис. 5.11.
Таким образом, наличие молекулярных часов в эволюции
ВИЧ-1 позволяет датировать начало эпидемий. Отметим также,
что, хотя анализ нуклеотидных дистанций между индивиду*
альными последовательностями гена env, для которого харак-
терна высокая скорость эволюции, вариантов ВИЧ-1 и их об-
щим предком и выявляет достоверное возрастание дистанции
во времени, использование нуклеотидных дистанций приводит
к датированию начала эпидемии ВИЧ-1 в США временным
интервалом между 1953-1967 гг., что не согласуется с датой
(значительно предшествует ей), установленной в ретроспектив-
5.3. Анализ молекулярных часов 181
ных (серо)эпидемиологических исследованиях (см. рис. 5.11).
Раздельный анализ синонимичных и несинонимичных дистан-
ций показывает, что причиной этого несоответствия является
эволюция несинонимичных позиций: в гене с высокой скоростью
эволюции, все возможные (разрешенные, с учетом необходимо-
сти сохранения функциональности гена) несинонимичные заме-
ны накапливаются быстро, после чего несинонимичные дистан-
ции не возрастают или, по крайней мере, возрастают гораздо
медленнее. Это явление может быть определено как эффект
функционального насыщения в отличие от обычного насыщения
(разд. 3.2.6) [Lukashov and Goudsmit, 1997а].
Датирование начала эпидемии ВИЧ-1 в США при анализе
синонимичных дистанций позволяет установить 1974-1976 гг.
как дату начала эпидемии (95% доверительные интервалы
для различных филогенетических методов находятся в пределах
1969-1977 гг., рис. 5.11), что согласуется с ретроспективными
(серо)эпидемиологическими данными. Таким образом, для гене-
тического участка ВИЧ-1 с высокой скоростью эволюции надеж-
ные результаты дает анализ синонимичных замен [Sale mi et al.,
2001; Lukashov and Goudsmit, 2002b].
Анализ глобальных вариантов ВИЧ-1 субтипа В позволил
отнести начало диверсификации этого субтипа (т. е. общий фи-
логенетический узел, дату существования последнего общего
предка субтипа В, дату начала эпидемии субтипа В) к концу
1960-х — началу 1970-х гг. (рис. 5.12). Кроме последовательно-
стей участка гена env, в этом анализе были использованы и по-
следовательности участка гена pol, скорость эволюции которого
гораздо меньше. Поскольку функциональное насыщение несино-
нимичных замен в гене pol не достигнуто, анализ нуклеотидных
замен в гене pol приводит к тем же результатам, что и анализ
синонимичных замен в гене env.
Наиболее ранним известным вариантом ВИЧ-1 является
вариант, полученный из образца крови пациента из Заира
в 1959 г. [Zhu et al., 1998]. Филогенетический анализ генетиче-
ских последовательностей этого варианта показал его близость
к общему узлу субтипов В и D (см. рис. 5.10), что было ис-
пользовано для датирования разделения между эволюционными
линиями субтипов В и D. Однако отметим, что в любой ис-
следованной эпидемии в любой момент времени, даже и годы
спустя после начала эпидемии, присутствуют варианты ВИЧ-1,
близкие к общему предку эпидемии (рис. 5.11 и 5.12) [Lukashov
and Goudsmit, 1997а; Lukashov and Goudsmit, 2002b]. Это яв-
182 Глава 5. Отдельные задачи эволюционного анализа
год год
6 е
D015l . D 0.21
0.12
0.09
0.06
0.03
0.00 -----1---1----1----1---1-----1 0.00 ----1---1---1---1---1---1---1
1972 1976 1980 1984 1988 1992 1996 1968 1972 1976 1980 1984198819921996
год год
Ж
D 0.30-
0.27-
0.24-
0.21-
0.18-
0.15-
0.12
0.09-
0.06-
0.03-
0.00-
1968 1972 1976 1980 1984 1988 1992 1996
год
5.3. Анализ молекулярных часов 183
дяется отражением вариаций в скорости эволюции различных
индивидуальных линий ВИЧ-1. Таким образом, существование
варианта ВИЧ-1, близкого к общему предку (филогенетическо-
му узлу), само по себе не является эволюционным событием,
т.е. не характерно только для определенного периода в эволюции
ВИЧ-1. Соответственно ошибочным будет прямое датирование
какого-либо эволюционного события на основании того, что
выделенный в этот год вариант ВИЧ-1 был близок к этому
филогенетическому узлу.
Описанная выше модель для анализа молекулярных часов
в недавней эволюции ВИЧ-1 была использована и для дати-
рования предыдущих событий в истории группы М, вклю-
чая ее появление и разделение между линиями субтипов
ВИЧ-1 [Korber et al., 2000]. Однако существенной проблемой
здесь может являться принципиальная разница закономерно-
стей (и скоростей) эволюции ВИЧ-1 до и после образования
современных субтипов. Нет оснований считать, что скорость
эволюции ВИЧ-1 (длина ветвей за год) была хотя бы приблизи-
Рис. 5.11. Эволюционные дистанции последовательностей участка гена
env вариантов ВИЧ-1 субтипа В из США от их общего предка в ходе
эпидемии. Проанализированы последовательности, полученные между
1982 и 1994 гг. Каждая точка показывает дистанцию индивидуальной
последовательности, полученной в определенный год, или среднюю
дистанцию всех последовательностей в определенный год (5) от общего
предка всех последовательностей. Использованы следующие методы
установления длин ветвей последовательностей от общего предка: ме-
тод NJ (а) и метод ML (б) для нуклеотидных дистанций, метод NJ
для несинонимичных дистанций (в), метод NJ для индивидуальных
(г) и средних за год (5) синонимичных дистанций, метод ML для
несинонимичных (е) и синонимичных (ж) дистанций. Показаны 95%
доверительные интервалы, рассчитанные соответствующими методами
с использованием индивидуальных точек. Статистика: а — г — 0,27,
г2 = 0,07, наклон 0,0021, пересечение с осью X —1953, р = 0,02;
б— г = 0,41, г2 = 0,17, наклон 0,0037, пересечение с осью X —1967,
р < 0.001; в — г = 0.14, г2 = 0.02, наклон 0,0012, пересечение с осью
X—1925, р > 0,1; г — г = 0,48, г2 = 0,23, наклон 0,0043, пересечение
с осью X —1976, р < 0.00001; д — г = 0.98, г2 = 0.97, наклон 0,0047,
пересечение с осью X—1976, р < 0.000001; е — г = 0.11, г2 = 0.01,
наклон 0,0015, пересечение с осью X —1950, р > 0,1; ж — г = 0,32,
г2 = 0,10, наклон 0,0058, пересечение с осью X —1974, р < 0,01.
По Лукашову и Годсмиту [Lukashov and Goudsmit, 2002b]
184 Глава 5. Отдельные задачи эволюционного анализа
а
б
D 0.22 -
0.20-
0.18-
0.16-
0.14-
0.12-
0.10-
0.08-
0.06-
0.04
0.02-
0.00-1-
1974 1978 1982 1986 1990 1994
г
D 0.07-1
0.06-
год
год
Рис. 5.12. Эволюционные дистанции последовательностей участков ге-
нов env и pol от их общего предка для глобально полученных вариантов
ВИЧ-1 субтипа В. Использованы последовательности из США, Нидер-
ландов, Бразилии, Таиланда и России. Анализ проведен аналогично
приведенному на рис. 5.11. (а, б) — соответственно для индивидуальных
и средних за год синонимичных дистанций, метод NJ для гена env,
(в, г) — соответственно для индивидуальных и средних нуклеотидных
дистанций за год, метод ML для гена pol. Указаны 95% доверительные
интервалы, определенные с использованием индивидуальных дистан-
ций и соответствующего метода. Статистика: а — г = 0.33, г2 = 0.1L
наклон 0,0044, пересечение с осью X — 1976, р < 0,0000001; б — г = 0,06,
г2 = 0,92, наклон 0,0043, пересечение с осью X —1976, р < 0,00001;
в — г = 0,44, г2 = 0.20, наклон 0,0015, пересечение с осью X —1977.
р < 0.000001; г — г = 0.97, г2 = 0.94, наклон 0,0017, пересечение с
осью X — 1979, р < 0,000001. По Лукашову и Годсмиту [Lukashov and
Goudsmit, 2002b]
5.4. Анализ митохондриальной ДНК 185
тельно одинаковой до (общая ветвь субтипа) и после (индивиду-
альные ветви внутри субтипа) начала диверсификации внутри
субтипов, т. е. во время и после адаптации предка ВИЧ-1 (ви-
руса шимпанзе) к hobomj хозяину, человеку. В ранней истории
ВИЧ-1, до начала эпидемии массивные ограничения вирусной
гетерогенности по типу бутылочного горлышка (разд. 1.12) поз-
волили выжить только ограниченному числу вирусных вари-
антов — возможно, одному варианту для каждого субтипа, что
и привело к наличию длинных общих для каждого субтипа
ветвей. Дистанции таких выживших вирусов от общего для всей
группы М предка являются случайной величиной, и, по мнению
автора книги, скорость эволюции ВИЧ-1 в тот период не может
быть сколь-нибудь достоверно определена на основании дан-
ных о современной скорости эволюции этого вируса. Дискуссия
по этим вопросам между автором книги и группой Корбер
опубликована [Korber et al., 1998; Korber et al., 2000; Lukashov
and Goudsmit, 2000; Lukashov and Goudsmit, 2002b; Lukashov
and Goudsmit, 2003; Smith et al., 2003].
Так, наблюдая за изменением длины ростка (развитием эпи-
демии), мы можем ретроспективно достаточно точно определить
дату прорастания семени (начала эпидемии). Однако эти данные
ни в коей мере не помогут нам даже приблизительно определить
момент, когда это семя было брошено в землю и, тем более,
сколько времени это семя провело в сумке садовника.
5.4. Анализ митохондриальной ДНК
5.4.1. Особенности эволюционного анализа
митохондриальной ДНК
У подавляющего большинства эукариот, генетический материал
представлен не только ядерной, но и митохондриальной ДНК
(мтДНК), имеющей независимое эволюционное происхождение.
Кроме уже рассмотренных нами различий между универсаль-
ным и митохондриальными генетическими кодами (разд. 1.4,
табл. 1.3), генетика мтДНК имеет целый ряд принципиальных
особенностей по сравнению с ядерной ДНК, и эволюционный
анализ мтДНК по ряду причин представляет специфический
интерес.
Прежде всего для мтДНК характерна высокая скорость
эволюции при сохранении ее генетической структуры (числа
и видов кодируемых генов). В целом скорость эволюции мтДНК
186 Глава 5. Отдельные задачи эволюционного анализа
превышает таковую для ядерной ДНК в 5-20 раз [Brown et al
1979]. Высокая скорость эволюции мтДНК связана как с от'
сутствием эффективных систем репарации при ее репликд.
ции, так и с протекающими в митохондриях окислительно,
восстановительными процессами, приводящими к высокому со-
держанию свободных радикалов-мутагенов [Richter et al., 1988]
Более того, в последовательности мтДНК есть участки с еще
более высокой скоростью эволюции — два гипервариабельных
контрольных сегмента (Ziypervariable control regions, HVR1
и HVR2). Более высокая скорость эволюции позволяет исполь-
зовать анализ мтДНК для установления филогенетических от-
ношений между близкородственными организмами (например,
на уровне подвидов одного вида). При этом генетическая струк-
тура мтДНК весьма консервативна. Так, у всех млекопитаю-
щих длина мтДНК варьирует в узких пределах от 15 до 17
тысяч нуклеотидов и кодирует одни и те же 37 генов: 22 гена
транспортных РНК, 2 гена рибосомальных РНК и 13 генов
белков (подавляющее большинство митохондриальных белков
кодируются генами ядерной ДНК). Небольшой размер (совре-
менные методы позволяют легко определять полноразмерные
последовательности) при консервативности структуры (удобство
сравнения) делают анализ мтДНК весьма эффективным методом
установления филогенетических отношений. Даже1 у растений
и грибов, для которых характерны несравнимо большие раз-
меры мтДНК (миллионы нуклеотидов), мтДНК содержит гены,
присутствующие у всех эукариот.
Анализ мтДНК широко используется в филогенетических ис-
следованиях для установления эволюционных отношений между
формами жизни. В качестве примера рассмотрим анализ эволю-
ционных отношений между представителями семейства лошади-
ных, Equidae, отряда непарнокопытных, Perissodactyla. В насто-
ящее время (исключая вымершие виды) это семейство вклю-
чает в себя только один род лошадей, Equus, объединяющий
собственно лошадей, ослов, куланов и зебр (представители рода,
известные под другими тривиальными именами, как, например,
кианги, вероятно являются разновидностями этих видов, подви-
дами). Реконструкция эволюции семейства лошадиных, когда-то
весьма многочисленной группы (включающей 31 вымерший род),
непарнокопытных вообще (отряд включает в себя 17 семейств,
из которых до настоящего времени сохранились представители
только трех: лошадиные, тапировые, Tapiridae, и носороги,
Rhinocerotidae), является классическим примером достижений
5.4. Анализ митохондриальной ДНК 187
различные породы
► домашних
лошадей (2п = 64)
98(97)
японская порода домашних лошадей (2п = 64)
— лошадь Пржевальского (2п = 66)
I----монгольская порода домашних
лошадей (2п = 64)
-----осел Е. asinus (2п = 62)
- пустынная зебра Е. grevyi (2п = 46)
горная зебра Е. zebra (2п = 32)
Рис. 5.13. Филогенетические отношения между представителями
рода лошадей на основе анализа их мтДНК. Указаны значения
бутстрэп-анализа (при использовании двух различных филогенетиче-
ских методов). Указано число хромосом (2п). По Ишиде с соавт.
[Ishida et al., 1995]
палеонтологии. Тем не менее эволюционные отношения между
представителями рода лошадей до последнего времени являлись
предметом дискуссий (в некоторой степени, остаются спорными
до сих пор).
Одним из предметов дискуссии являются эволюционные отно-
шения между домашними лошадьми (вид Е. caballus) и лошадью
Пржевальского — единственной сохранившейся в мире настоя-
щей дикой лошадью (остальные дикие лошади, как, например,
американские мустанги, являются одичавшими домашними ло-
шадьми). Различными исследователями лошадь Пржевальско-
го классифицировалась и как отдельный вид, Е. przewalskii,
и как подвид дикой лошади, Е. ferus przewalskii, и как подвид
домашней лошади, Е. caballus przewalskii. Родственные отно-
шения между домашней лошадью и лошадью Пржевальского
настолько близки, что при скрещивании они дают плодовитое
потомство. При этом число хромосом у всех пород домашней
лошади и лошади Пржевальского различается и составляет (2п)
64 и 66 соответственно [Benirschke et al., 1965]. На протяжении
Длительного времени лошадь Пржевальского рассматривалась
Как предок домашних лошадей, а разница в числе хромосом
объяснялась слиянием хромосом у домашних лошадей. Одна-
Ко проведенный анализ мтДНК представителей рода лошадей
(рис.5.13) показал, что эволюционная ветвь лошади Пржеваль-
ского не является внешней по отношению к группе домашних
188 Глава 5. Отдельные задачи эволюционного анализа
лошадей [Ishida et al., 1995]. Данные этого анализа показываю?
что лошадь Пржевальского не может рассматриваться как пре.
док домашних лошадей. Более вероятным сценарием является
не слияние хромосом в эволюционной линии домашних лошадей
но разделение хромосом в линии лошади Пржевальского.
Другой особенностью генетики мтДНК, представляющей спе-
циальный интерес для филогенетических исследований, явля-
ется механизм наследования мтДНК исключительно (или прак-
тически исключительно) по материнской линии. У размножа-
ющихся половым путем организмов каждая соматическая клет-
ка содержит диплоидный набор ядерной ДНК две копии, оцня
из которых происходит от отцовского, а другая — от материн-
ского генома. В отличие от ядерной ДНК мтДНК содержится
в клетке не в двух, но в сотнях и тысячах копий (каждая
митохондрия содержит собственную мтДНК), и в подавляющем
большинстве видов (практически) все эти сотни и тысячи ко-
пий наследуются от матери. Причиной этого является то, что
митохондрии сперматозоида, расположенные в его хвостике,
либо вообще не проникают в яйцеклетку при оплодотворении,
либо разрушаются после проникновения [Sutovsky et al., 1999;
Sutovsky and Schatten, 2000]. Таким образом, все дети одной
матери (кроме гомозиготных близнецов) будут иметь различные
наборы ядерных генов, но все дети одной матери, даже от раз-
ных отцов, будут иметь одинаковую мтДНК. Более того, все дети
дочерей этой матери, т. е. все ее внуки по женской линии, также
будут иметь ее мтДНК.
На этом феномене наследования мтДНК исключительно
по материнской линии базируется сформулированная Уилсоном
концепция митохондриальной Евы (mitochondrial Eve), обще-
го предка мтДНК группы организмов, широко используемая,
в частности, при изучении происхождения человека [Сапп et al~,
1987; Di Rienzo and Wilson, 1991; Vigilant et al., 1991; Wilson and
Cann, 1992].
5.4.2. Концепция митохондриальной Евы и использование
анализа мтДНК при изучении происхождения человека
Все живущие сегодня люди (обозначим сегодняшнее поколение
как поколение П) унаследовали мтДНК от своих матерей (поко-
ление П — 1). Аналогично все эти матери поколения П — 1 ун»'
следовали мтДНК от их матерей (поколение П — 2), и так далее,
5.4. Анализ митохондриальной ДНК 189
Рис. 5.14. Митохондриальная Ева и семь последующих поколений.
Женщины (обозначены кружками) каждого поколения передают (со-
единение чертой) свою мтДНК дочерям. При репликации мтДНК могут
возникать мутации (различные мутантные формы мтДНК, гаплотипы,
обозначены кружками разной раскраски). Ряд женщин не оставляют
после себя дочерей (либо вообще не имеют детей, либо рождены только
сыновья). У женщин поколения 7 присутствуют различные гаплотипы
мтДНК, однако все они могут быть прослежены обратно во времени
к единому предку в поколении 0 митохондриальной Евы
по восходящей линии. Пример наследования мтДНК приведен
на рис. 5.14.
Рассматривая эту восходящую линию и исходя из принципа
наследования мтДНК по материнской линии, можно исключить
из анализа всех мужчин вообще. Более того, можно исключить
из анализа всех женщин, либо вообще не имевших детей, либо
имевших только сыновей: их мтДНК будет исключена из эволю-
ционного процесса. В каждом поколении следует рассматривать
только матерей, передающих свою мтДНК дочерям.
Естественно, что число живущих сегодня (поколение П) жен-
щин больше числа их матерей: среди живущих сегодня женщин
есть сестры, имеющие одну и ту же мать (соответственно, ее
мтДНК). Аналогично среди матерей сегодняшних женщин так-
же имелись сестры, унаследовавшие мтДНК от одной и той же
матери. Таким образом, в эволюционной истории человечества
190 Глава 5. Отдельные задачи эволюционного анализа
каждое предыдущее поколение имело все меньше и меньще
женщин, мтДНК которых унаследовано сегодняшним поколени-
ем П (строго говоря, меньше или столько же, но никогда не
больше).
Рассмотрим численный пример. Пусть некая современная
человеческая популяция, поколение П, включает в себя 100 жен-
щин. Из них две являются сестрами. Соответственно вся совре-
менная популяция женщин имеет мтДНК, унаследованную от
99 матерей предыдущего поколения П — 1. Пусть в поколении
П — i ни одна из женщин не имела сестер — соответственно все
эти женщины унаследовали мтДНК от 99 матерей предыдущего
поколения П — 2. Пусть в поколении П — 2 было три сестры.
Соответственно поколение П — 2 унаследовало мтДНК от 97 ма-
терей поколения П—3. Естественно, что и современное поколение
П унаследовало мтДНК от тех же 97 матерей поколения П — 3
(прабабушек поколения П). Эта логика справедлива для любого
поколения: каждое предыдущее поколение имеет все меньше
и меньше матерей, мтДНК которых было этим конкретным по-
колением и, значит, современным поколением П — унаследовано.
Соответственно, прослеживая эволюционную историю чело-
вечества обратно во времени, мы рано или поздно обязательно
придем к некому поколению 0, в котором была только одна
женщина, передавшая свою мтДНК всем живущим сегодня
людям: митохондриальная Ева (см. рис. 5.14). Естественно, эти
рассуждения применимы не только для человеческой популя-
ции, но для любых форм жизни, в которых мтДНК передается
исключительно по материнской линии.
Положение о том, что все современное человечество уна-
следовало мтДНК от одной женщины — безусловно, не означа-
ет того, что мтДНК всех людей одинакова. За время эволю-
ции человека в мтДНК происходили мутации (см. рис. 5.14),
и мтДНК человечества представлена рядом мутантных форм —
гаплотипов, объединенных в гаплогруппы (рис. 5.15, а). Но все
эти мутантные формы являются результатом эволюции мтДНК
одной женщины, происходят от митохондриальной Евы.
Подчеркнем, что существование митохондриальной ЕвЫ
не означает, что другие женщины и мужчины не внесли сво-
его вклада в генофонд человечества: речь идет исключительно
о мтДНК, в ядерной ДНК содержится как отцовский, так и мате-
ринский (в том числе и тех женщин, которые не имели дочерей)
генетический материал. Таким образом, митохондриальная Ева
не является последним общим предком человеческой популяции-
5.4. Анализ митохондриальной ДНК 191
а
Гаплогруппы мтДНК человека
предок человечества
L0 _______________________Ы_____________________
L2 L3 L4 L5 L6 L7
______м ________н_________________
QZD Е G Q Е A S Y IWX
С Z В F pre-HV pre-JT Р UK IWX
ну JT UK
Н V J т
Рис. 5.15. Отношения между гаплогруппами мтДНК человека (а)
и одна из возможных географических схем исторического заселе-
ния человеком земного шара (б). Суммарные данные многочисленных
исследований
Анализ распределения гаплогрупп в различных человеческих
расах и этнических группах является эффективным инструмен-
том при изучении происхождения современного человека (вид
Homo sapiens, человек разумный), заселения им земного шара,
формирования человеческих рас [Сапп et al., 1987; Di Rienzo
and Wilson, 1991; Vigilant et al., 1991; Wilson and Cann, 1992;
Torroni et al., 2006]. Филогенетический анализ последователь-
ностей мтДНК коренного населения Европы, Азии, Северной
и Южной Америки и Океании позволил установить эволюци-
192 Глава 5. Отдельные задачи эволюционного анализа
онное происхождение гаплогрупп их мтДНК от гаплогруппщ L3
давшей начало гаплогруппам М и N с их последующей дивер,
сификацией (рис. 5.15). Географически заселение земного Шара
можно проследить из региона Африканского Рога (территория
современных Эфиопии, Кении, Танзании) (рис. 5.15, б показывает
одну из вероятных схем миграции человека, наиболее общепри-
нятую, хотя по ряду вопросов дискуссии продолжаются). Более
того, молекулярные данные позволяют датировать различные
этапы происхождения и развития современного человека как
вида, заселения человеком земного шара (независимо от, но
дополнительно к палеонтологическим и археологическим дан-
ным).
Несмотря на общепринятость концепции митохондриальной
Евы, полученные с использованием этой концепции данные
имеют ряд ограничений. Кроме естественных технических огра-
ничений — в частности, по точности датирования событий (по-
нятно, что разброс результатов может достигать тысяч и даже
десятков тысяч лет), — подчеркнем фундаментальное положе-
ние: митохондриальная Ева современного человека, подвида Н.
sapiens sapiens, необязательно является представителем этого
подвида. Концепция митохондриальной Евы позволяет просле-
дить историю развития современного человека до того време-
ни, когда жила некая женская особь, от которой современ-
ным человеком была унаследована мтДНК — однако концепция
не подразумевает, что эта женская особь являлась представи-
телем вида Н. sapiens, подвида Н. sapiens sapiens. Так, эво-
люционный анализ относит митохондриальную Еву к перио-
ду в 130000-200000 лет тому назад. Принципиальный вопрос
вызывает информативность самой даты существования мито-
хондриальной Евы: что именно установленная дата говорит
о происхождении человека? Анализ мтДНК позволил достаточно
четко установить порядок заселения земного шара человеком
(хотя дискуссия по ряду вопросов продолжается) — однако этот
анализ в своей основе не направлен на решение вопроса о том,
сформировался ли современный человек, подвид Н. sapiens
sapiens, до или после заселения земного шара. В частности,
в какой степени неандертальцы (классифицируемые либо как
отдельный вид, Н. neanderthalensis, либо как подвид человека
разумного, Н. sapiens neanderthalensis) участвовали в формиро-
вании современного человека, его генофонда (если участвовали
вообще) [Green et al., 2006; Noonan et al., 2006; Wall and Kim,
2007].
5.5. Молекулярная эпидемиология 193
Как и мтДНК, Y-хромосома человека наследуется толь-
ко по одной родительской линии — отцовской. Аналогичная
рассуждениям о мтДНК логика может быть использована
и в отношении Y-хромосомы — в этом случае говорят об
Y-хромосомном Адаме. Исследование позволили датировать су-
ществование Y-хромосомного Адама периодом в 60000-90000
лет тому назад [Hammer, 1995; Jobling and Tyler-Smith, 1995;
Ke et al., 2001; Underhill et al., 2001a; Underhill et al.,
2001b].
5.5. Молекулярная эпидемиология
5.5.1. Задачи и принципы молекулярной эпидемиологии
Ранее в этой книге мы рассмотрели многочисленные вопросы,
связанные с теоретическими основами молекулярной эволюции
и практическими аспектами проведения эволюционного анализа.
В настоящее время эволюционный анализ широко применяется
в различных областях науки, в том числе и не связанных
исторически с вопросами эволюции. Рассмотрим возможности
эволюционного анализа в одном из традиционных направлений
медицины — эпидемиологии.
Понятие эпидемии (epidemic), изначально определяемое как
вспышка и распространение инфекционного заболевания, в на-
стоящее время понимается более широко и применяется
и к неинфекционным заболеваниям. Тем не менее изуче-
ние эпидемий инфекционных заболеваний и разработка мер
по их предотвращению и ограничению безусловно являют-
ся ключевыми задачами эпидемиологии и включают в себя
спектр вопросов, связанных с установлением источника и пу-
тей распространения инфекции, мониторингом появления но-
вых, более опасных, вариантов патогена (например, вариан-
тов с лекарственной устойчивостью) и прогнозом развития
эпидемии. Возможность быстрого получения и анализа боль-
шого количества генетической информации позволяет решать
зти традиционные задачи эпидемиологии на молекулярном
уровне.
Рассмотрим роль и возможности эволюционного анализа
в изучении эпидемиологии инфекционных заболеваний на при-
мере эпидемии ВИЧ-1.
Как уже отмечалось, для многих вирусов, в особенности
РНК-содержащих вирусов, включая ВИЧ-1, вирусы гриппа, ге-
13 —2238
194 Глава 5. Отдельные задачи эволюционного анализа
патита С и др., характерна высокая скорость эволюции, превы-
шающая для наиболее изменчивых участков генома 1% в год.
Соответственно обнаружение у инфицированных лиц близко-
родственных вариантов вируса свидетельствует о том, что эти
лица были (недавно) инфицированы из одного источника. Таким
образом, высокая изменчивость вирусов дает возможность уста-
навливать эпидемиологическую связь между инфицированными
лицами на основании анализа филогенетических отношений
между инфицирующими их вариантами вируса: чем ближе эво-
люционные отношения между вариантами вируса, тем, в об-
щем случае, ближе эпидемиологическая связь между лицами,
от которых они получены. Это положение является базовым
принципом молекулярной эпидемиологии. В наиболее простых
случаях этот принцип применяется при установлении источника
заражения инфицированного лица для доказательства эпиде-
миологической связи между донорами и реципиентами вируса
(разд. 5.5.2). Однако возможности применения этого принципа
гораздо шире.
Как отмечалось в разд. 5.3.7, неравномерность распределения
генетических субтипов ВИЧ-1 в мире является результатов
многочисленных эффектов основателя (разд. 1.12), имевших ме-
сто в истории пандемии ВИЧ-1, когда, попав в восприимчи-
вую человеческую популяцию, специфический вариант виру-
са распространяется в ней (разд. 5.5.3). По мере расширения
знаний о механизмах и пределах изменчивости ВИЧ-1, а так-
же накопления значительного числа генетических последова-
тельностей ВИЧ-1 из различных стран и групп риска, растет
понимание того, что пандемия ВИЧ-1 может быть рассмотре-
на как сумма отдельных вспышек или эпидемий, протекаю-
щих в географически, социально и/или поведенчески очерчен-
ных человеческих популяциях, или эпидемиологических сетях.
Соответственно понимание эпидемического процесса подразу-
мевает изучение эпидемиологических характеристик этих се-
тей, включая изучение их конфигурации, стабильности и вза-
имосвязей между различными эпидемиологическими сетями.
При этом варианты ВИЧ-1, циркулирующие в различных эпи-
демиологических сетях, вообще говоря, генетически различ-
ны. Генетические различия между циркулирующими в раз-
личных популяциях вариантами ВИЧ-1 могут быть исполь-
зованы в качестве генетических маркеров для исследования
многочисленных традиционных эпидемиологических вопросов
(разд. 5.5.3).
5.5. Молекулярная эпидемиология 195
5.5.2. Установление источника заражения
Одним из первых опытов практического применения базового
принципа молекулярной эпидемиологии являлся анализ возмож-
ного непреднамеренного инфицирования зараженным ВИЧ-1 ме-
дицинским сотрудником нескольких пациентов при проведении
медицинских манипуляций в ротовой полости, так называе-
мый случай дантиста из Флориды, США [Ou et al., 1992].
В дальнейшем аналогичный подход был использован в ряде
исследований при анализе эпидемиологической связи между
донорами и реципиентами вируса. В ряде случаев возможного
преднамеренного заражения ВИЧ-1, молекулярные данные были
использованы в качестве судебных доказательств [Albert et al.,
1994; Cornelissen et al., 1995].
Были выработаны и математически проанализированы основ-
ные принципы такого молекулярно-эпидемиологического подхо-
да. Используемым критерием при таком анализе является стати-
стически достоверная филогенетическая связь между генетиче-
скими последовательностями представляющих интерес вирусов
(т.е. вирусами возможных донора и реципиента), при отсутствии
связи этих вирусов с так называемыми местными контролями,
т. е. вирусами, полученными из той же человеческой популяции
(та же географическая местность и группа риска). Для статисти-
ческой оценки филогенетических отношений широко использу-
ется бутстрэп-анализ (разд. 4.4). При этом в анализе использу-
ются нуклеотидные последовательности, более информативные
по сравнению с аминокислотными. Большинство предыдущих
исследований такого рода были выполнены на основе анализа
генетических последовательностей области V3 поверхностного
белка gpl20 ВИЧ-1, что связано со значительной изменчивостью
этого участка генома ВИЧ-1, расширяющей шкалу измерений,
а также с наличием большого числа предварительно полученных
местных контролей. Однако молекулярно-эпидемиологический
анализ может быть в принципе выполнен и при использовании
любой другой области генома ВИЧ-1.
5.5.3. Анализ эпидемиологических сетей
Как уже отмечалось в разд. 5.3.7, варианты ВИЧ-1 образуют три
генетические группы: наиболее многочисленную (> 99% всех
инфекций ВИЧ-1 в мире) главную группу М, а также группы О
и N (см. рис. 5.10). Эти три группы ВИЧ-1 являются результатом
трех независимых заносов вирусов иммунодефицита шимпанзе,
196 Глава 5. Отдельные задачи эволюционного анализа
SIVcpz, в человеческую популяцию. В группе М выделяют ге-
нетические субтипы A-К (кроме Е). Для некоторых субтипов
выделяют также суб-субтипы (например, А1 и А2). Кроме то-
го, существуют варианты ВИЧ-1, являющиеся рекомбинантами
различных субтипов. Выделяют уникальные рекомбинантные
формы (УРФ, URF, unique recombinant form), обнаруженные
у одного или нескольких инфицированных лиц, и циркулирую-
щие рекомбинантные формы (ЦРФ, CRF, circulating recombinant
form), присутствующие у значительного числа инфицированных
лиц [Robertson et al., 2000]. Согласно принятой классификации,
наименование циркулирующей рекомбинантной формы включает
присвоенный ей порядковый номер и субтипы родительских ге-
номов (например, CRF14 BG — циркулирующая рекомбинантная
форма 14, результат рекомбинации между вариантами ВИЧ-1
субтипов В и G). В настоящее время официально выделяют
более сорока различных ЦРФ. Ряд вариантов ВИЧ-1 группы
М не могут быть однозначно классифицированы как принадле-
жащие определенному субтипу или являющиеся рекомбинантом
каких-либо субтипов. Такие варианты называют неклассифици-
руемыми и обозначают индексом U (unclassified).
Анализ вариантов ВИЧ-1, циркулирующих в различных ре-
гионах мира, показал неравномерность глобального распределе-
ния генетических субтипов ВИЧ-1. Географически наиболее рас-
пространенным субтипом ВИЧ-1 является субтип В [Myers et al.,
1995]. Варианты ВИЧ-1 этого субтипа связаны с развитием
эпидемий во многих развитых странах мира, включая страны
Северной Америки, Западной Европы, Австралию, Японию и др.
Однако субтип В не является доминирующим с точки зрения
числа вызванных инфекций и случаев СПИД, поскольку эпиде-
мии в эпицентре пандемии СПИД — странах Африки к югу от
Сахары — обусловлены вирусами других субтипов, прежде всего
субтипа С. Варианты ВИЧ-1 именно субтипа, циркулирующие
также в Индии и Китае, вызвали наибольшее число инфек-
ций ВИЧ-1 в мире. Эпидемия ВИЧ-1 на территории бывшего
СССР обусловлена распространением вариантов ВИЧ-1 субтипа
А. В целом около 90% случаев инфекции ВИЧ-1 в мире связаны
с вирусами субтипов А, В и С, в то время как для некоторых
субтипов, как, например, субтипы I или К, описаны только
единичные случаи инфекции.
Такое неравномерное распределение субтипов ВИЧ-1 в ми-
ре является результатом многочисленных эффектов основателя
(разд. 1.12): попавший в восприимчивую человеческую популя-
5.5. Молекулярная эпидемиология 197
цию, определенную эпидемиологическую сеть, специфический
вариант вируса быстро распространяется в ней.
Для того чтобы продемонстрировать подходы и возможности
генетического анализа при изучении распространения эпидемии
ВИЧ-1, установления характеристик эпидемиологических сетей,
рассмотрим несколько примеров.
В Нидерландах на протяжении 1980-1990-х гг. основными
группами риска для инфекции ВИЧ-1 и СПИД являлись мужчи-
ны с гомо- и бисексуальной ориентацией (ГБМ) и инъекционные,
или внутривенные, наркоманы (ВВН). Первые случаи СПИД
среди ГБМ в Нидерландах были зарегистрированы в 1982 г.,
среди ВВН — в 1985 г. Ретроспективные серологические исследо-
вания показали, что первые случаи инфекции ВИЧ-1 среди ГБМ
и ВВН относятся к 1977 и 1980 гг. соответственно. На основании
этих данных, считалось, что начало эпидемии ВИЧ-1 среди ВВН
в Нидерландах положила передача ВИЧ-1 от ГБМ к ВВН.
Циркулирующие в обеих группах риска варианты ВИЧ-1
относятся к одному и тому же субтипу В ВИЧ-1. Однако филоге-
нетический анализ выявил различия между вариантами ВИЧ-1
от ГБМ и ВВН в Нидерландах: полученные от ВВН последова-
тельности ВИЧ-1 формировали монофилетическую группу, спе-
цифичную для этой группы риска [Kuiken and Goudsmit, 1994;
Kuiken et al., 1996]. Дальнейший анализ последовательностей
вариантов ВИЧ-1, циркулирующих среди ГБМ и ВВН в Нидер-
ландах, выявил ряд генетических различий между ними в раз-
личных участках генома. Наиболее консервативной, характер-
ной генетической особенностью вирусов, циркулирующих среди
ВВН, является синонимичная замена в кодоне второго глицина
верхушечного тетрапептида GPGX области V3 гена env [Kuiken
and Goudsmit, 1994; Kuiken et al., 1996]. У вариантов ВИЧ-1
от ВВН этот глицин кодируется кодоном GGC, в то время как у
вариантов ВИЧ-1, циркулирующих среди ГБМ, эта аминокислота
кодируется другими синонимичными кодонами (здесь и далее
в кодонах подчеркнуты нуклеотиды, специфичные для ВВН).
Подобных различий между вариантами ВИЧ-1 от ГБМ и ВВН
не выявлено в ряде других стран. Исходя из этого, а также из си-
нонимичной природы большинства различий между вариантами
ВИЧ-1 от ГБМ и ВВН в Нидерландах, можно заключить, что эти
различия являются не результатом адаптации ВИЧ-1 к разным
группам риска, но следствием эффекта основателя (разд. 1.12).
Дальнейшие исследования показали, что филогенетически близ-
кие варианты ВИЧ-1 с кодоном GGC циркулируют среди ВВН
198 Глава 5. Отдельные задачи эволюционного анализа
в США (рис. 5.16) [Lukashov et al., 1996b]. Таким образом, наи-
более вероятным сценарием начала эпидемии ВИЧ-1 среди ВВЦ
в Нидерландах является ограниченное число заносов (возможно,
даже один занос) вируса из географически более отдаленной, но
поведенчески единой эпидемиологической сети — ВВН из США.
Специфичность замены GGC для ВВН в Нидерландах поз-
воляет использовать эту генетическую особенность в качестве
маркера при исследованиях характеристик эпидемиологических
сетей: наличие вируса с такой заменой позволяет с высокой
степенью уверенности предполагать, что этот вирус эпидемио-
логически связан с ВВН. Показано, что в дальнейшей эпи-
демии ВИЧ-1 в Нидерландах новые случаи инфекций среди
ГБМ и ВВН вызваны специфичными для этих групп риска
вирусами [Lukashov and Goudsmit, 1997b]. Эти данные свиде-
тельствуют о том, что на протяжении всей эпидемии популяции
ГБМ и ВВН в Нидерландах являются двумя стабильными,
практически не зависимыми друг от друга эпидемиологически-
ми сетями, несмотря на определенное перекрывание этих двух
человеческих популяций. Передачи вирусов происходят главным
образом внутри, а не между этими популяциями. С другой
стороны, продемонстрированы эпидемиологические связи между
популяциями ВВН в различных европейских странах, приводя-
щие к проникновению и распространению вирусов с признаком
GGC среди ВВН в странах Центральной и Южной Европы, где
начало эпидемий ВИЧ-1 было связано с циркуляцией других
вирусов [Op de Coul et al., 2001b]. Взятые вместе, эти данные
демонстрируют значительную роль социальных связей в распро-
странении ВИЧ-1 в США и Европе.
Последующее развитие эпидемии ВИЧ-1 в Нидерландах,
с середины 1990-х гг. по настоящее время, характеризуется
ростом пропорции лиц, инфицированных гетеросексуальным
путем, в общем числе инфицированных ВИЧ-1. Анализ гене-
тических последовательностей вариантов ВИЧ-1, циркулирую-
щих в этой группе риска, выявил несравнимо большую их
гетерогенность, по сравнению с вариантами ВИЧ-1 у ГБМ
и ВВН [Lukashov et al., 1996а]. В этой группе риска были выяв-
лены варианты вируса, представляющие практически все субти-
пы ВИЧ-1. При этом основным фактором, связанным с инфици-
рованием тем или иным субтипом ВИЧ-1, являлось географиче-
ское происхождение и принадлежность инфицированного лица
к определенной этнической группе. У лиц, постоянно прожива-
ющих в Нидерландах, но происходящих из имеющих эпидемио-
5.5. Молекулярная эпидемиология 199
IUK.4660
HDE3497
IUK4663C1
HUK4861
HDE3499
Н0316 85
Н0457 85
IUS13211 91
IUS11437 89
Н0545 88
HUK4686
Н0491 85
Н116087
< 1US131 89
Н1182 86/ ~
87 / H1029 86
89
HUK4656
Н0169 8
TUS11316 91
10138 89
HUK4665C1
Н0371.88
Н0658 86
1083 87
13048
88
IUK4657c2
ч 1US10714 90
1UK4662
14008 85
87\ “
IUS10101 88
IUK4658 HUK4664
IUS10164 89
Hl 161 87
H0207 8
H0495 85
10085 90 H0001 85
H1211-87
1031187
Н0434
1306292
Hl234 88
HRUS4-™M
ARV-2 Hl047 88
H0547 88
/H0026J86
HRU
IUS11629 91
Hl135-86
14004_88
11035 87
JK4655T
13007 86
[US11&4J91
последовательности от ВВН
Рис. 5.16. Филогенетический анализ последовательностей области V3
гена env вариантов ВИЧ-1 от ГБМ и ВВН из Европы и США. Выделен
филогенетический кластер последовательностей от ВВН. В кодах после-
довательностей первая буква указывает группу риска (Н—ГБМ, I —
ВВН), следующие две или три буквы — страну происхождения образцов
(DE — Германия, RUS — Россия, UK — Великобритания, US — США, не
указано — Нидерланды), последующие цифры и буквы — лабораторный
код образца, последние две цифры после знака «_»— год получения
образца. Последовательности с другими кодами использованы в качестве
контролей. По Лукашову с соавт. [Lukashov et al., 1996b]
200 Глава 5. Отдельные задачи эволюционного анализа
логические связи с эндемичными для ВИЧ-1 регионами, как пра-
вило, присутствуют субтипы ВИЧ-1, специфичные для этих ре-
гионов [Lukashov et al., 1996а]. Таким образом, в отличие от ГБМ
и ВВН, лица, инфицированные гетеросексуальным путем, не об-
разуют в Нидерландах единую независимую эпидемиологиче-
скую сеть. Этнические (географические) отношения играют су-
щественную роль в распространении ВИЧ-1 в этой группе риска.
Несмотря на значительное распространение не-В субтипов,
вирусы субтипа В были обнаружены в Нидерландах более
чем у половины лиц, инфицированных гетеросексуальным пу-
тем [Lukashov et al., 1998b; Op de Coul et al., 2001а]. Фи-
логенетический анализ последовательностей ВИЧ-1 субтипа В,
полученных от лиц, инфицированных гетеросексуальным путем,
выделил две группы последовательностей, соответствующих на-
личию или отсутствию признака GGC (см. рис. 5.17). Последу-
ющий анализ вариантов ВИЧ-1 субтипа В показал, что из 54
проанализированных вирусов, 22 (41%) имеют специфичный
для ВВН признак GGC (рис. 5.17). Кроме того, у последователь-
ностей с признаком GGC достоверно чаще наблюдались и другие
специфичные для ВВН генетические маркеры ВИЧ-1. Признак
ТСС был обнаружен у 17 из 22 (77%) последовательностей
с признаком GGC и только у 1 из 32 (3%) последовательно-
стей без признака GGC. Треонин в отмеченной на рис. 5.17
позиции был выявлен у 25 из 32 (78%) последовательностей
без признака GGC, в то время как только у 19 из 22 (86%)
последовательностей с признаком GGC в этой позиции содержа-
лась другая аминокислота (признак не-Т). Наличие изолейцина
в отмеченной на рис. 5.17 позиции было выявлено у 16 из 22
(73%) последовательностей с признаком GGC, по сравнению с 5
из 32 (16%) последовательностей без этого признака (признак I).
Статистические данные для всех признаков указаны на рис. 5.17.
Все, кроме одной последовательности с признаком GGC,
принадлежали к единой с консенсусом ВВН из Амстердама
филогенетической группе, в то время как все, кроме одной
последовательности без признака GGC, принадлежали к другой
группе. Бутстрэп-анализ был выполнен отдельно для каждой по-
следовательности с признаком GGC, исключая все остальные по-
следовательности с признаком GGC. При таком подходе среднее
значение бутстрэп-анализа для группирования индивидуальных
последовательностей с консенсусом вариантов ВИЧ-1 от ВВН
составляло 73 (от 60 до 96, рис. 5.17).
5.5. Молекулярная эпидемиология 201
Таким образом, вирусы с признаком GGC были обнаружены
почти у половины лиц, инфицированных гетеросексуальным
путем вариантами ВИЧ-1 субтипа В. Эти данные показывают
что в Нидерландах заносы ВИЧ-1 от ВВН являлись важным
источником эпидемии среди лиц, инфицированных гетеросексу-
альным путем.
Рассмотрим развитие эпидемии ВИЧ-1 на территории быв-
шего СССР. В этой эпидемии выделяются два периода. Началь-
ный период эпидемии с середины 1980-х до 1994-1995 гг. ха-
рактеризовался относительно низким уровнем распространения
ВИЧ-1, когда общее число зарегистрированных на территории
бывшего СССР случаев инфекции ВИЧ-1 составляло около 1000.
Показано, что этот период эпидемии был связан с циркуля-
цией различных субтипов ВИЧ-1 в различных группах риска:
варианты ВИЧ-1 субтипов А и С доминировали среди лиц,
инфицированных гетеросексуальным путем, субтипа В — среди
ГБМ, субтипа G — среди лиц, инфицированных при проведении
медицинских манипуляций (так называемая элистинская вспыш-
ка) [Bobkov et al., 1994а; Bobkov et al., 1994b; Lukashov et al.,
1995]. При этом филогенетический анализ показал, что уровень
генетической гетерогенности среди вариантов ВИЧ-1 одного
субтипа, кроме элистинского случая, является характерным
для эпидемиологически прямо не связанных между собой слу-
чаев инфекций. Эти молекулярные данные позволяют сделать
заключение о том, что большинство случаев инфекции ВИЧ-1
в тот период были связаны с независимыми заносами ВИЧ-1
из разных эпидемиологических очагов. При этом молекулярные
данные позволяют заключить, что значительное число инфекций
ВИЧ-1 среди ГБМ в бывшем СССР связано с эпидемией ВИЧ-1
субтипа В среди ГБМ в Европе. В свою очередь значительное
число случаев инфекций ВИЧ-1 среди лиц, инфицированных
гетеросексуальным путем, связано с эпидемией ВИЧ-1 в Аф-
рике к югу от Сахары. В целом, эпидемия ВИЧ-1 в бывшем
СССР в этот период характеризовалась значительно большим
генетическим разнообразием циркулирующих вариантов ВИЧ-1,
по сравнению с эпидемиями в других странах Европы и США,
связанных с одним субтипом ВИЧ-1 — субтипом В.
После середины 1990-х гг. на территории бывшего СССР
регистрировался лавинообразный рост эпидемии ВИЧ-1 — как
по числу инфицированных ВИЧ-1 лиц, так и по числу затрону-
тых эпидемией регионов. Общее число зарегистрированных за
этот период случаев инфекции ВИЧ-1 составляет сотни тысяч,
не-GGC последовательности
значение
GG£ ТСС не-Т I бутстрап-анализа
консен-В WIRSENFTDNAKTIIVQLNESVEINCTRPNNNTRKS IHIGPGRAFYTTGEIIGDIRQAHCNI SRAKWNNTLKQNIKKLREQFGN -KTIVF -----
конс-ГБМ мгпос л м v ______ д —K— AT -———P—
(jjl IN — _ — у А XX /XI 1 . . т ГКА-К R м ___ д г» ——— a RGE
INUyULZ ЫГПО1 1 D —— * 1 UAA А — —1* — IN A U охги . = jq у д р р д г) — Q Т NI I. -G HN —F
INtiJjy 1 IO TTTDOT Ofl v >ж ** ж т w т *д ~ r д т G к X-
1 U КУ I ZU TT'Dfl! от I A Т AT R—V К G Т
l ОКУ1ZI 1 А 1 А I А 1 XX VI 1 кт т ти ш v т Г' Q д ЛТП—ТСП Е V V— ——
NED9122 Гч 1 |Н 1 К I <л О A Vr vriy FxJJ Ki V V . .
Г11ГО1 от j j qrj j Д— —T—CIV R V T К G— V GGE GN N
CriiyiZo CTTDO1 ОЛ т v т " т U PT pm — О Т.ТП Г) AD KV —N — I
bUKUlZD Г' ppnoon I XX 1 1 XX ~Г i — Fin ч lx 1 X> U IX' 1 ’ A лм I imA G RP. — v (?) TN n Q -—V I _ _
vr Hi tvy Zoo U IN lx FxU/x Vr HU X1 W Нт и _ 4 » . . * A KT T V -.Г1 — A — T TI П T К
TUR9340 A N I— — К G A ~ —— Lx 1 1 U 1 XX
EUR9346 _ T VMD_n_T_V - N — Д— M— ТП M_ - V—
— — L, ixJr Г W 1 V — — — — — — — — — — — IN —— л IN 1U IN v . . д g_ p T, т —D—R—AN R— . . —————
NED9454 KI V A T —Г' nv т О R R— R T —
NED9456 у у ——A A l lx 1 Vt о Xj . XX 1
икал «хс SKT — GR — — Д D T, ED T G К
N у T *" AD I T EK R AM G R _
CUR9461 > cm " TN VA— TN К - E R—VAT Q NK T — —
MOK94t>3 | OlN V 1 —N< lj 1У I* A UN il 1X IV VAI INXX . . X I 17 п к д V AD _____ -K ROA— E I
SUR9465 | -y и A V A U xX о w xx xx . . x N РД V T I MT TT П— N KN T N A R
IND9466 zr K'_P_K'_Z_L ~ П A n___— EGE R R—V—E K— ——
DOM9467 у —ЧЛ xx—Г—xx — — —— v_r — и HiIjCj О XX V Xj xx . . IT (1 T R N — W N — — T STR П T К I
AKG95Z0 к U __—£ __________ IVIN W IN lx О X 1 v XX X XX ..X T N_ A A UP A I. PR VAI К V —
SUR9580 1 *N AA — К f A lx Г IV VAI A V . . ki T7 MT_I7 GKN G E KN ——
FRA9588 y- —jn I—lx ______ VxxxJN Cj xxxt .
TUR9590 L T К P S A A- D D R T К . - — — — — —
SUR9591 ! -A—IS Q S A D GKQ D RV Q-H-NK. . 1- E T TTP tv vni T T Д n—— — т О I К
DOM9594 —______ —I Ax — — —I V V UIx Lx 1 A 1 * XX . . vr Л v p k T ЕД П _____ KGE R V- E К
DOM9596 У A—Г—xx 1 TA lx rxVlXx XV V Xx XX . IM V n V — — — n kt G KY V — A
SO K95100 ———jj— A U V ———————— ——— у xxi тл xxi v . - xx t т _v_i> .A д ri П E K— T —
TUR96102 1 А Г A A — U XX Hl XX X
NED96104 N ! TR-R 1 A ES N—К Y T-NG S QKV К . . 1 V P К- П T W——— PC. T KTI. n — E г> T К NITI - - + -
NED96110 — ——— A F A U 1 xl IVLx Lx A1 Lx U u xx x XX . .1'1 x ж л О P XI A I Д П—niCVAR—— —— А
NED96114 DOM96115 К Xl —————— A lx А и ЧЛУАЛ . . n -I E T T-V TOL L—T—A—D EK Q 1 К . . A- - - - + -
GGC последовательности
коне-ВВН
NED881
NED883
CUR885
NED897
FRA898
NED899
NED9013
РАК9117
BEL9125
SUR9231
SUR9236
ANT9237
NED9339
NED9349
MOR9573
RLS9578
ТНА9585
W$32
NED96106
NED96108
NED96109
----D---f--------А-----------------------------------L---D-----j-------к_ ----
------------------------------------------------------------------------------s—N---------------------------------------------------------------------------N--------------------------------------------------------------------------GT-D-------К—I----. .-
--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------1-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------р------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------КТ----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------д—у-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------. .----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------
--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------N 1-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------L L--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------L—Т—KD—RK-AI--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------Е-. . 1
--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------А N 1---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------N--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------Р-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------А------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------D—Е---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------I------Y—. .-
-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------IS—S-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------1------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------А-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------Р----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------Q—А-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------R—I----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------. .-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------
-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------А—S Т------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------Q-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------Т----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------К---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------L— Е—К ЕК—I-НК..
-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------А- —Т I------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------S-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------А--------------------D—L— Е—D------------М-1---------LN . . Т-
-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------N V--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------V-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------Р------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------D-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------L----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------Q-D-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AI —G--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------К- . . *------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------
-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------N----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------j--д-G-T-Д-v-L-D-------i _G---к -s
---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AN-S-К—V-A—S A—Q-L—TD—D-1-EQFRN-
----A------V-----------Q----------S-R---A DV---------L---------D--------T-.NT
--------------------------------------------------------------------------A-T I-NP-L—ТЕ-N-M-N-. .-
I---SN----S---- A------------------- T--A----------------------AT------KEKN---
----A---T-I------NP-K------------К-------------------L—ТЕ------N-M-----К . .----
--------------------------------------------------------------------------------N j-----------------------------------------------------------------------------KP---------------------------------------------------------------------------p---------------------D--------------------L-KTQ—E—R—AI—К------KT. .-
-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------N------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------у----R—p-д—D---TA-R— j-- -
I-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------s N 1-Q-Д TL A DV-К ED К AI-V-. . E-
—V—A-----T-S--------------S---------G---A T-------------К--------AI----N- . .---------------------
----N 1 ---------Q---------------s ST-----------К--------GO D----К— I-. .-----
------------------------------------------------------------------------------N N---------------------------------------------------------------------------у--------------------------------------------------------------------------PM--Д-д T__ ДЛ-------K—E_ .-
------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------A N S-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------KD—К---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------NM-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------A-EQ-V-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------L-E—D----------1---------К E- . .-1 -
----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------т 1--Q-P----D-E-E E -A
ft----------------------------------------------------------------------------t-t
TC£ не-Т GGC I
корреляция с признаком GGC, коэффициент Пирсона
p<0,001 для всех корреляций
0,77 0,63 0,58
Рис. 5.17. Аминокислотные последовательности области V3 гена env вариантов ВИЧ-1 субтипа В от лиц в
Нидерландах, инфицированных гетеросексуальным путем. Последовательности представлены в сравнении с кон-
сенсусами ВИЧ-1 субтипа В (конс-В) и вариантов ВИЧ-1 от ГБМ (конс-ГБМ) и ВВН (конс-ВВН). Индивидуальные
последовательности отнесены к двум группам, в соответствии с наличием у них признака GGC. Позиции
и признаки, отличающие последовательности ГБМ и ВВН, указаны внизу. Справа указано наличие (+) или
отсутствие (—) признаков у индивидуальных последовательностей, а также значение бутстрэп-анализа для их
принадлежности к филогенетической группе ВВН. По Лукашову с соавт. [Lukashov et al., 1998b]
204 Глава 5. Отдельные задачи эволюционного анализа
по оценкам — превышает миллион. Эпидемиологические данные
показывают, что этот резкий рост эпидемии связан с быстрым
распространением ВИЧ-1 среди ВВН, а позднее — среди лиц,
инфицированных в результате гетеросексуальных связей.
Генетический анализ вариантов ВИЧ-1, полученных из раз-
личных регионов бывшего СССР, показал, что этот лавино-
образный период эпидемии связан с быстрым распростране-
нием гомогенной популяции ВИЧ-1 субтипа А, обозначенной
ВВН-А [Liitsola et al., 1996; Bobkov et al., 1997; Bobkov et al.,
1998; Novitsky et al., 1998; Lukashov et al., 1998a; Lukashov et al.,
1999; Nabatov et al., 2002; Lazouskaya et al., 2005]. В целом
этот вариант ВИЧ-1 обусловил более 95% всех случаев инфекции
на территории бывшего СССР. Кроме того, было показано, что
вспышка эпидемии ВИЧ-1 в Калининградской области связана
с распространением мозаичного вируса, обозначенного ВВН-А/В
и являющегося результатом рекомбинации между вариантом
ВВН-А и вирусом субтипа В [Bobkov et al., 1998; Liitsola et al.,
1998; Lukashov et al., 1999].
Появление этого рекомбинантного вируса подразумевало, что
его второй родительский вирус, вариант ВИЧ-1 субтипа В, обо-
значенный ВВН-В, также циркулирует в популяции ВВН на тер-
ритории бывшего СССР. Однако на протяжении нескольких лет
циркуляция этого вируса в каком-либо эпидемическом очаге
на территории бывшего СССР выявлена не была. В дальнейших
исследованиях было показано, что вариант ВВН-В циркулирует
среди ВВН в Николаеве, Украина [Nabatov et al., 2002], что
позволило реконструировать историю развития лавинообразной
эпидемии ВИЧ-1 на территории бывшего СССР с помощью
анализа генетических данных (рис. 5.18).
Начало этой эпидемии положили два независимых заноса
ВИЧ-1 в среду ВВН на юге Украины незадолго до 1995 г.: вируса
субтипа А (ВВН-А) в Одессе и вируса субтипа В (ВВН-В) в Нико-
лаеве [Nabatov et al., 2002]. Данные о большей гетерогенности
популяций ВИЧ-1 в этих двух городах, по сравнению с други-
ми очагами эпидемии, соответствуют большему возрасту этих
эпидемий [Nabatov et al., 2002]. На основании показанных гене-
тических особенностей вариантов ВИЧ-1 субтипа В среди ВВН
в Европе и бывшем СССР можно заключить, что эти две эпиде-
мии эпидемиологически не связаны между собой [Bobkov et al.,
2000]. Из двух начальных вирусных популяций, ВВН-А и ВВН-В,
вариант субтипа А получил значительно большее распростране-
ние и вызвал последующие эпидемии ВИЧ-1: сначала (1996 г.)
5.5. Молекулярная эпидемиология 205
Рис. 5.18. Происхождение и распространение лавинообразной эпидемии
ВИЧ-1 на территории бывшего СССР
на юге России и в Беларуси, затем — по всей территории России,
в Молдавии, Латвии, Казахстане, Узбекистане и других странах
на территории бывшего СССР. Циркуляция двух различных
популяций ВИЧ-1, ВВН-А и ВВН-В, в одной группе риска
привела к их рекомбинации и образованию циркулирующей
рекомбинантной формы ВВН-А/В, вызвавшей эпидемию в Ка-
лининградской области России.
Доминирующая в эпидемии на территории бывшего СССР по-
пуляция вирусов ВВН-А отличается высокой гомогенностью. Ге-
нетические дистанции между последовательностями вариантов
ВИЧ-1, полученными в конце 1990-х гг. из очагов, разделенных
тысячами километров, не превышали в среднем 1-2%. В преде-
лах отдельных очагов гомогенность вирусных популяций была
еще выше. Так, полученные от 12 из 20 инфицированных ВИЧ-1
лиц из Светлогорска, Гомельская область, Беларусь, последова-
тельности области V3 были идентичными, а оставшиеся 8 по-
следовательностей имели от 1 до 7 нуклеотидных замен по срав-
206 Глава 5. Отдельные задачи эволюционного анализа
KOHC-Св VMlRSEXlTDNGKlllVqLTEn'NlTClRPaNNTBTSlRlGraqTrYATGDVlGDlRKAYCNSSlAjmeTLqKlBTQUtKYFNNK'nir
А974696
А974697 ----------------- ----------
А974698 - --------------------- -----------------------------------------
А974699 --------------------------------- ----------------------------------------------— ________
А974700 -----------------
A97470I -N - ------------ -----------------------------------------------------Я
А974703 -
А974704 -------------------------------------------------------------------------------- -_______
А97470 ------------------------------
А974706 — К-------------- --С-- -----------------------------------«-
А974707 ---- ------ -Н --------------------------------------------------- -------------------
А974708
А974709 - ------------ ----------------
A9747I0 -------К-- ---------------------- -------------------------------------------------------
A9747I1 -------------------- ———--------------------------------------------------------------В
A9747I2 ---------------------------------------------------------------------
A9747I3 ——К -К----------- N ------------------ ---------------- --------1
А974714 I------К------Т------------- -А-Т--------------------------Н- - -R---------—
А974715 -------К- ----------------------А------------ ----------------------
А974724 ---------- - ---------------------- --------- ------- ---------------- _
кокс-А IV -N-A-T- VK К N-T—N— -K-VH A -I Q Н- -ТЕ--К QVAB-
КОНС-УР ------------------------------------------- -----------
Рис. 5.19. Аминокислотные последовательности участка V3 гена env
вариантов ВИЧ-1, циркулирующих в Светлогорске, Беларусь. Получен-
ные от 20 инфицированных ВИЧ-1 лиц последовательности (индекс
А) представлены в сравнении с консенсусной последовательностью
Светлогорской вспышки (конс-Св). Консенсусы субтипа А (конс-А) и ва-
риантов ВВН-А с Украины и из России (конс-УР, ранее опубликованные
последовательности имели меньшую длину) приведены для сравнения.
По Лукашову с соавт. [Lukashov et al., 1998а]
нению с консенсусом этого очага (рис. 5.19) [Lukashov et al.,
1998а]. Такая — или даже близкая к такой — гомогенность попу-
ляции ВИЧ-1 не была описана ранее ни для одной человеческой
популяции. Для сравнения, средняя генетическая дистанция
между последовательностями гена env вариантов ВИЧ-1 субтипа
В среди ВВН в Нидерландах превышает 10% (или, в пересчете
на длину проанализированных последовательностей, — 25-30 за-
мен, рис. 5.17) [Lukashov et al., 1998b].
Такая высокая гомогенность вирусных популяций среди ВВН
в бывшем СССР, по-видимому, является результатом действия
нескольких факторов. Ее главной причиной является значитель-
но меньший возраст эпидемии, начавшейся на 10-15 лет позже
эпидемий в Западной Европе и США (разд. 5.3.7). Кроме того, из-
вестно, что особенности методов приготовления и использования
наркотиков, практикуемые ВВН на территории бывшего СССР»
5.5. Молекулярная эпидемиология 207
значительно отличаются от принятой среди ВВН в Западной
Европе практики. Можно предположить, что эти особенности
приводят к резкому снижению роли феномена бутылочного гор-
лышка при трансмиссии ВИЧ-1 (разд. 1.12), что, в свою очередь,
приводит к сохранению гомогенности ВИЧ-1 в популяции.
Быстрое распространение одних и тех же отличных от цир-
кулирующих в Европе вариантов ВИЧ-1 на территории стран
СНГ и Балтии позволяет заключить, что территория бывшего
Советского Союза остается в значительной степени единым
эпидемиологическим пространством.
Рассмотренные выше примеры демонстрируют эффектив-
ность и перспективность использования эволюционного анализа
при изучении эпидемиологии инфекционных заболеваний.
Глава 6
КОМПЬЮТЕРНЫЕ ПРОГРАММЫ
ДЛЯ ЭВОЛЮЦИОННОГО АНАЛИЗА
6.1. Типы компьютерных программ
В предыдущих главах мы обсудили цели, задачи и возможности
эволюционного анализа, теоретические и практические вопросы,
возникающие при проведении анализа генетической информа-
ции, и подходы к их решению. На практике эволюционный ана-
лиз проводится с помощью различных компьютерных программ.
К настоящему времени для решения различных вопросов эво-
люционного анализа в мире разработаны сотни компьютерных
программ. Далеко не все эти программы представляют широкий
практический интерес: среди них есть и устаревшие, и уступаю-
щие аналогичным, и недостаточно проверенные, и направленные
на решение весьма узких вопросов, редко возникающих на прак-
тике, и т.д. В подавляющем большинстве случаев для практиче-
ской работы достаточно трех-четырех компьютерных программ.
Как правило, практическая работа с генетической информа-
цией представляет собой получение новых генетических после-
довательностей (обычно — с помощью автоматических приборов
для определения нуклеотидных последовательностей), их редак-
тирование и хранение, сравнение с ранее полученными в мире
последовательностями, выравнивание и филогенетический ана-
лиз. Собственно, эта логика практической работы отражена
и в классификации существующих программ. В соответствии
с решаемыми ими задачами, компьютерные программы можно
классифицировать следующим образом:
— программы для редактирования генетической информа-
ции, поступающей с приборов для определения нуклеотид-
ных последовательностей;
— программы для хранения и редактирования последова-
тельностей;
— базы данных, хранящие международную генетическую ин-
формацию;
— программы для выравнивания последовательностей;
— программы для филогенетического анализа последователь-
ностей;
— программы для решения специальных задач.
6.1. Типы компьютерных программ 209
Как правило, компьютерные программы для анализа гене-
тической информации являются некоммерческими и доступны
для установки или пользования через Интернет бесплатно. Про-
тивные случаи оговорены при описании программ.
Поскольку первые программы в списке выше не предназначе-
ны для эволюционного анализа, они в нашей книге не рассмат-
риваются. Как правило, эти программы поставляются вместе
с приборами для определения нуклеотидных последовательно-
стей — например, Sequence Scanner Software компании ABI.
Кроме того, существуют и отдельные программы, как, например,
CodonCode Aligner (программа платная).
В этой главе мы приводим описание задач и возможностей
наиболее распространенных компьютерных программ для эво-
люционного анализа, руководства по пользованию программами
изложены в инструкциях.
Подчеркнем, что компьютерные программы дают исследо-
вателю широкие возможности для выбора параметров анали-
за: системы штрафов при выравнивании последовательностей,
той или иной эволюционной модели и используемых в ней
переменных при расчете эволюционных дистанций и т. д. По
умслчанию исследователя программа использует заложенные
в нее параметры. Соответственно исследователь должен знать
параметры анализа, используемые программой по умолчанию,
проверить, насколько они оптимальны для решения конкретной
задачи, и, при необходимости, изменить их. В лабораторной
работе экспериментальная ошибка приведет к отсутствию ре-
зультата, что будет очевидно. При эволюционном анализе некий
результат (скажем, филогенетическое дерево) будет получен да-
же при допущенных исследователем ошибках, но этот результат,
естественно, будет неправильным. Таким образом, эволюцион-
ный анализ налагает на исследователя особую ответственность
в плане проверки, доказательства правильности результатов
анализа. Отметим еще раз положение, выделенное нами в пре-
дисловии к этой книге:
Любая компьютерная программа проанализирует заложенные
в нее исследователем данные, используя выбранные исследователем
параметры, — и выдаст результаты анализа. Вопрос о том, насколь-
ко заложенные в программу данные и выбранные при анализе пара-
метры правильны, соответственно — насколько результаты анализа
отражают истинные эволюционные события, — является вопросом,
на которые должен ответить сам исследователь. Среди прочих
методов — ис помощью компьютерных программ.
14- 2238
210 Глава 6. Компьютерные программы для эволюционного анализа
Интернет-адреса
компьютерных программ и баз данных1
1. Программы для редактирования генетической информа-
ции, поступающей с приборов для определения нуклео-
тидных последовательностей:
— Sequence Scanner Software
https://products.appliedbiosystems.com/ab/ en/US/adirect/
ab?cmd=catNavigate2catID=600583tab= Detailinfo
— CodonCode Aligner
http: /1 www.codoncode .com
2. Программы для хранения и редактирования последова-
тельностей:
— BioEdit
http://www.mbio.ncsu.edu/ BioEdit/bioedit.html
— Artemis
http://www.sanger.ac.uk/Software /Artemis/
3. Международные базы генетических данных и используе-
мые в них программы:
— GenBank
http://www.ncbi.nlm.nih.gov/Genbank/index.html
— связь с PubMed
http://www.ncbi.nlm .nih.gov/sites/entrez?db=pubmed
— Entrez
http://www.ncbi.nlm.nih.gov/sites/entrez
— BLAST
http:I /blast.ncbi.nlm .nih .gov/ Blast.cgi
— Sequin
http://www.ncbi.nlm.nih.gov/Sequin/index.html
— Bankit
http: / /www.ncbi.nlm .nih.gov / Bankit/
— EMBL
http: /1 www.ebi.ac .uk/embl / index .html
— DDBJ
http://www.ddbj.nig.ac.jp/
— PIR
http: / / pir.georgetown .edu /
— SWISS-PROT
http:/ / www.expasy.org/ sprot/
Интернет-адреса приведены по состоянию на 1 марта 2009 г
6.1. Типы компьютерных программ 211
— HIV Sequence Database
http://www.hiv.lanl.gov/content/sequence/HIV/mainpage.html
— Ensemble
http:11 www.ensembl.org/index .htm 1
4. Программы для выравнивания последовательностей:
— ClustalW, ClustalX
http://www.clustal.org/
— Программой Clustal можно пользоваться в режиме
он-лайн на мощных компьютерах ряда сайтов Интер-
нета.
http://www.ebi.ac.uk/Tools/clustalw2/index.html
http:/ / www.ch.embnet.org/ software / ClustalW .htm 1
— T-Coffee
http:// tcoffee.vital-it.ch/cgi-bin/ TcoffeeI tcoffeecgi/ index .cgi
— MAFFT
http://ahgn.bmr.kyushu-u.ac.jp/mafft/software/
— PCMA
ftp://iole.swmed.edu/pub/PCMA/
— MUSCLE
http://www.drive5.com/muscle/
— MARNA
http://www.bioinf.uni-freiburg.de/Software/MARNA/
index.html
— SPEM
http://sparks.informatics.iupui.edu/index.php?pageLoc=
Downloads
— MANGO
http: /1 www.bioinfo.org.cn/ mango/
5. Программы для филогенетического анализа:
— PHYLIP
http://evolution.genetics.washington.edu/phylip.html
— MEGA
http:// www.megasoftware.net/
— PAML
http://abacus.gene.ucl.ac.uk/software/paml.html
— MrBayes
http://mrbayes.csit.fsu.edu/index.php
— PAUP*
http://paup.csit.fsu.edu/
— BEAST
http://beast.bio.ed.ac.uk/
212 Глава 6. Компьютерные программы для эволюционного анализа
— TREE-PUZZLE
http://www.tree-puzzle.de/
— SplitsTree
http://www.splitstree.org/
6. Другие программы:
— Sim Plot
http://sray.med.som.jhmi.edu/SCRoftware/
— RDP
http://darwin.uvigo.es/rdp/rdp.html
— GZ-GAMMA
http:/ / www.bio.psu.edu/People/ Faculty / Nei/ Lab/ gamma-
readme2.htm
— CodonDist запрос о получении программы через элек-
тронную почту
biolab-services@amc.uva.nl (subject=CodonDist)
7. Дополнительные справочные сайты Интернета по вопро-
сам молекулярной эволюции, списки программ, исполь-
зуемых в молекулярной эволюции и филогенетическом
анализе:
— список Фельзенштейна
http:/ / evolution.genetics. Washington .edu/ phy lip/ software.htm 1
— Bio Netbook
http://wwwl.pasteur.fr/recherche/BNB/bnb-en.html
— Bioinformatics Organization
http:/ / www.bioinformatics.org/
— Digital Taxonomy
http:/ / digitaltaxonomy.infobio.net/
— GeneBee
http://www.genebee.msu.su/genebee.html
Отметим также, что целым рядом компьютерных программ
можно пользоваться не только после их установки на персональ-
ный компьютер, но и на целом ряде сайтов Интернета, использу-
ющих возможности мощных быстродействующих компьютеров,
что, естественно, значительно сокращает время выполнения рас-
четов.
Описываемые в этой главе программы и базы данных при их
первом упоминании в тексте выделены жирным шрифтом, их
адреса в Интернете приведены в списке на с. 210-212. Как
правило, сайты программ содержат их детальную спецификацию
и руководства по пользованию программами.
6.2. Программы для хранения и редактирования 213
Описания программ и баз данных в нашей книге соответству-
ют их последним версиям по состоянию на 1 марта 2009 г. Ссыл-
ки в Интернете также действительны по состоянию на 1 марта
2009 г.
6.2. Программы для хранения
и редактирования последовательностей
Задачей программ этого класса является подготовка групп ге-
нетических последовательностей к их дальнейшему анализу,
проведение необходимых для этого манипуляций. Такие про-
граммы позволяют визуально анализировать последовательно-
сти, добавлять или убирать последовательности, редактировать
последовательности и их идентификаторы (название, уникаль-
ный международный номер, живая система, от которой они
получены, и т. д.), выбирать определенные участки последова-
тельностей для дальнейшего анализа, транслировать генетиче-
ские последовательности в аминокислотные, а также перефор-
матировать последовательности — сохранять их в виде файлов
различного формата.
Форматом в биоинформатике называют запись последователь-
ностей определенным образом, как правило, в виде текстово-
го файла, в котором название файла, начало и конец каж-
дой последовательности, идентификаторы последовательностей
и другая информация отмечены особым образом. Существова-
ние различных форматов (EMBL/Swissprot, FASTA, GenBank,
NEXUS, NBRF/PIR, PHYLIP и др.) в первую очередь связано
с историческими причинами, когда разработчики различных
программ генетического анализа независимо друг от друга созда-
вали форматы текстовых файлов, распознаваемые их програм-
мами. В качестве примеров различных форматов приведем одни
и те же последовательности в программе FASTA:
> seq_l
ATACGGGAAAACGCAATCCGCGAGA
ATGCC
> seq_2
ATACGTGAAAACGCAATCCGCGAGA
ATGAG
214 Глава 6. Компьютерные программы для эволюционного анализа
и в двух различных вариантах формата PHYLIP:
2 30
seq_l ATACGGGAAA ACGCAATCCG CGAGAATGCC
seq_2 ATACGTGAAA ACGCAATCCG CGAGAATGAG
или
2 30
seq_l ATACGGGAAA ACGCAATCCG
seq_2 ATACGTGAAA ACGCAATCCG
CGAGAATGCC
CGAGAATGAG
Как видно, в формате FASTA начало каждой новой последова-
тельности обозначается знаком «>», после чего следует название
последовательности, а сама последовательность начинается с но-
вой строки. В формате PHYLIP в первой строке указаны число
и длина последовательностей в файле.
Одной из наиболее распространенных и удобных программ
этого класса является программа BioEdit [Hall, 1999]. Кроме
вышеперечисленных манипуляций с последовательностями, про-
грамма BioEdit позволяет проводить ряд простых анализов, на-
пример, определять нуклеотидный и аминокислотный состав по-
следовательностей, рассчитывать консенсусные последовательно-
сти, искать потенциальные открытые рамки считывания и т. д-
Интерфейс программы BioEdit позволяет использовать и другие,
внешние, программы генетического анализа, например, програм-
му ClustalW (разд. 6.4) для выравнивания последовательностей
или PHYLIP (разд. 6.5) для филогенетического анализа. Есте-
ственно, эти внешние программы должны быть дополнительно
установлены на компьютер.
Кроме того, программа BioEdit предоставляет возможность
представлять последовательности в удобном для публикации
виде (опция graphic view).
Другой удобной для хранения, визуализации и простых ана-
лизов генетической информации программой является програм-
ма Artemis [Rutherford et al., 2000].
6.3. Международные базы генетических данных 215
6.3. Международные базы
генетических данных
Как правило, эволюционное исследование не основывается толь-
ко на экспериментальных данных (генетической информации),
полученных непосредственно в этом исследовании: важно срав-
нить вновь полученные генетические последовательности с по-
следовательностями (близкородственных форм жизни), получен-
ными ранее.
Для хранения и анализа полученной генетической информа-
ции существует целый ряд международных баз данных, или
генетических банков (в подавляющем большинстве случаев —
свободно доступных любому исследователю для работы даже без
какой-либо регистрации). Все эти базы данных можно разделить
на общие и специализированные. Основной задачей общих баз
данных является хранение и систематизация всех получаемых
в мире генетических последовательностей. Специализированные
базы данных занимаются анализом какой-либо конкретной про-
блемы: скажем, анализом данных о генетической изменчивости
вируса гепатита С (hepatitis С virus, HCV), иммунореактивных
эпитопах или мутациях, вызывающих устойчивость к анти-
ретровирусным препаратам у ВИЧ-1, использовании кодонов
в геномах различных форм жизни и т. д.
Изначально создание общих баз генетических данных велось
независимо друг от друга целым рядом генетических банков,
включая GenBank Национального центра биотехнологической
информации США (National Center for Biotechnology Information,
USA), EMBL-Bank Европейской молекулярно-биологической ла-
боратории (European Л/olecular Biology Laboratory, European
Bioinformatics Institute, UK), DDBJ Японии (DNA Data Bank
of Japan) для данных о нуклеотидных последовательностей,
PIR Национального биомедицинского исследовательского фон-
да США (Protein identification Resource, National Biomedical
Research Foundation, USA) и SWISS-PROT Университета Же-
невы и Института биоинформатики, Швейцария, для данных
об аминокислотных последовательностях. Сотрудники этих бан-
ков сами собирали опубликованную в международной науч-
ной литературе генетическую информацию. В настоящее время
развитие Интернета позволило объединить данные всех этих
общих генетических банков в единую систему, единый между-
народный банк генетической информации, а депонирование но-
вых генетических данных стало задачей самого исследователя:
216 Глава 6. Компьютерные программы для эволюционного анализа
при публикации научной статьи, сообщающей новые генетиче-
ские последовательности, требуется подтверждение того, что эта
информация уже направлена в международный генетический
банк.
При направлении новой последовательности в генетический
банк исследователь также сообщает дополнительную информа-
цию об этой последовательности: от какой формы жизни эта
последовательность получена, является ли она кодирующей или
нет и, если да, то какой ген она кодирует (если это известно),
каким методом эта последовательность получена, кем последо-
вательность получена и в какой статье планируется ее публи-
кация и т. д. Каждой новой генетической последовательности,
поступившей в генетический банк, присваивается уникальный
индекс, или номер (accession number), и эта последовательность
депонируется в банке с сопровождающей ее дополнительной
информацией (последовательность является аннотированной).
В настоящее время любой генетический банк не просто яв-
ляется местом хранения генетической информации, но и предо-
ставляет исследователям различные компьютерные программы,
позволяющие эту информацию анализировать. Наиболее гиб-
кую и эффективную (и потому — наиболее часто используемую)
платформу для доступа к этой генетической информации и ее
анализу предоставляет GenBank [Benson et al., 2008].
Этот банк предоставляет возможность найти и получить
генетическую информацию о какой-либо группе форм жизни
или генов (скажем, найти все последовательности, полученные
от пикорнавирусов, или все последовательности генов гемогло-
бина различных организмов), все последовательности, опублико-
ванные той или иной группой авторов, содержит данные о таксо-
номии и строении геномов различных форм жизни и т. д. Кроме
того, GenBank интегрирован с международной базой данных
научной литературы Pubmed, что позволяет найти публикации,
в которых та или иная генетическая информация (последо-
вательности с определенными уникальными индексами) была
получена или использована. Все эти возможности поиска необ-
ходимой исследователю информации предоставляются системой
Entrez [Geer and Sayers, 2003], являющейся интегрированной
составляющей GenBank.
Кроме этой функции хранения и обеспечения доступа к ан-
нотированной генетической информации, позволяющей най-
ти и получить (заранее) интересующую исследователя ге-
нетическую последовательность или группу последовательно-
6.3. Международные базы генетических данных 217
стей, GenBank предоставляет исследователю ряд возможно-
стей для анализа собственных, вновь полученных, генетиче-
ских последовательностей. Ключевым элементом этого анализа
является поиск в базе данных последовательностей, схожих
с анализируемой. Такая возможность предоставляется системой
сравнения последовательностей BLAST (basic Zocal alignment
search tool) [Altschul et al., 1990; Gish and States, 1993;
Madden et al., 1996; Altschul et al., 1997; Zhang and Madden,
1997; Zhang et al., 2000].
Программа BLAST позволяет исследователю загрузить инте-
ресующую его последовательность и сравнить ее со всеми после-
довательностями, полученными в мире ранее, найти самые по-
хожие последовательности (возможно, близкородственные). При
этом можно сравнивать как собственно нуклеотидные последо-
вательности, так и, возможно, кодируемые ими аминокислотные
последовательности. В базе данных будут найдены последова-
тельности, максимально схожие с анализируемой нуклеотидной
(и комплементарной ей, поскольку направление анализируемой
последовательности, 5'-3' или 3'-5', может быть и неизвест-
ным) и, возможно, кодируемой ею аминокислотной последова-
тельностью (учитывая все шесть возможных открытых рамок
считывания, трех в прямом и трех в обратном направлении).
В настоящее время алгоритмы программы BLAST позволяют
и учитывать возможные вставки и делеции при поиске схожих
последовательностей. Поскольку такой поиск требует выравни-
вания анализируемой последовательности со всеми полученными
ранее в мире, а выравнивание их по всей длине неоптимально,
программа BLAST использует алгоритмы локального выравни-
вания (разд. 2.3.2).
Естественно, что идентификация исследователем в базе дан-
ных последовательностей, схожих с анализируемой, важна, но
не является доказательством гомологичности (т.е. общего проис-
хождения, близкородственности, разд. 1.10) этих последователь-
ностей.
Для депонирования генетических последовательностей
в GenBank используется программы Sequin или Bankit [Ben-
son et al., 2008].
Описанный выше общий банк генетических данных GenBank
является примером первичной базы данных, получающей гене-
тическую информацию непосредственно от исследователей. В от-
личие от этого большинство специализированных баз данных
являются вторичными, получают интересующую их информа-
218 Глава 6. Компьютерные программы для эволюционного анализа
цию из первичных баз данных. Понятно, что специализирован-
ные банки не имеют универсального устройства: организация
банка, предоставляемые им возможности для генетического
анализа определяются конкретными целями и задачами этого
банка.
В качестве примера специализированного банка генетиче-
ских данных рассмотрим базу данных генетических последо-
вательностей ВИЧ HIV Sequence Database, поддерживаемую
Национальной лабораторией Лос-Аламоса (Los Alamos National
Laboratory), США.
Этот банк собирает из первичных банков (GenBank) депони-
рованные генетические последовательности ВИЧ и SIV и гено-
типирует их, относя к той или иной генетической группе и суб-
типу (разд. 5.3.7). Исследователю предоставляется возможность
получить интересующие его генетические последовательности
(полноразмерных геномов или какого-либо конкретного гена)
той или иной группы ВИЧ/SIV в уже выровненном виде. Банк
предоставляет информацию о строении геномов циркулирующих
рекомбинантных форм ВИЧ-1. С помощью специальных про-
грамм, исследователь может проверить, к какому именно суб-
типу ВИЧ-1 принадлежит полученная им последовательность,
не является ли она рекомбинантной формой и т. д.
Одной из важных и быстроразвивающихся вторичных баз
данных является база данных Ensemble, обеспечивающая ана-
лиз полноразмерных геномов высших организмов [Hubbard
et al., 2002; Clamp et al., 2003].
6.4. Программы для выравнивания
последовательностей
Одной из наиболее эффективных и часто используемых про-
грамм для (множественного) выравнивания последовательно-
стей является (упоминавшаяся ранее в разд. 2.4 и 6.2) про-
грамма Clustal в вариантах ClustalW и ClustalX [Higgins and
Sharp, 1988; Higgins and Sharp, 1989; Thompson et al., 1994;
Thompson et al., 1997; Jeanmougin et al., 1998; Chenna et al.,
2003; Larkin et al., 2007]. Эти два варианта используют один
и тот же алгоритм выравнивания, но вариант ClustalX име-
ет более удобный для пользователя графический интерфейс.
При обсуждении алгоритма и возможностей программы Clustal
в этом разделе, непринципиальные технические различия меж-
ду ее вариантами опущены. Программой можно пользоваться
6.4. Программы для выравнивания последовательностей 219
как на персональном компьютере, так и на ряде сайтов Ин-
тернета, используя быстродействующие компьютеры. Примеры
таких сайтов, их интернет-адреса, также приведены в списке на
с. 210-212.
Алгоритм программы Clustal (и многих других программ
выравнивания) основан на уже упомянутой в разд. 2.4 идее
прогрессивного множественного выравнивания, состоящей в сле-
дующем. На первом этапе производится попарное выравнива-
ние всех анализируемых последовательностей, т. е. индивиду-
альное выравнивание первой последовательности — со второй,
затем — с третьей и т. д. Программа позволяет проводить та-
кое выравнивание как методами динамического программиро-
вания (медленно-аккуратно, разд. 2.3.2), так и методами слов
(быстро-приблизительно, разд. 2.3.3). Для каждой пары после-
довательностей определяется генетическая дистанция между
последовательностями, в простейшем случае — процент несов-
падающих нуклеотидов или аминокислот. Можно использовать
и более комплексные модели. Так, при установлении амино-
кислотных дистанций можно использовать и матрицы вероят-
ностей аминокислотных замещений классов РАМ и BLOSUM
(разд. 3.4). При этом программа автоматически использует раз-
личные матрицы одного выбранного исследователем класса
при анализе более схожих (с меньшими эволюционными ди-
станциями) и менее схожих между собой последовательно-
стей.
Полученная матрица попарных генетических дистанций меж-
ду последовательностями используется для построения филоге-
нетического дерева методом присоединения соседей (разд. 4.2.6).
На основании такого анализа выявляются группы более схожих
между собой последовательностей. В дальнейшем программа
выполняет выравнивание последовательностей в каждой та-
кой группе, после чего — выравнивание групп между собой.
Важным элементом этого этапа является взвешивание групп
последовательностей: группа из многих схожих между собой
последовательностей будет учитываться как немногочисленная
группа, в противном случае эта группа в значительной степени
определяла бы итоговое выравнивание всех остальных последо-
вательностей.
Кроме этих ключевых элементов, алгоритм выравнивания
программы Clustal включает в себя и ряд дополнительных
элементов, скажем, штрафы за пробелы рядом с глицином
снижаются, поскольку экспериментально показано, что глицин
220 Глава 6. Компьютерные программы для эволюционного анализа
часто встречается рядом с пробелами [Pascarella and Argos,
1992].
Кроме общей задачи выравнивания всех последовательностей
анализируемой группы, программа Clustal предоставляет воз-
можность добавить одну или несколько новых последовательно-
стей к уже существующей выровненной группе последователь-
ностей, а также выровнять две заранее выровненные группы
последовательностей между собой.
В целом программа Clustal, безусловно, является и надежной,
и одной из наиболее быстродействующих программ для вырав-
нивания последовательностей. При выравнивании кодирующих
нуклеотидных последовательностей более быстрое и надежное
выравнивание будет проведено после трансляции этих после-
довательностей в аминокислотные. Отметим, однако, что даже
при использовании этой быстродействующей программы на со-
временном персональном компьютере выравнивание, скажем,
100 последовательностей длиной в 10000 нуклеотидов каждая
может занимать несколько дней компьютерного времени. Быст-
родействие программы Clustal определяется тем, что ее алго-
ритм является иерархическим, прогрессивным: завершив некий
шаг, этап при решении задачи множественного выравнивания
(скажем, выровняв попарно первую и вторую последователь-
ности), программа, в общем случае, больше к этому этапу
не возвращается.
Однако понятно, что данные следующих шагов выравнивания
могут содержать информацию, важную для предыдущего шага
анализа. Скажем, анализ третьей последовательности, ее по-
парное выравнивание с первой и второй последовательностями,
может содержать информацию, важную для уже завершенного
выравнивания первой и второй последовательностей между со-
бой.
Кроме того, как отмечалось в разд. 2.4, понятно, что биологи-
чески оптимальное выравнивание (установление гомологичных
позиций и участков последовательностей) должно учитывать
и структуру гена, и возможное наличие чужеродного гене-
тического материала (скажем, ретроэлементов) в выравнивае-
мых последовательностях, и ряд других возможных эволюци-
онных событий. Некоторые перспективные подходы к реше-
нию этих (и других) проблем, возникающих при выравнива-
нии последовательностей, намечены в ряде других программ
для выравнивания: MUSCLE (multiple sequence comparison by
/og-c xpectation) [Edgar, 2004] — по мнению ряда исследователей,
6.5. Программы для филогенетического анализа 221
эта программа превосходит Clustal, Т-Coffee (consistency based
objective /unction for alignment evaluation) [Notredame et al.,
2000; Notredame and Suhre, 2004; Poirot et al., 2004;
Wallace et al., 2006; Moretti et al., 2007; Moretti et al., 2008;
Wilm et al., 2008], MAFFT (multiple Alignment based on /ast
Fourier transform) [Katoh et al., 2002], PCMA (profile consistency
multiple alignment) [Pei et al., 2003], MARNA (rz/ultiple alignment
of RNA) [Siebert and Backofen, 2005], SPEM (sequence and
secondary structure profiles enhanced multiple alignment) [Zhou
and Zhou, 2005], MANGO (multiple alignment with n gapped
oligos) [Zhang et al., 2007] и др. [Riaz et al., 2005; Sandelin,
2005; Simossis et al., 2005; Wallace et al., 2005; Wang and
Lefkowitz, 2005; Armougom et al., 2006; Zhang and Kahveci, 2007;
Subramanian et al., 2008].
При безусловной перспективности целого ряда этих программ
и предлагаемых в них алгоритмов выравнивания возможности
и характеристики этих программ на сегодняшний день изучены
недостаточно, а по своему быстродействию многие из них усту-
пают (в ряде случаев — значительно) программе Clustal.
6.5. Программы для филогенетического анализа
В отличие от рассмотренных выше программ для редактиро-
вания и выравнивания последовательностей среди программ
для филогенетического анализа нет одной доминирующей про-
граммы. В значительной степени это связано с разнообрази-
ем имеющихся методов построения филогенетических деревьев
(гл. 4), каждый из которых имеет свои преимущества и недо-
статки (разд. 4.6).
На протяжении многих лет наиболее распространенной
программой для филогенетического анализа была программа
PHYLIP (phylogeny inference package) [Felsenstein, 1990]. Трудно
переоценить значение этой программы, вклад ее автора Фель-
зенштейна в развитие идей и популяризацию методов филогене-
тического анализа. Постоянно совершенствуемая и расширяемая
автором программа и сегодня, спустя почти 30 лет после вы-
хода его первой версии, является математически современной,
надежной, гибкой программой, предоставляющей возможности
для решения широкого спектра вопросов филогенетического
анализа. Ее интерфейс, однако, в известной степени усложняет
пользование программой (Фельзенштейн: «At this stage we do not
have a mouse-windows interface for PHYLIP»). PHYLIP является
222 Глава 6. Компьютерные программы для эволюционного анализа
не единой программой, но пакетом из нескольких десятков
взаимосвязанных подпрограмм, модулей, каждый из которых
направлен на решение конкретной подзадачи. Если, скажем, за-
дачей исследователя является построение филогенетического де-
рева для некой группы (предварительно выровненных) нуклео-
тидных последовательностей с помощью метода NJ (разд. 4.2.6),
то на первом этапе следует рассчитать эволюционные дистан-
ции между анализируемыми последовательностями с помощью
модуля DNADIST. Полученную матрицу попарных дистанций
следует сохранить в виде файла, после чего этот файл следует
загрузить в следующий модуль программы, NEIGBOR, устанав-
ливающей филогенетические отношения с помощью метода NJ.
Наконец, файл, сохраненный этим модулем, следует загрузить
в следующий, графический, модуль программы, который позво-
ляет визуализировать построенное дерево. Если исследователю
необходимо получить дерево с корнем, то следует использо-
вать модуль DRAWGRAM, а для дерева без корня — модуль
DRAWTREE. Для манипуляций с этим деревом (изменением
положения его корня, изменением индексов анализируемых по-
следовательностей и т.д.) следует использовать модуль RETREE.
Если исследователь хочет оценить достоверность этого дерева
при помощи, скажем, бутстрэп-анализа (разд. 4.4), то необходимо
использовать модули SEQBOOT и CONSENSE и т. д. Безусловно,
необходимость сохранения всех промежуточных файлов услож-
няет пользование программой.
Важнейшей характеристикой пакета PHYLIP является
предоставляемая им возможность проведения филогенетиче-
ского анализа с использованием методов дискретных призна-
ков (разд. 4.3), МР (разд. 4.3.2) и ML (разд. 4.3.3), в теоре-
тическую разработку которых автор программы внес весо-
мый вклад [Felsenstein, 1973; Felsenstein, 1981; Cavender and
Felsenstein, 1987; Felsenstein, 1988; Kuhner and Felsenstein, 1994;
Felsenstein, 1996; Felsenstein, 2001; Felsenstein, 2004; Felsenstein,
2005; Felsenstein, 2006; Felsenstein, 2008].
Надежной, удобной в пользовании программой для филоге-
нетического анализа, рекомендуемой, в частности, для начи-
нающего исследователя, является программа MEGA (molecular
evolutionary genetics Analysis) [Kumar et al., 1993; Kumar et al.,
1994; Kumar et al., 2001; Kumar et al., 2004; Tamura et al., 2007;
Kumar et al., 2008]. Наиболее сильной стороной этой программы
является хорошо проработанная в ней возможность исполь-
зования дистанционных методов построения филогенетических
6.5. Программы для филогенетического анализа 223
деревьев (разд. 4.2), в особенности — метода NJ (разд. 4.2.6) в со-
четании с бутстрэп-анализом (разд. 4.4). При этом исследователю
предоставляется широкий выбор между использованием того
или иного метода установления эволюционных дистанций, от са-
мых простых p-дистанций (разд. 3.1) до Г-дистанций (разд. 3.2.5).
Особо отметим предоставляемые программой возможности раз-
дельного анализа синонимичных и несинонимичных дистанций
(разд. 1.6.2 и 3.3). Программа проста и удобна в пользовании,
позволяет, например, исключить те или иные последовательно-
сти из анализа, объединить ряд последовательностей в группы
и рассчитать средние дистанции внутри и между группами, про-
вести раздельный анализ тех или иных генетических участков
анализируемых последовательностей и т.д. Кроме того, графиче-
ский интерфейс программы предоставляет широкие возможно-
сти для манипуляций с полученным филогенетическим деревом
при его подготовке к презентации или публикации: изменения
размера шрифта и шкалы дистанций, выделения тех или иных
эволюционных линий и т. д.
Значительно более мощный математический аппарат исполь-
зован в целом ряде других филогенетических программ.
Программа PAML (phylogenetic analysis by znaximum
likelihood) [Yang, 1997; Yang, 2007] не позиционируется ее
автором как программа для собственно построения филогене-
тических деревьев (Янг: «PAML is not good for tree making»),
однако безусловно относится к числу важных в филогенетиче-
ском анализе программ.
В отличие от программы MEGA эта программа не просто
предоставляет самому исследователю возможность выбрать ту
или иную эволюционную модель (гл.З) при проведении фило-
генетического анализа, но и дает возможность оценить соот-
ветствие эволюции анализируемой группы последовательностей
той или иной эволюционной модели и рассчитать параметры
этой модели. Такая оценка может быть проведена при помощи
сравнительного статистического анализа правдоподобия дере-
вьев (разд. 4.3.3) при использовании той или иной эволюционной
модели — проводимого, в частности, с помощью теста отноше-
ния правдоподобий, LRT (разд. 5.3.3). Если результаты исполь-
зования более сложной эволюционной модели не отличаются
достоверно от результатов более простой эволюционной моде-
ли, то следует выбрать и использовать более простую модель
(разд. 3.2.6).
224 Глава 6. Компьютерные программы для эволюционного анализа
Кроме выбора наиболее оптимальной эволюционной модели,
сравнение правдоподобия различных сценариев эволюции мо-
жет быть использовано и для анализа наличия молекулярных
часов в эволюции анализируемой группы последовательностей,
и для расчета генетической последовательности наиболее ве-
роятного последнего общего предка анализируемых последова-
тельностей (разд. 5.3.3), и для выявления эволюционных линий
в анализируемых последовательностях, конкретных позиций
этих последовательностей, которые подвергаются положитель-
ному отбору (разд. 1.11 и 3.3), и т. д.
Подходы к построению деревьев на основе байесовской стати-
стики, скрытых марковских моделей и симуляций Монте-Карло
использованы в программе MrBayes [Huelsenbeck and Ronquist,
2001; Ronquist and Huelsenbeck, 2003].
Отметим, что подобные анализы, математические расчеты,
чрезвычайно трудоемки в плане необходимого компьютерного
времени и, как правило, требуют использования компьютеров,
превосходящих по своим возможностям персональные компью-
теры даже последнего поколения. Для подавляющего большин-
ства простых филогенетических задач, такие анализы не явля-
ются необходимыми.
Одной из наиболее полных филогенетических программ,
в плане возможности использования различных филогенетиче-
ских подходов, является программа PAUP* (phylogenetic analysis
using parsimony, сокращенное название программы произносится
как «поп-стар», программа платная, включая даже ее бета-
версию) [Rogers and Swafford, 1998; Swafford, 2002; Wilgenbusch
and Swofford, 2003]. Ссылка на конкретный филогенетический
метод в названии программы отражает историю ее создания: со-
временная версия этой программы позволяет проводить гораздо
более широкий спектр филогенетических анализов, используя
как дистанционные методы построения филогенетических дере-
вьев (разд. 4.2), так и различные методы анализа дискретных
признаков (разд. 4.3).
Отметим также филогенетическую программу BEAST (Bay-
esian evolutionary analysis by sampling trees) [Drummond et al-,
2002; Drummond et al., 2005; Drummond et al., 2006;
Shapiro et al., 2006; Drummond and Rambaut, 2007], одними
из основных задач которой являются анализ наличия молеку-
лярных часов в эволюции анализируемой группы последователь-
ностей и датирование эволюционных событий.
6.6. Другие программы 225
Кроме того, упомянутый нами в разд. 4.6 метод головоло-
мок квартетов использован в компьютерной программе TREE-
PUZZLE [Strimmer and von Haeseler, 1996; Strimmer et al.,
1997; Schmidt et al., 2002; von Haeseler and Strimmer, 2004],
а метод отображения эволюционных отношений в виде филоге-
нетических сетей — в программе SplitsTree [Huson, 1998].
6.6. Другие программы
Как уже отмечалось в начале этой главы, в мире созданы сотни
компьютерных программ, направленных на решение различных
вопросов молекулярной эволюции и филогенетического анализа,
и существуют десятки баз генетических данных и серверов, поз-
воляющих проводить филогенетические исследования. Наиболее
важные из них, с точки зрения их полноты и надежности, опи-
саны в нашей книге. При поиске других программ и баз данных
рекомендуется пользоваться имеющимися в Интернете списками
программ и системами их поиска. Ряд этих списков приведен
на с. 210. Так, только список Фельзенштейна содержит ссылки
на Солее чем 380 филогенетических программ и интернет-адреса
более чем 50 бесплатных серверов, позволяющих проводить
различные виды компьютерного эволюционного анализа.
В этом разделе отметим несколько программ, направленных
на решение специальных, более узких задач филогенетического
анализа, обсужденных нами в книге.
Оценка распределения частот замен в различных позициях
анализируемых последовательностей, установление параметра а,
необходимого при расчете Г-дистанций (разд. 3.2.5), может быть
проведена, в частности, при помощи программы GZ-GAMMA
[Gu and Zhang, 1997].
Рекомбинационный анализ (разд. 5.1) может быть вы-
полнен при помощи программ SimPlot (similarity plot,
рис. 5.3) [Lole et al., 1999] или RDP (recombination detection
program) [Martin and Rybicki, 2000].
Данные по использованию кодонов (разд. 5.2.2) могут быть
трансформированы в генетические дистанции при помощи про-
граммы CodonDist [nan Hemert et al., 2007].
15
2238
книги
ДЛЯ ДОПОЛНИТЕЛЬНОГО ЧТЕНИЯ
Филогенетика является одной из ключевых составляющих мо-
лекулярной эволюции как науки, биоинформатики в целом. Как
и практически любое современное направление науки, филоге-
нетика использует данные различных наук, включая общую био-
логию и математику, генетику и органическую химию. Другим
ключевым направлением биоинформатики является сравнитель-
ная геномика, изучающая взаимоотношения между структурами
и функциями макромолекул.
Являясь самостоятельными разделами биоинформатики, со
своими собственными задачами и методами, филогенетика
и сравнительная геномика разделяют один и тот же объект
исследования: макромолекулы, их изменения в ходе эволюции.
Филогенетика использует данные сравнительной геномики —
и сравнительная геномика во многих случаях опирается на ре-
зультаты филогенетических исследований.
В заключение нашей книги, приведем список книг, реко-
мендуемых автором для дополнительного чтения по вопросам
биоинформатики. Ряд этих книг переиздавался, и их выходные
данные могут варьировать. Выбор этих книг и их краткая
аннотация:
1. Li, W.H. (1997). Molecular evolution. Sunderland, USA: Sinauer
Associates.
Одна из наиболее полных на сегодняшний день книг по молекуляр-
ной эволюции.
2. Felsenstein, J. (2004). Inferring phylogenies. Sunderland, USA:
Sinauer Associates.
Книга детально описывает математические аспекты филогенетиче-
ского анализа, подходы к решению проблем. Книга полезна скорее
математикам, чем биологам.
3. Hall, B.G. (2007). Phylogenetic trees made easy: a how-to manual-
Sunderland, USA: Sinauer Associates.
4. Salemi, M. and Vandamme, A. M. (Editors) (2004). The phylogenetic
handbook: a practical approach to DNA and protein phylogeny-
Cambridge, UK: Cambridge University Press.
Книги для дополнительного чтения 227
Отличительными особенностями этих книг является детальное опи-
сание практических вопросов работы с различными филогенетиче-
скими программами.
5. Вейр, Б. (1995). Анализ генетических данных. М.: Мир.
Книга посвящена вопросам анализа популяционных генетических
данных.
6. Haubold, В. and Wiehe, Т. (2007). Introduction to computational
biology: an evolutionary approach. Basel, Switzerland: Birkhauser.
Книга описывает ряд вопросов популяционной генетики, сравни-
тельной геномики и филогенетики, с акцентом на математические
аспекты решения проблем.
7. Claverie, J.M. and Notredame, С. (2003). Bioinformatics for dummies.
New York, USA: Willey Publishing.
Книга для начального чтения по вопросам биоинформатики.
8. Galperin, M.Y and Koonin, E.V. (Editors) (2003). Frontiers in
computational genomics (Functional genomics series). Norfolk, UK:
Caister Academic Press.
9. Koonin, E.V. and Galperin, M.Y. (2002). Sequence — evolution —
function: computational approaches in comparative genomics. Kluwer
Academic Publishers.
10. Mushegian, A. R. (2007). Foundations of comparative genomics.
Burlington, USA: Elsevier Academic Press.
Книги посвящены различным вопросам сравнительной геномики.
ЛИТЕРАТУРА
1. Вьюгин, В.В., Гельфанд, М.С., Любецкий, В.А. (2002). Согласо-
вание деревьев: реконструкция эволюции видов по филогенетиче-
ским деревьям генов. Молекулярная биология, 36, 807-816.
2. Вьюгин, В.В., Гельфанд, М.С., Любецкий, В.А. (2005). Идентифи-
кация горизонтально перенесенных генов на основе филогенетиче-
ских данных. Молекулярная биология, 37, 673-687.
3. Гельфанд, М.С. (2005). Апология биоинформатики. Биофизика 50,
752-766.
4. Грант, В. (1980). Эволюция организмов. М.: Мир.
5. Иорданский, Н.Н. (2001). Эволюция жизни. М.: Академия.
6. Кимура, М. (1985). Молекулярная эволюция: теория нейтрально-
сти. М.: Мир.
7. Ратнер, В. А., Жарких, А. А., Колчанов, Н.А., Родин, С.Н., Соло-
вьев, В. В., Шамин, В. В. (1985). Проблемы теории молекулярной
эволюции. Новосибирск: Наука.
8. Abebe, A., Lukashov, V. V., Pollakis, G., Kliphuis, A., Fontanet, A.L.,
Goudsmit, J., and de Wit, T.F. (2001a). Timing of the HIV-1 subtype
C epidemic in Ethiopia based on early virus strains and subsequent
virus diversification. AIDS 15, 1555-1561.
9. Abebe, A., Lukashov, V. V., Rinke De Wit, T.F., Fisseha, B., Tegbaru,
B., Kliphuis, A., Tesfaye, G., Negassa, H., Fontanet, A.L., Goudsmit,
J., and Pollakis, G. (2001b). Timing of the introduction into Ethiopia of
subcluster C’ of HIV type 1 subtype C. AIDS Res Hum Retroviruses
17, 657-661.
10. Albert, J., Wahlberg, J., Leitner, T., Escanilla, D., and Uhlen, M.
(1994). Analysis of a rape case by direct sequencing of the human
immunodeficiency virus type 1 pol and gag genes. J Virol 68, 5918-
5924.
11. Altschul, S.F. (1991). Amino acid substitution matrices from an
information theoretic perspective. J Mol Biol 219, 555-565.
12. Altschul, S.F., Gish, W., Miller, W., Myers, E.W., and Lipman, D. J-
(1990). Basic local alignment search tool. J Mol Biol 215, 403-410.
13. Altschul, S.F., Madden, T.L., Schaffer, A.A., Zhang, J., Zhang, Z.,
Miller, W., and Lipman, D. J. (1997). Gapped BLAST and PSI-BLAST:
a new generation of protein database search programs. Nucleic Acids
Res 25, 3389-3402.
Литература 229
14. Argos, Р., Rossman, M.G., Grau, U.M., Zuber, H., Frank, G., and
Tratschin, J. D. (1979). Thermal stability and protein structure.
Biochem 18, 5698-5703.
15. Armougom, F., Poirot, O., Moretti, S„ Higgins, D.G., Bucher,
P., Keduas, V., and Notredame, C. (2006). APDB: a web server
to evaluate the accuracy of sequence alignments using structural
information. Bioinformatics 22, 2439-2440.
16. Arvestad, L., Berglund, A.C., Lagergren, J., and Sennblad, B. (2003).
Bayesian gene/species tree reconciliation and orthology analysis using
MCMC. Bioinformatics 19 Suppl 1, i7-15.
17. Atchley, W.R. and Fitch, W.M. (1991). Gene trees and the origins of
inbred strains of mice. Science 254, 554-558.
18. Bebenek, K., Abbotts, J., Roberts, J. D., Wilson, S.N., and Kunkel,
T.A. (1989). Specificity and mechanism of error-prone replication by
human immunodeficiency virus-1 reverse transcriptase. J Biol Chem
264,28, 16948-16956.
19. Beiko, R.G. and Hamilton, N. (2006). Phylogenetic identification of
lateral genetic transfer events. BMC Evol Biol 6, 15.
20. Benirschke, K., Malouf, N., Low, R.J., and Heck, H. (1965).
Chromosome complement: differences between Equus caballus and
Equus przevalskii, poliakoff. Science 148, 382-383.
21. Bennetzen, J. L. and Hall, B. D. (1982). Codon selection in yeast. J
Biol Chem 257, 3026-3031.
22. Benson, D.A., Karsch-Mizrachi, I., Lipman, D.J., Ostell, J., and
Wheeler, D.L. (2008). GenBank. Nucleic Acids Res 36, D25-D30.
23. Bergsten, J. (2005). A review of long-branch attraction. Cladistics 21,
163-193.
24. Berkhout, B., Das, A.T., and Beerens, N. (2001). HIV-1 RNA editing,
hypermutation, and error-prone reverse transcription. Science 292, 7.
25. Berkhout, B., Grigoriev, A., Bakker, M., and Lukashov, V.V. (2002).
Codon and amino acid usage in retroviral genomes is consistent with
virus-specific nucleotide pressure. AIDS Res Hum Retroviruses 18,
133-141.
26. Berkhout, B. and van Hemert, F.J. (1994). The unusual nucleotide
content of the HIV RNA genome results in a biased amino acid
composition of HIV proteins. Nucleic Acids Res 22, 1705-1711.
27. Bernardi, G. (1993). The vertebrate genome: isochores and evolution.
Mol Biol Evol 10, 186-204.
28. Bernardi, G. (2000a). Isochores and the evolutionary genomics of
vertebrates. Gene 241, 3-17.
29. Bernardi, G. (2000b). The compositional evolution of vertebrate
genomes. Gene 259, 31-43.
230 Литература
30. Bernardi, G., Olofsson, В., Filipski, J., Zerial, M., Salinas, J., Cuny,
G., Meunier-Rotival, M., and Rodier, F. (1985). The mosaic genome
of warm-blooded vertebrates. Science 228, 953-958.
31. Beutler, E., Gelbart, T., Han, J.H., Koziol, J. A., and Beutler, B.
(1989). Evolution of the genome and the genetic code: selection at
the dinucleotide level by methylation and polyribonucleotide cleavage.
Proc Natl Acad Sci USA 86, 192-196.
32. Bird, A. P. (1986). CpG-rich islands and the function of DNA
methylation. Nature 321, 209-213.
33. Bobkov, A., Cheingsong-Popov, R., Garaev, M., Rzhaninova, A.,
Kaleebu, P., Beddows, S., Bachman, M.H., Mullins, J. I., Louwagie,
J., Janssens, W., van der Groen, G., McCutchen, F., and Weber, J.
(1994a). Identification of an env G subtype and heterogeneity of HIV-1
strains in the Russian Federation and Belarus. AIDS 8, 1649-1655.
34. Bobkov, A., Cheingsong-Popov, R., Selimova, L., Ladnaya, N.,
Kazennova, E., Kravchenko, A., Fedotov, E., Saukhat, S., Zverev,
S., Pokrovsky, V., and Weber, J. (1997). An HIV type 1 epidemic
among injecting drug users in the former Soviet Union caused by
a homogeneous subtype A strain. AIDS Res Hum Retroviruses 13,
1195-1201.
35. Bobkov, A., Garaev, M.M., Rzhaninova, A., Kaleebu, P., Pitman,
R., Weber, J.N., and Cheingsong-Popov, R. (1994b). Molecular
epidemiology of HIV-1 in the former Soviet Union: analysis of env
V3 sequences and their correlation with epidemiologic data. AIDS 8,
619-624.
36. Bobkov, A., Kazennova, E., Selimova, L., Bobkova, M., Khanina, T.,
Ladnaya, N., Kravchenko, A., Pokrovsky, V., Cheingsong-Popov, R.,
and Weber, J. (1998). A sudden epidemic of HIV type 1 among
injecting drug users in the former Soviet Union: identification of
subtype A, subtype B, and novel gagA/envB recombinants. AIDS Res
Hum Retroviruses 14, 669-676.
37. Bobkov, A. F., Lukashov, V.V., Goudsmit, J., and Weber, J.N. (2000).
Silent mutation in the V3 region characteristic of HIV type 1 env
subtype В strains from injecting drug users in the former Soviet
Union. AIDS Res Hum Retroviruses 16, 291-294.
38. Boudraa, M. and Perrin, P. (1987). CpG and TpA frequencies in the
plant system. Nucleic Acids Res 15, 5729-5737.
39. Bronson, E.C. and Anderson, J.N. (1994). Nucleotide composition
as a driving force in the evolution of retroviruses. J Mol Evol 38,
506-532.
40. Brown, W.M., George, M., Jr., and Wilson, A.C. (1979). Rapid
evolution of animal mitochondrial DNA. Proc Natl Acad Sci USA
76, 1967-1971.
Литература 231
41. Buneman, Р. (1971). The recovery of trees from measures of
dissimilarity. In: Mathematics in archeological and historical
sciences, pp. 387-395. Edited by Hodson, F.R., Kendall, D.G., and
Taute, P. Edinburgh, UK: Edinburgh Univ. Press.
42. Buneman, P. (1974). A note on the metric properties of trees. J Comb
Theory 17, 48-50.
43. Buonagurio, D.A., Nakada, S., Parvin, J. D., Krystal, M., Palese, P.,
and Fitch, W.M. (1986). Evolution of human influenza A viruses
over 50 years: rapid, uniform rate of change in NS gene. Science 232,
980-982.
44. Burge, C., Campbell, A.M., and Karlin, S. (1992). Over- and under-
representation of short oligonucleotides in DNA sequences. Proc Natl
Acad Sei USA 89, 1358-1362.
45. Cann, R.L., Stoneking, M., and Wilson, A.C. (1987). Mitochondrial
DNA and human evolution. Nature 325, 31-36.
46. Cardon, L.R., Burge, C., Clayton, D.A., and Karlin, S. (1994).
Pervasive CpG suppression in animal mitochondrial genomes. Proc
Natl Acad Sci USA 91, 3799-3803.
47. Cavalli-Sforza, L.L. and Edwards, A. W. (1967). Phylogenetic analysis.
Models and estimation procedures. Am J Hum Genet 19, 233-257.
48. Cavender, J. A. (1978). Taxonomy with confidence. Math Biosci 40,
271-280.
49. Cavender, J. A. (1989). Mechanized derivation of linear invariants.
Mol Biol Evol 6, 301-316.
50. Cavender, J.A. and Felsenstein, J. (1987). Invariants of phylogenies:
simple case with discrete states. J Classif 4, 57-71.
51. Chenna, R., Sugawara, H., Koike, T., Lopez, R., Gibson, T. J., Higgins,
D.G., and Thompson, J.D. (2003). Multiple sequence alignment with
the Clustal series of programs. Nucleic Acids Res 31, 3497-3500.
52. Clamp, M., Andrews, D., Barker, D., Bevan, P., Cameron, G., Chen,
Y., Clark, L., Cox, T., Cuff, J., Curwen, V., Down, T., Durbin, R.,
Eyras, E., Gilbert, J., Hammond, M., Hubbard, T., Kasprzyk, A.,
Keefe, D., Lehvaslaiho, H., Iyer, V., Melsopp, C., Mongin, E., Pettett,
R., Potter, S., Rust, A., Schmidt, E., Searle, S., Slater, G., Smith,
J., Spooner, W., Stabenau, A., Stalker, J., Stupka, E., Ureta-Vidal,
A., Vastrik, I., and Birney, E. (2003). Ensembl 2002: accommodating
comparative genomics. Nucleic Acids Res 31, 38-42.
53. Comeron, J. M. (1995). A method for estimating the numbers of
synonymous and nonsynonymous substitutions per site. J Mol Evol
41, 1152-1159.
54. Comeron, J.M. and Aguade, M. (1998). An evaluation of measures of
synonymous codon usage bias. J Mol Evol 47, 268—274.
55. Cooper, D.N. (1983). Eukaryotic DNA methylation. Hum Genet 64,
315-333.
132 Литература
56. Cooper, D.N., Taggart, М.Н., and Bird, A.P. (1983). Unmethylated
domains in vertebrate DNA. Nucleic Acids Res 11, 647 658.
57. Cornelissen, M., Mulder Kainpinga, G., Veenstra, J., Zorgdrager,
F., Kuiken, C., Hartman, S., Dekker, J., van der Hoek, L., Sol,
C., Coutinho, R., and Goudsmit, J. (1995). Syncitium-inducing
(SI) phenotype suppression at seroconversion after intramuscular
inoculation of a non-syncytium-inducing/SI phenotypically mixed
human immunodeficiency virus population. J Virol 69, 1810-1818.
58. Cowe, E. and Sharp, P. M. (1991). Molecular evolution of
bacteriophages: discrete patterns of codon usage in T4 genes are
related to the time of gene expression. J Mol Evol 33, 13-22.
59. Crick, F.H. (1958). On protein synthesis. Symp Soc Exp Biol 12,
138-163.
60. Cross, S.H. and Bird, A.P. (1995). CpG islands and genes. Curr Opin
Genet Dev 5, 309-314.
61. Czelusniak, J., Goodman, M., Hewett-Emmett, D., Weiss, M.L.,
Venta, P.J., and Tashian, R. E. (1982). Phylogenetic origins and
adaptive evolution of avian and mammalian haemoglobin genes.
Nature 298, 297 300.
62. D’Erchia, A. M., Gissi, C., Pesole, G., Saccone, C., and Arnason, U.
(1996). The guinea-pig is not a rodent. Nature 381, 597- 600.
63. Darwin, C. (1871). The Descent of Man and Selection in Relation to
Sex. New York, USA: Appleton.
64. Dayhoff, M.O. (1972). Atlas of Protein Sequence and Structure.
Washington, USA: National Biomedical Research Foundation.
65. Dayhoff, M.O. (1978). Atlas of Protein Sequence and Structure.
Washington, USA: National Biomedical Research Foundation.
66. de A. Zanotto, P.M., Gould, E.A., Gao, G.F., Harvey, P.H., and
Holmes, E.C. (1996). Population dynamics of flaviviruses revealed
by molecular phylogenies. Proc Natl Acad Sci USA 93, 548-553.
67. De Amicis, F. and Marchetti, S. (2000). Intercodon dinucleotides
affect codon choice in plant genes. Nucleic Acids Res 28, 3339 3345.
68. Deininger, P. L. and Batzer, M.A. (2002). Mammalian retroeleinents.
Genome Res 12, 1455-1465.
69. Deininger, P. L., Moran, J. V., Batzer, M.A., and Kazazian, H.H., Jr.
(2003). Mobile elements and mammalian genome evolution. Curr Opin
Genet Dev 13, 651 658.
70. Di Rienzo, A. and Wilson, A.C. (1991). Branching pattern in the
evolutionary tree for human mitochondrial DNA. Proc Natl Acad
Sci USA 88, 1597-1601.
71. Diallo, A. B., Makarenkov, V., and Blanchette, M. (2007). Exact and
heuristic algorithms for the Indel Maximum Likelihood Problem. J
Comput Biol 14, 446-461.
Литература 233
72. Doolittle, W.F. (1999). Phylogenetic classification and the universal
tree. Science 284, 2124-2129.
73. Dopazo, J., Dress, A., and von Haeseler, A. (1993). Split
decomposition: a technique to analyze viral evolution. Proc Natl Acad
Sci USA 90, 10320-10324.
74. Drake, J. W., Charlesworth, B., Charlesworth, D., and Crow, J. F.
(1998). Rates of spontaneous mutation. Genetics 148, 1667-1686.
75. Drummond, A. J., Ho, S.Y., Phillips, M.J., and Rambaut, A. (2006).
Relaxed phylogenetics and dating with confidence. PLoS Biol 4, e88.
76. Drummond, A.J., Nicholls, G.K., Rodrigo, A.G., and Solomon,
W. (2002). Estimating mutation parameters, population history and
genealogy simultaneously from temporally spaced sequence data.
Genetics 161, 1307-1320.
77. Drummond, A. J. and Rambaut, A. (2007). BEAST: Bayesian
evolutionary analysis by sampling trees. BMC Evol Biol 7, 214.
78. Drummond, A. J., Rambaut, A., Shapiro, B., and Pybus, O.G. (2005).
Bayesian coalescent inference of past population dynamics from
molecular sequences. Mol Biol Evol 22, 1185-1192.
79. Eck, R.V. and Dayhoff, M.O. (1966). Atlas of Protein Sequence
and Structure. Washington, USA: National Biomedical Research
Foundation.
80. Eddy, S.R. (1996). Hidden Markov models. Curr Opin Struct Biol 6,
361-365.
81. Eddy, S.R. (1998). Profile hidden Markov models. Bioinformatics 14,
755-763.
82. Eddy, S. R. (2004a). What is a hidden Markov model? Nat Biotechnol
22, 1315-1316.
83. Eddy, S.R. (2004b). What is Bayesian statistics? Nat Biotechnol 22,
1177-1178.
84. Eddy, S.R. (2004c). What is dynamic programming? Nat Biotechnol
22, 909-910.
85. Eddy, S.R. (2004d). Where did the BLOSUM62 alignment score
matrix come from? Nat Biotechnol 22, 1035-1036.
86. Edgar, R.C. (2004). MUSCLE: multiple sequence alignment with high
accuracy and high throughput. Nucleic Acids Res 32, 1792-1797.
87. Eigen, M. and Nieselt-Struwe, K. (1990). How old is the
immunodeficiency virus? AIDS 4, S85-S93.
88. Ellsworth, D. L., Hewett-Emmett, D., and Li, W.H. (1993). Insulin-
like growth factor II intron sequences support the hominoid rate-
slowdown hypothesis. Mol Phylogenet Evol 2, 315-321.
89. Farris, J.S. (1977). On the phenetic approach to vertebrate
classification. In: Major patterns in vertebrate evolution, pp. 823-
850. Edited by Hecht, M.K., Goody, P.C., and Hecht, B.M. New York,
USA: Plenum.
234 Литература
90. Felsenstein, J. (1973). Maximum-likelihood estimation of evolutionary
trees from continuous characters. Am J Hum Genet 25, 471-492.
91. Felsenstein, J. (1978). The number of evolutionary trees. Syst Zool
27, 27-33.
92. Felsenstein, J. (1981). Evolutionary trees from DNA sequences: a
maximum likelihood approach. J Mol Evol 17, 368-376.
93. Felsenstein, J. (1988). Phylogenies from molecular sequences:
inference and reliability. Annu Rev Genet 22, 521-565.
94. Felsenstein, J. (1990). Phylip manual version 3.2. Berceley, USA:
University Herbarium, University of California.
95. Felsenstein, J. (1996). Inferring phylogenies from protein sequences
by parsimony, distance, and likelihood methods. Methods Enzymol
266, 418-427.
96. Felsenstein, J. (2001). Taking variation of evolutionary rates between
sites into account in inferring phylogenies. J Mol Evol 53, 447-455.
97. Felsenstein, J. (2004). Inferring Phylogenies. Sunderland, USA:
Sinauer Associates.
98. Felsenstein, J. (2005). Using the quantitative genetic threshold model
for inferences between and within species. Philos Trans R Soc bond
В Biol Sei 360, 1427-1434.
99. Felsenstein, J. (2006). Accuracy of coalescent likelihood estimates: do
we need more sites, more sequences, or more loci? Mol Biol Evol 23,
691-700.
100. Felsenstein, J. (2008). Comparative methods with sampling error and
within-species variation: contrasts revisited and revised. Am Nat 171,
713-725.
101. Fitch, W.M. (1971). Towards defining the course of evolution:
minimum change for a specific tree topology. Syst Zool 20, 406-416.
102. Fitch, W.M. (1977). On the problem of discovering the most
parsimonious tree. Am Nat 111, 223-257.
103. Fitch, W.M. (1981). A non-sequential method for constructing trees
and hierarchical classifications. J Mol Evol 18, 30-37.
104. Fitch, W.M. and Atchley, W.R. (1985). Evolution in inbred strains of
mice appears rapid. Science 228, 1169-1175.
105. Fitch, W.M. and Langley, C.H. (1976). Protein evolution and the
molecular clock. Fed Proc 35, 2092-2097.
106. Fitch, W.M., Leiter, J.M.E., Li, X., and Palese, P. (1991). Positive
Darwinian evolution in human influenza A viruses. Proc Natl Acad
Sci USA 88, 4270-4274.
107. Fitch, W.M. and Margoliash, E. (1967). Construction of phylogenetic
trees. Science 155, 279-284.
108. Frank, A.C. and Lobry, J.R. (1999). Asymmetric substitution
patterns: a review of possible underlying mutational or selective
mechanisms. Gene 238, 65-77.
Литература 235
109. Fu, Y.X. (1995). Linear invariants under Jukes’ and Cantor’s one-
parameter model. J Theor Biol 173, 339-352.
110. Fu, Y.X. and Li, W.H. (1992). Construction of linear invariants in
phylogenetic inference. Math Biosci 109, 201-228.
111. Gardiner, K. (1995). Human genome organization. Curr Opin Genet
Dev 5, 315-322.
112. Gasbarra, D., Pirinen, M., Sillanpaa, M. J., Salmela, E., and Arjas, E.
(2007). Estimating genealogies from unlinked marker data: a Bayesian
approach. Theor Popul Biol 72, 305-322.
113. Gascuel, O. (1994). A note on Sattath and Tversky’s, Saitou and Nei’s,
and Studier and Keppler’s algorithms for inferring phylogenies from
evolutionary distances. Mol Biol Evol 11, 961-963.
114. Geer, R.C. and Sayers, E. W. (2003). Entrez: making use of its power.
Brief Bioinform 4, 179-184.
115. Gillespie, J. H. (1986a). Natural selection and the molecular clock.
Mol Biol Evol 3, 138-155.
116. Gillespie, J.H. (1986b). Statistical aspects of the molecular clock. In:
Evolutionary processes and theory, pp. 255-274. Edited by Karlin, S.
and Nevo, E. New York, USA: Academic Press.
117. Gillespie, J.H. (1986c). Variability of evolutionary rates of DNA.
Genetics 113, 1077-1091.
118. Gillespie, J.H. (1993). Episodic evolution of RNA viruses. Proc Natl
Acad Sci USA 90, 10411-10412.
119. Gillespie, J.H. and Langley, C.H. (1979). Are evolutionary rates really
variable? J Mol Evol 13, 27-34.
120. Gish, W. and States, D.J. (1993). Identification of protein coding
regions by database similarity search. Nat Genet 3, 266-272.
121. Glazko, G.V., Koonin, E.V., and Rogozin, LB. (2005). Molecular
dating: ape bones agree with chicken entrails. Trends Genet 21, 89-92.
122. Gojobori, T. and Moriyama, E.N. (1990). Molecular clock of viral
evolution, and the neutral theory. Proc Natl Acad Sci USA 87,
10015-10018.
123. Gojobori, T., Moriyama, E.N., Ina, Y., Ikeo, K., Miura, T., Tsujimoto,
H., Hayami, M., and Yokoyama, S. (1990). Evolutionary origin of
human and simian immunodeficiency viruses. Proc Natl Acad Sci
USA 87, 4108-4111.
124. Goldman, N. (1993). Statistical tests of models of DNA substitution.
J Mol Evol 36, 182-198.
125. Gonnet, G.H., Cohen, M.A., and Benner, S.A. (1992). Exhaustive
matching of the entire protein sequence database. Science 256, 1443—
1445.
126. Goodman, M. (1981a). Decoding the pattern of protein evolution. Prog
Biophys Mol Biol 38, 105-164.
236 Литература
127. Goodman, М. (1981b). Globin evolution was apparently very rapid
in early vertebrates: a reasonable case against the rate-constancy
hypothesis. J Mol Evol 17, 114-120.
128. Grantham, R., Gautier, C., Gouy, M., Mercier, R., and Pave, A. (1980).
Codon catalog usage and the genome hypothesis. Nucleic Acids Res
8, r49-r62.
129. Graur, D., Hide, W.A., and Li, W.H. (1991). Is the guinea-pig a
rodent? Nature 351, 649-652.
130. Graur, D., Hide, W.A., Zharkikh, A., and Li, W.H. (1992). The
biochemical phylogeny of guinea-pigs and gundis, and the paraphyly
of the order rodentia. Comp Biochem Physiol В 101, 495-498.
131. Graur, D. and Martin, W. (2004). Reading the entrads of chickens:
molecular timescales of evolution and the illusion of precision. Trends
Genet 20, 80-86.
132. Green, R. E., Krause, J., Ptak, S.E., Briggs, A. W., Ronan, M.T.,
Simons, J.F., Du, L., Egholm, M., Rothberg, J.M., Paunovic, M., and
Paabo, S. (2006). Analysis of one million base pairs of Neanderthal
DNA. Nature 444, 330-336.
133. Grigoriev, A. (1999). Strand-specific compositional asymmetries in
double-stranded DNA viruses. Virus Res 60, 1-19.
134. Gu, X. and Li, W.H. (1992). Higher rates of amino acid substitution
in rodents than in humans. Mol Phylogenet Evol 1, 211-214.
135. Gu, X. and Zhang, J. (1997). A simple method for estimating the
parameter of substitution rate variation among sites. Mol Biol Evol
14, 1106-1113.
136. Haeckel, E. (1879). The Evolution of Man: a Popular Exposition of
the Principal Points of Human Ontogeny and Phylogeny. 2 vols.
London, UK: Kegan Paul.
137. Hall, T.A. (1999). BioEdit: a user-friendly biological sequence
alignment editor and analysis program for Windows 95/98/NT. Nucl
Acids Res, Symposium Series 41, 95-98.
138. Hammer, M.F. (1995). A recent common ancestry for human Y
chromosomes. Nature 378, 376-378.
139. Hasegawa, M., Cao, Y., Adachi, J., and Yano, T. (1992). Rodent
polyphyly? Nature 355, 595.
140. Hasegawa, M., Kishino, H., and Yano, T. (1985). Dating of the human-
ape splitting by a molecular clock of mitochondrial DNA. J Mol Evol
22, 160-174.
Hedges, S.B., Chen, H., Kumar, S., Wang, D.Y., Thompson, A.S.,
and Watanabe, H. (2001). A genomic timescale for the origin of
eukaryotes. BMC Evol Biol 1, 4.
141. Hedges, S.B. and Kumar, S. (2003). Genomic clocks and evolutionary
timescales. Trends Genet 19, 200-206.
Литература 237
142. Hedges, S.B. and Kumar, S. (2004). Precision of molecular time
estimates. Trends Genet 20, 242-247.
143. Henikoff, S. and Henikoff, J.G. (1992). Amino acid substitution
matrices from protein blocks. Proc Natl Acad Set USA 89, 10915-
10919.
144. Hennig, W. (1950). Grundzuege einer Theorie der phylogenetischen
Systematik. Berlin, Germany: Deutscher Zentralverlag.
145. Higgins, D.G. and Sharp, P.M. (1988). CLUSTAL: a package for
performing multiple sequence alignment on a microcomputer. Gene
73, 237-244.
146. Higgins, D.G. and Sharp, P.M. (1989). Fast and sensitive multiple
sequence alignments on a microcomputer. Comput Appl Biosci 5, 151-
153.
147. Hillis, D.M., Bull, J. J., White, M.E., Badgett, M.R., and Molineux,
I. J. (1992). Experimental phylogenetics: generation of a known
phylogeny. Science 255, 589-592.
148. Ho, D.D., Neumann, A. U., Perelson, A.S., Chen, W., Leonard, J.M.,
and Markowitz, M. (1995). Rapid turnover of plasma virions and CD4
lymphocytes in HIV-1 infection. Nature 373, 123-126.
149. Hoffmann, J. W., Steffen, D., Gusella, J., Tabin, C., Bird, S., Cowing,
D., and Weinberg, R. A. (1982). DNA methylation affecting the
expression of murine leukemia proviruses. J Virol 44, 144-157.
150. Holmes, S. (2003). Statistics for phylogenetic trees. Theor Popul Biol
63, 17-32.
151. Hubbard, T„ Barker, D., Birney, E„ Cameron, G., Chen, Y., Clark, L.,
Cox, T., Cuff, J., Curwen, V., Down, T., Durbin, R., Eyras, E., Gilbert,
J., Hammond, M., Huminiecki, L., Kasprzyk, A., Lehvaslaiho, H.,
Lijnzaad, P., Melsopp, C., Mongin, E., Pettett, R., Pocock, M., Potter,
S., Rust, A., Schmidt, E., Searle, S., Slater, G., Smith, J., Spooner,
W., Stabenau, A., Stalker, J., Stupka, E., Ureta-Vidal, A., Vastrik, L,
and Clamp, M. (2002). The Ensembl genome database project. Nucleic
Acids Res 30, 38-41.
152. Huelsenbeck, J. P., Larget, B., Miller, R. E., and Ronquist, F. (2002).
Potential applications and pitfalls of Bayesian inference of phylogeny.
Syst Biol 51, 673-688.
153. Huelsenbeck, J. P. and Ronquist, F. (2001). MRBAYES: Bayesian
inference of phylogenetic trees. Bioinformatics 17, 754-755.
154. Huson, D.H. (1998). SplitsTree: analyzing and visualizing
evolutionary data. Bioinformatics 14, 68-73.
155. Huson, D.H. and Bryant, D. (2006). Application of phylogenetic
networks in evolutionary studies. Mol Biol Evol 23, 254-267.
156. Ikemura, T. (1981). Correlation between the abundance of Escherichia
coli transfer RNAs and the occurrence of the respective codons in
238 Литература
its protein genes: a proposal for a synonymous codon choice that is
optimal for the E. coli translational system. J Mol Biol 151, 389-409.
157. Ikemura, T. (1985). Codon usage and tRNA content in unicellular and
multicellular organisms. Mol Biol Evol 2, 13-34.
158. Ikemura, T. and Ozeki, H. (1983). Codon usage and transfer RNA
contents: organism-specific codon-choice patterns in reference to the
isoacceptor contents. Cold Spring Harb Symp Quant Biol 47, 1087-
1097.
159. Ishida, N., Oyunsuren, T., Mashima, S., Mukoyama, H., and Saitou,
N. (1995). Mitochondrial DNA sequences of various species of the
genus Equus with special reference to the phylogenetic relationship
between Przewalskii’s wild horse and domestic horse. J Mol Evol 41,
180-188.
160. Jabbari, K. and Bernardi, G. (2004). Cytosine methylation and CpG,
TpG (CpA) and TpA frequencies. Gene 333, 143-149.
161. Jackson, A.P. (2004). Cophylogeny of the Ficus microcosm. Biol Rev
Camb Philos Soc 79, 751-768.
162. Jackson, A.P. and Charleston, M.A. (2004). A cophylogenetic
perspective of RNA-virus evolution. Mol Biol Evol 21, 45-57.
163. Jeanmougin, F., Thompson, J.D., Gouy, M., Higgins, D.G., and
Gibson, T.J. (1998). Multiple sequence alignment with Clustal X.
Trends Biochem Sci 23, 403-405.
164. Jin, L. and Nei, M. (1990). Limitations of the evolutionary parsimony
method of phylogenetic analysis. Mol Biol Evol 7, 82-102.
165. Jobling, M.A. and Tyler-Smith, C. (1995). Fathers and sons: the Y
chromosome and human evolution. Trends Genet 11, 449-456.
166. Jones, D.T., Taylor, W.R., and Thornton, J.M. (1992a). The rapid
generation of mutation data matrices from protein sequences. Comput
Appl Biosci 8, 275-282.
167. Jones, P.A. and Laird, P. W. (1999). Cancer epigenetics comes of age.
Nat Genet 21, 163-167.
168. Jones, P.A., Rideout, W.M., III, Shen, J.C., Spruck, C.H., and Tsai,
Y.C. (1992b). Methylation, mutation and cancer. BioEssays 14, 33-36.
169. Jukes, Т.Н. and Cantor, C.R. (1969). Evolution of protein molecules.
In: Mammalian protein metabolism, pp. 21-123. Edited by Munro,
H. N. New York, USA: Academic Press.
170. Kagawa, Y., Nojima, H., Nukiwa, N., Ishizuka, M„ Nakajima, T.,
Yasuhara, T., Tanaka, T„ and Oshima, T. (1984). High guanine
plus cytosine content in the third letter of codons of an extreme
thermophile. DNA sequence of the isopropylmalate dehydrogenase of
Thermus thermophilus. J Biol Chem 259, 2956-2960.
171. Kaplan, D.L. and Steitz, T.A. (1999). DnaB from Thermus aquaticus
unwinds forked duplex DNA with an asymmetric tail length
dependence. J Biol Chem 274, 6889-6897.
Литература 239
172. Karlin, S., Doerfler, W., and Cardon, L.R. (1994). Why is CpG
suppressed in the genomes of virtually all small eukaryotic viruses
but not in those of large eukaryotic viruses? J Virol 68, 2889-2897.
173. Katoh, K., Misawa, К., Kuma, K., and Miyata, T. (2002). MAFFT:
a novel method for rapid multiple sequence alignment based on fast
Fourier transform. Nucleic Acids Res 30, 3059-3066.
174. Ke, Y., Su, B., Song, X., Lu, D., Chen, L., Li, H., Qi, C., Marzuki,
S., Deka, R., Underhill, P., Xiao, C., Shriver, M., Lell, J., Wallace,
D., Wells, R.S., Seielstad, M., Oefner, P., Zhu, D., Jin, J., Huang,
W., Chakraborty, R., Chen, Z., and Jin, L. (2001). African origin
of modern humans in East Asia: a tale of 12,000 Y chromosomes.
Science 292, 1151-1153.
175. Kimura, M. (1962). On the probability of fixation of mutant genes in
a population. Genetics 47, 713-719.
176. Kimura, M. (1968). Evolutionary rate at the molecular level. Nature
217, 624-626.
177. Kimura, M. (1980). A simple method for estimating evolutionary
rates of base substitutions through comparative studies of nucleotide
sequences. J Mol Evol 16, 111-120.
178. Kimura, M. (1983). The neutral theory of evolution. Cambridge, UK:
Cambridge University Press.
179. Kimura, M. and Ohta, T. (1969). The average number of generations
until fixation of a mutant gene in a finite population. Genetics 61,
763-771.
180. King, J.L. and Jukes, Т.Н. (1969). Non-Darwinian evolution. Science
164, 788-798.
181. Klotz, L.C., Komar, N., Blanken, R. L., and Mitchell, R.M. (1979).
Calculation of evolutionary trees from sequence data. Proc Natl Acad
Sci USA 76, 4516-4520.
182. Kohne, D.E. (1970). Evolution of higher-organism DNA. Q Rev
Biophys 3, 327-375.
183. Koonin, E.V. (2003). Horizontal gene transfer: the path to maturity.
Mol Microbiol 50, 725-727.
184. Koonin, E.V., Makarova, K.S., and Aravind, L. (2001). Horizontal
gene transfer in prokaryotes: quantification and classification. Annu
Rev Microbiol 55, 709-742.
185. Koop, B.F., Goodman, M., Xu, P., Chan, K., and Slightom, J. L.
(1986). Primate eta-globin DNA sequences and man’s place among
the great apes. Nature 319, 234-238.
186. Korber, B., Muldoon, M., Theiler, J-, Gao, F., Gupta, R., Lapedes, A.,
Hahn, B.H., Wolinsky, S., and Bhattacharya, T. (2000). Timing the
ancestor of the HIV-1 pandemic strains. Science 288, 1789-1796.
240 Литература
187. Korber, В., Theiler, J., and Wolinsky, S. (1998). Limitations of a
molecular clock applied to considerations of the origin of HIV-l.
Science 280, 1868-1871.
188. Kress, C., Thomassin, H., and Grange, T. (2001). Local DNA
demethylation in vertebrates: how could it be performed and targeted?
FEBS Lett 494, 135-140.
189. Kuhner, M.K. and Felsenstein, J. (1994). A simulation comparison
of phylogeny algorithms under equal and unequal evolutionary rates.
Mol Biol Evol 11, 459-468.
190. Kuiken, C.L., Cornelissen, M.T.E., Zorgdrager, F., Hartman, S.,
Gibbs, A. J., and Goudsmit, J. (1996). Consistent risk group-associated
differences in human immunodeficiency virus type 1 vpr, vpu and V3
sequences despite independent evolution. J Gen Virol 77, 783-792.
191. Kuiken, C.L. and Goudsmit, J. (1994). Silent mutation pattern in
V3 sequences distinguishes virus according to risk group in Europe.
AIDS Res Hum. Retroviruses 10, 319-320.
192. Kumar, S. and Hedges, S.B. (1998). A molecular timescale for
vertebrate evolution. Nature 392, 917-920.
193. Kumar, S., Nei, M., Dudley, J., and Tamura, K. (2008). MEGA:
a biologist-centric software for evolutionary analysis of DNA and
protein sequences. Brief Bioinform 9, 299-306.
194. Kumar, S., Tamura, K., Jakobsen, I.B., and Nei, M. (2001). MEGA2:
molecular evolutionary genetics analysis software. Bioinformatics 17,
1244-1245.
195. Kumar, S., Tamura, K. and Nei, M. (1993). Molecular evolutionary
genetics analysis (MEGA). University Park, USA: Institute of
Molecular Evolutionary Genetics, Pennsylvania State University.
196. Kumar, S., Tamura, K., and Nei, M. (1994). MEGA: Molecular
Evolutionary Genetics Analysis software for microcomputers. Comput
Appl Biosci 10, 189-191.
197. Kumar, S., Tamura, K., and Nei, M. (2004). MEGA3: Integrated
software for Molecular Evolutionary Genetics Analysis and sequence
alignment. Brief Bioinform 5, 150-163.
198. Kushiro, M., Shimizu, M., and Tomita, K. (1987). Molecular cloning
and sequence determination of the tuf gene coding for the elongation
factor Tu of Thermus thermophilus HB8. Eur J Biochem 170, 93-98.
199. Laird, C.D., McConaughy, B.L., and McCarthy, B.J. (1969). Rate of
fixation of nucleotide substitutions in evolution. Nature 224, 149-154.
200. Lake, J.A. (1987). A rate-independent technique for analysis of
nucleic acid sequences: evolutionary parsimony. Mol Biol Evol 4,
167-191.
201. Langley, C.H. and Fitch, W.M. (1974). An examination of the
constancy of the rate of molecular evolution. J Mol Evol 3, 161-177.
Литература 241
202. Larkin, М.А., Blackshields, G., Brown, N.P., Chenna, R.,
McGettigan, P.A., Me William, H., Valentin, F., Wallace, I.M., Wilm,
A., Lopez, R., Thompson, J. D., Gibson, T.J., and Higgins, D.G.
(2007). Clustal W and Clustal X version 2.0. Bioinformatics 23,
2947-2948.
203. Lazouskaya, N. V., Eremin, V.F., Adema, K.W., Gasich, E.L., Baan,
E., and Lukashov, V. V. (2005). The HIV type 1 epidemic in Belarus:
predominance of Eastern European subtype A strains and circulation
of subtype В viruses. AIDS Res Hum Retroviruses 21, 830-833.
204. Li, P. and Bousquet, J. (1992). Relative-rate test for nucleotide
substitutions between two lineages. Mol Biol Evol 9, 1185-1189.
205. Li, W.H. (1993). Unbiased estimation of the rates of synonymous and
nonsynonymous substitution. J Mol Evol 36, 96-99.
206. Li, W.H. (1997). Molecular Evolution. Sunderland, USA: Sinauer
Associates.
207. Li, W.H., Hide, W.A., and Graur, D. (1992). Origin of rodents and
guinea-pigs. Nature 359, 277-278.
208. Li, W.H. and Tanimura, M. (1987). The molecular clock runs more
slowly in man than in apes and monkeys. Nature 326, 93-96.
209. Li, W.H., Tanimura, M., and Sharp, P. M. (1987). An evaluation of
the molecular clock hypothesis using mammalian DNA sequences. J
Mol Evol 25, 330-342.
210. Li, W.H., Tanimura, M., and Sharp, P.M. (1988). Rates and dates of
divergence between AIDS virus nucleotide sequences. Mol Biol Evol
5, 313-330.
211. Li, W.H., Wu, C.I., and Luo, C.C. (1985). A new method for
estimating synonymous and nonsynonymous rates of nucleotide
substitution considering the relative likelihood of nucleotide and codon
changes. Mol Biol Evol 2, 150-174.
212. Liitsola, K., Laukkanen, T., Denisova, A., Grishkevichius, A.,
Smolskaja, T., Ustina, V., Vlasov, N., Leinikki, P., and Salminen,
M.O. (1996). Genetic characterization of HIV-1 strains in the Baltic
countries and Russia. Scand J Infect Dis 28, 537-541.
213. Liitsola, K„ Tashkinova, I., Laukkanen, T., Korovina, G., Smolskaja,
T„ Momot, O., Mashkilleyson, N., Chaplinskas, S., Brummer-
Korvenkontio, H., Vanhatalo, J., Leinikki, P., and Salminen, M.O.
(1998). HIV-1 genetic subtype A/В recombinant strain causing an
explosive epidemic in injecting drug users in Kaliningrad. AIDS 12,
1907-1919.
214. Lin, Y.H., McLenachan, P.A., Gore, A.R., Phillips, M.J., Ota,
R„ Hendy, M.D., and Penny, D. (2002a). Four new mitochondrial
genomes and the increased stability of evolutionary trees of mammals
from improved taxon sampling. Mol Biol Evol 19, 2060-2070.
242 Литература
215. Lin, Y.H., Waddell, P.J., and Penny, D. (2002b). Pika and vole
mitochondrial genomes increase support for both rodent monophyly
and glires. Gene 294, 119-129.
216. Linnaei, C. (1735). Systema naturae. Lugduni Batavorum: Leiden,
The Netherlands. Apud Theodorum Haak. Ex Typographia Joannis
Wilhelmi de Groot.
217. Lobry, J.R. (1996). Origin of replication of Mycoplasma genitalium.
Science 272, 745-746.
218. Lole, K.S., Bollinger, R.C., Paranjape, R.S., Gadkari, D., Kulkarni,
S.S., Novak, N.G., Ingersoll, R., Sheppard, H. W., and Ray, S.C.
(1999). Full-length human immunodeficiency virus type 1 genomes
from subtype C-infected seroconverters in India, with evidence of
intersubtype recombination. J Virol 73, 152-160.
219. Long, M. and Gillespie, J.H. (1991). Codon usage divergence of
homologous vertebrate genes and codon usage clock. J Mol Evol 32,
6-15.
220. Lukashov, V.V., Cornelissen, M.T.E., Goudsmit, J., Papuashvilli,
M.N., Rytik, P.G., Khaitov, R. M., Karamov, E.V., and de Wolf,
F. (1995). Simultaneous introduction of distinct HIV-1 subtypes into
different risk groups in Russia, Byelorussia and Lithuania. AIDS 9,
435-439.
221. Lukashov, V.V. and Goudsmit, J. (1997a). Evolution of the human
immunodeficiency virus type 1 subtype-specific V3 domain is
confined to a sequence space with a fixed distance to the subtype
consensus. J Virol 71, 6332-6338.
222. Lukashov, V.V. and Goudsmit, J. (1997b). Founder virus population
related to route of virus transmission: a determinant of intrahost
human immunodeficiency virus type 1 evolution? J Virol 71, 2023-
2030.
223. Lukashov, V.V. and Goudsmit, J. (1998). HIV-1 intrasubtype
diversification originates from the moment of introduction of virulent
HIV-1 strains into current AIDS risk groups in the 1960s and 1970s.
Keystone Symposium «HIV Pathogenesis and Treatment», March
13-19, 1998, Park City, USA, p.95 (Abstract 4058).
224. Lukashov, V.V. and Goudsmit, J. (2000). The pros and cons of
studying HTV evolution in real time. Cell 101, 129-131.
225. Lukashov, V.V. and Goudsmit, J. (2001). Evolutionary relationships
among parvoviruses: virus-host coevolution among autonomous
primate parvoviruses and links between adeno-associated and avian
parvoviruses. J Virol 75, 2729-2740.
226. Lukashov, V.V. and Goudsmit, J. (2002a). Evolutionary relationships
among Astroviridae. J Gen Virol 83, 1397-1405.
227. Lukashov, V.V. and Goudsmit, J. (2002b). Recent evolutionary
history of human immunodeficiency virus type 1 subtype B:
Литература 243
reconstruction of epidemic onset based on sequence distances to the
common ancestor. J Mol Evol 54, 680-691.
228. Lukashov, V. V. and Goudsmit, J. (2003). Recent evolutionary history
of HIV-1 subtype B. J Mol Evol 56, 645-647.
229. Lukashov, V.V., Huismans, R., Rakhmanova, A. G., Lisitsina, Z.N.,
Akhtyrskaya, N.A., Vlasov, N.N., Melnick, O.B., and Goudsmit, J.
(1999). Circulation of subtype A and gagA/envB recombinant HIV
type 1 strains among injecting drug users in St. Petersburg, Russia,
correlates with geographical origin of infections. AIDS Res Hum
Retroviruses 15, 1577-1583.
230. Lukashov, V.V., Karamov, E.V., Eremin, V.F., Titov, L.P., and
Goudsmit, J. (1998a). Extreme founder effect in an HIV type 1
subtype A epidemic among drug users in Svetlogorsk, Belarus. AIDS
Res Hum Retroviruses 14, 1299-1303.
231. Lukashov, V.V., Kuiken, C.L., Boer, K., and Goudsmit, J. (1996a).
HIV type 1 subtypes in The Netherlands circulating among women
originating from AIDS endemic regions. AIDS Res Hum Retroviruses
12, 951-953.
232. Lukashov, V.V., Kuiken, C.L., Vlahov, D., Coutinho, R.A., and
Goudsmit, J. (1996b). Evidence for HIV type 1 strains of U.S.
intravenous drug users as founders of AIDS epidemic among
intravenous drug users in Northern Europe. AIDS Res Hum
Retroviruses 12, 1179-1183.
233. Lukashov, V.V., Op de Coul, E.L., Coutinho, R.A., and Goudsmit,
J. (1998b). HIV-1 strains specific for Dutch injecting drug users in
heterosexually infected individuals in The Netherlands. AIDS 12,
635-641.
234. Madden, T.L., Tatusov, R.L., and Zhang, J. (1996). Applications of
network BLAST server. Methods Enzymol 266, 131-141.
235. Makarenkov, V., Kevorkov, D., and Legendre, P. (2006). Phylogenetic
network reconstruction approaches. Appl Mycol Biotechnol,
International Elsevier Series, Bioinformatics, vol. 6, 61-97.
236. Margoliash, E. (1963). Primary structure and evolution of cytochrome
C. Proc Natl Acad Sci USA 50, 672-679.
237. Martin, A.P. (2000). Choosing among alternative trees of multigene
families. Mol Phylogenet Evol 16, 430-439.
238. Martin, D. and Rybicki, E. (2000). RDP: detection of recombination
amongst aligned sequences. Bioinformatics 16, 562-563.
239. Maruyama, T. and Kimura, M. (1974). A note on the speed of
gene frequency changes in reverse directions in a finite population.
Evolution 28, 162-163.
240. Mayr, E. (1954). Changes in genetic environment and evolution. In:
Evolution as a process, pp. 157-180. Edited by Huxley, J., Hardy,
A.C., and Ford, E.B. London, UK: Allen and Unwin.
244 Литература
241. Mayr, Е. (1963). Animal Species and Evolution. Cambridge, USA:
Harvard University Press.
242. McGeoch, D.J., Cook, S., Dolan, A., Jamieson, F.E., and Telford,
E.A. R. (1995). Molecular phylogeny and evolutionary timescale for
the family of mammahan herpesviruses. J Mol Biol 247, 443-458.
243. Mirkin, B.G., Fenner, T.I., Galperin, M. Y., and Koonin, E. V. (2003).
Algorithms for computing parsimonious evolutionary scenarios for
genome evolution, the last universal common ancestor and dominance
of horizontal gene transfer in the evolution of prokaryotes. BMC Evol
Biol 3, 2.
244. Moretti, S., Armougom, F., Wallace, I.M., Higgins, D.G., Jongeneel,
C.V., and Notredame, C. (2007). The М-Coffee web server: a meta-
method for computing multiple sequence alignments by combining
alternative alignment methods. Nucleic Acids Res 35, W645-W648.
245. Moretti, S., Wilm, A., Higgins, D.G., Xenarios, I., and Notredame, C.
(2008). R-Coffee: a web server for accurately aligning noncoding RNA
sequences. Nucleic Acids Res.
246. Morris, S.C. (1998). Metazoan phylogenies: falling into place or falling
to pieces? A palaeontological perspective. Curr Opin Genet Dev 8,
662-667.
247. Muse, S. V. and Weir, B.S. (1992). Testing for equality of evolutionary
rates. Genetics 132, 269-276.
248. Muto, A. and Osawa, S. (1987). The guanine and cytosine content of
genomic DNA and bacterial evolution. Proc Natl Acad Sci USA 84,
166-169.
249. Muto, A., Yamao, F., Hori, H., and Osawa, S. (1986). Gene
organization of Mycoplasma capricolum. Adv Biophys 21, 49-56.
250. Myers, G., Korber, B., Hahn, B.H., Jeang, K.-T., Mellors, J. W.,
McCutchen, F.E., Henderson, L.E. and Pavlakis, G.N. (1995).
Human Retroviruses and AIDS: a Compilation and Analysis
of Nucleic Acid and Amino Acid Sequences. Los Alamos, USA:
Theoretical Biology and Biophysics, Group T-10, Los Alamos National
Laboratory.
251. Nabatov, A. A., Kravchenko, O.N., Lyulchuk, M.G., Shcherbinskaya,
A. M., and Lukashov, V. V. (2002). Simultaneous introduction of HIV
type 1 subtype A and В viruses into injecting drug users in southern
Ukraine at the beginning of the epidemic in the former Soviet Union.
AIDS Res Hum Retroviruses 18, 891-895.
252. Needleman, S.B. and Wunsch, C.D. (1970). A general method
applicable to the search for similarities in the amino acid sequence of
two proteins. J Mol Biol 48, 443-453.
253. Nei, M., Chakraborty, R.. and Fuerst, P.A. (1976). Infinite allele
model with varying mutation rate. Proc Natl Acad Sci USA 73,
4164-4168.
Литература 245
254. Nei, М. and Gojobori, T. (1986). Simple methods for estimating the
numbers of synonymous and nonsynonymous nucleotide substitutions.
Mol Biol Evol 3, 418-426.
255. Nichol, S.T., Rowe, J.E., and Fitch, W.M. (1993). Punctuated
equilibrium and positive Darvinian evolution in vesicular stomatitis
virus. Proc Natl Acad Sci USA 90, 10424-10428.
256. Nishiyama, M., Matsubara, N„ Yamamoto, K., lijima, S., Uozumi,
T., and Beppu, T. (1986). Nucleotide sequence of the malate
dehydrogenase gene of Thermus flavus and its mutation directing
an increase in enzyme activity. J Biol Chem 261, 14178-14183.
257. Noonan, J.P., Coop, G., Kudaravalli, S., Smith, D., Krause, J.,
Alessi, J., Chen, F., Platt, D., Paabo, S., Pritchard, J. K., and Rubin,
E.M. (2006). Sequencing and analysis of Neanderthal genomic DNA.
Science 314, 1113-1118.
258. Notredame, C., Higgins, D.G., and Heringa, J. (2000). Т-Coffee: A
novel method for fast and accurate multiple sequence alignment. J
Mol Biol 302, 205-217.
259. Notredame, C. and Suhre, K. (2004). Computing multiple
sequence/structure alignments with the T-coffee package. Curr
Protoc Bioinformatics Chapter 3, Unit3.
260. Novitsky, V.A., Montano, M.A., and Essex, M. (1998). Molecular
epidemiology of an HIV-1 subtype A subcluster among injection drug
users in the Southern Ukraine. AIDS Res Hum Retroviruses 14,
1079-1085.
261. O’hUigin, C. and Li, W.H. (1992). The molecular clock ticks regularly
in muroid rodents and hamsters. J Mol Evol 35, 377-384.
262. Ochman, H. and Wilson, A.C. (1987). Evolution in bacteria: evidence
for a universal substitution rate in cellular genomes. J Mol Evol 26,
74-86.
263. Ono, M., Matsuzawa, H., and Ohta, T. (1990). Nucleotide sequence and
characteristics of the gene for L-lactate dehydrogenase of Thermus
aquaticus YT-1 and the deduced amino acid sequence of the enzyme.
J Biochem 107, 21-26.
264. Op de Coul, E.L., Coutinho, R.A., van der Schoot, A., van Doornum,
G.J., Lukashov, V.V., Goudsmit, J., and Cornelissen, M. (2001a).
The impact of immigration on env HIV-1 subtype distribution among
heterosexuals in the Netherlands: influx of subtype В and non-B
strains. AIDS 15, 2277-2286.
265. Op de Coul, E.L., Prins, M., Cornelissen, M., van der Schoot A.,
Boufassa, F„ Brettle, R. P., Hernandez-Aguado, L., Schiffer, V.,
McMenamin, J., Rezza, G., Robertson, R., Zangerle. R-, Goudsmit,
J., Coutinho, R.A., and Lukashov, V.V. (2001b). Using phylogenetic
analysis to trace HIV-1 migration among western European injecting
drug users seroconverting from 1984 to 1997. AIDS , 257-266.
246 Литература
266. Orito, Е„ Mizokami, М., Ina, Y„ Moriyama, E.N., Kameshima, N.,
Yamamoto, M., and Gojobori, T. (1989). Host-independent evolution
and a genetic classification of the hepadnavirus family based on
nucleotide sequences. Proc Natl Acad Sci USA 86, 7059-7062.
267. Ou, C.-Y., Ciesielski, C.A., Myers, G., Bandea, C.I., Luo, C.-
С., Korber, B.T.M., Mullins, J. I., Schochetman, G., Berkelman,
R. L., Economou, A. N., Witte, J. J., Furman, L.J., Satten, G.A.,
Maclnnes, K.A., Curran, J. W., Jaffe, H. W., Laboratory Investigation
Group, and Epidemiologic Investigation Group (1992). Molecular
epidemiology of HIV transmission in a dental practice. Science 256,
1165-1171.
268. Pamilo, P. and Bianchi, N.O. (1993). Evolution of the Zfx and Zfy
genes: rates and interdependence between the genes. Mol Biol Evol
10, 271-281.
269. Pascarella, S. and Argos, P. (1992). Analysis of insertions/deletions
in protein structures. J Mol Biol 224, 461-471.
270. Pei, J., Sadreyev, R., and Grishin, N.V. (2003). PCMA: fast and
accurate multiple sequence alignment based on profile consistency.
Bioinformatics 19, 427-428.
271. Poirot, O., Suhre, K., Abergel, C., O’Toole, E., and Notredame, C.
(2004). 3DCoffee@igs: a web server for combining sequences and
structures into a multiple sequence alignment. Nucleic Acids Res 32,
W37-W40.
272. Posada, D. and Crandall, K.A. (2001a). Evaluation of methods for
detecting recombination from DNA sequences: computer simulations.
Proc Natl Acad Sci USA 98, 13757-13762.
273. Posada, D. and Crandall. K.A. (2001b). Selecting the best-fit model of
nucleotide substitution. Syst Biol 50, 580-601.
274. Preston, B.D., Poiesz, B. J., and Loeb, L.A. (1988). Fidelity of HIV-1
reverse transcriptase. Science 242, 1168-1171.
275. Pyre, K., Dijkman, R., Deng, L., Jebbink, M.F., Ross, H.A.,
Berkhout, B., and van der Hoek, L. (2006). Mosaic structure of human
coronavirus NL63, one thousand years of evolution. J Mol Biol 364,
964-973.
276. Querat, G., Audoly, G., Sonigo, P., and Vigne, R. (1990). Nucleotide
sequence analysis of SA-OMVV, a visna-related ovine lentivirus:
phylogenetic history of lentiviruses. Virol 175, 434-447.
277. Ranwez, V. and Gascuel, O. (2001). Quartet-based phylogenetic
inference: improvements and limits. Mol Biol Evol 18, 1103-1116.
278. Riaz, T., Yi, W., and Li, K.B. (2005). A tabu search algorithm for
post-processing multiple sequence alignment. J Bioinform Comput
Biol 3, 145-156.
Литература 247
279. Richter, С., Park, J.W., and Ames, B.N. (1988). Normal oxidative
damage to mitochondrial and nuclear DNA is extensive. Proc Natl
Acad Sci USA 85, 6465-6467.
280. Roberts, J. D., Preston, B. D., Johnston, L.A., Soni, A., Loeb,
L.A., and Kunkel, T.A. (1989). Fidelity of two retroviral reverse
transcriptases during DNA-dependent DNA synthesis in vitro. Mol
Cell Biol 9, 469-476.
281. Robertson, D.L., Anderson, J.P., Bradac, J. A., Carr, J.K., Foley, B.,
Funkhouser, R.K., Gao, F., Hahn, B.H., Kalish, M.L., Kuiken, C.,
Learn, G. H., Leitner, T., McCutchan, F., Osmanov, S., Peeters, M.,
Pieniazek, D., Salminen, M., Sharp, P.M., Wolinsky, S., and Korber,
B. (2000). HIV-1 nomenclature proposal. Science 288, 55-56.
282. Robinson-Rechavi, M., Ponger, L., and Mouchiroud, D. (2000).
Nuclear gene LCAT supports rodent monophyly. Mol Biol Evol 17,
1410-1412.
283. Rogers, J.S. and Swofford, D.L. (1998). A fast method for
approximating maximum likelihoods of phylogenetic trees from
nucleotide sequences. Sy st Biol 47, 77-89.
284. Ronquist, F. and Huelsenbeck, J. P. (2003). MrBayes 3: Bayesian
phylogenetic inference under mixed models. Bioinformatics 19,
1572-1574.
285. Rutherford, K., Parkhill, J., Crook, J., Horsnell, T., Rice, P.,
Rajandream, M.A., and Barrell, B. (2000). Artemis: sequence
visualization and annotation. Bioinformatics 16, 944-945.
286. Rzhetsky, A. and Nei, M. (1993). Theoretical foundation of the
minimum-evolution method of phylogenetic inference. Mol Biol Evol
10, 1073-1095.
287. Saitou, N. and Imanishi, T. (1989). Relative efficiencies of the Fitch-
Margoliash, maximum-parsimony, maximum-likelihood, minimum-
evolution, and neighbor-joining methods of phylogeuetic tree
construction in obtaining the correct tree. Mol Biol Evol 6, 514-525.
288. Saitou, N. and Nei, M. (1987). The neighbor-joining method: a new
method for reconstructing phylogenetic trees. Mol Biol Evol 4, 406-
425.
289. Saitou, N. and Ueda, S. (1994). Evolutionary rates of insertion and
deletion in noncoding nucleotide sequences of primates. Mol Biol
Evol 11, 504-512.
290. Salemi, M., Strimmer, K., Hall, W.W., Duffy, M., Delaporte, E.,
Mboup, S., Peeters, M., and Vandamme, A. M. (2001). Dating the
common ancestor of SIVcpz and HIV-1 group M and the origin of
HIV-1 subtypes using a new method to uncover clock-like molecular
evolution. FASEB J 15, 276-278.
248 Литература
291. Sanchez, С.М., Gebauer, F., Sune, C., Mendez, A., Dopazo, J., and
Enjuanes, L. (1992). Genetic evolution and tropism of transmissible
gastroenteritis coronaviruses. Virol 190, 92-105.
292. Sandelin, E. (2005). Extracting multiple structural alignments from
pairwise alignments: a comparison of a rigorous and a heuristic
approach. Bioinformatics 21, 1002-1009.
293. Sarich, V.M. and Wilson, A.C. (1973). Generation time and genomic
evolution in primates. Science 179, 1144-1147.
294. Sattath, S. and Tversky, A. (1977). Additive similarity trees.
Psychometrika 42, 319-345.
295. Scarano, E., laccarino, M., Grippo, P., and Parisi, E. (1967). The
heterogeneity of thymine methyl group origin in DNA pyrimidine
isostichs of developing sea urchin embryos. Proc Natl Acad Sci USA
57, 1394-1400.
296. Schmidt, H.A., Strimmer, K., Vingron, M., and von Haeseler, A.
(2002). TREE-PUZZLE: maximum likelihood phylogenetic analysis
using quartets and parallel computing. Bioinformatics 18, 502-504.
297. Schulmeister, S. (2004). Inconsistency of maximum parsimony
revisited. Syst Biol 53, 521-528.
298. Seino, S., Bell, G.I., and Li, W.H. (1992). Sequences of primate
insulin genes support the hypothesis of a slower rate of molecular
evolution in humans and apes than in monkeys. Mol Biol Evol 9,
193-203.
299. Sernova, N.V. and Gelfand, M.S. (2008). Identification of replication
origins in prokaryotic genomes. Brief Bioinform 9, 376-391.
300. Shackelton, L.A., Parrish, C.R., and Holmes, E.C. (2006).
Evolutionary basis of codon usage and nucleotide composition bias
in vertebrate DNA viruses. J Mol Evol 62, 551-563.
301. Shapiro, B., Rambaut, A., and Drummond, A. J. (2006). Choosing
appropriate substitution models for the phylogenetic analysis of
protein-coding sequences. Mol Biol Evol 23, 7-9.
302. Sharp, P.M. and Cowe, E. (1991). Synonymous codon usage in
Saccharomyces cerevisiae. Yeast 7, 657-678.
303. Sharp, P.M. and Li, W.H. (1987). The codon Adaptation Index — a
measure of directional synonymous codon usage bias, and its potential
applications. Nucleic Acids Res 15, 1281-1295.
304. Sharp, P.M. and Li, W. H. (1988). Understanding the origins of AIDS
viruses. Nature 336, 315.
305. Sharp, P.M., Rogers, M.S., and McConnell, D.J. (1985). Selection
pressures on codon usage in the complete genome of bacteriophage
T7. J Mol Evol 21, 150-160.
306. Sharp, P. M., Stenico, M., Peden, J. F., and Lloyd, A. T. (1993). Codon
usage: mutational bias, translational selection, or both? Biochem Soc
Trans 21, 835-841.
Литература 249
307. Sharp, Р. М., Tuohy, Т.М., and Mosurski, K.R. (1986). Codon usage
in yeast: cluster analysis clearly differentiates highly and lowly
expressed genes. Nucleic Acids Res 14, 5125-5143.
308. Shpaer, E.G. and Mullins, J. I. (1990). Selection against CpG
dinucleotides in lentiviral genes: a possible role of methylation in
regulation of viral expression. Nucleic Acids Res 18, 5793-5797.
309. Siebert, S. and Backofen, R. (2005). MARNA: multiple alignment and
consensus structure prediction of RNAs based on sequence structure
comparisons. Bioinformatics 21, 3352-3359.
310. Simossis, V.A., Kleinjung, J., and Heringa, J. (2005). Homology-
extended sequence alignment. Nucleic Acids Res 33, 816-824.
311. Smith, T.F. and Waterman, M.S. (1981). Identification of common
molecular subsequences. J Mol Biol 147, 195-197.
312. Smith, T.F., Waterman, M.S., and Burks, C. (1985). The statistical
distribution of nucleic acid similarities. Nucleic Acids Res 13, 645-
656.
313. Smith, U.R., Kuiken, C., and Korber, B.T. (2003). Recent
evolutionary history of human immunodeficiency virus type 1
subtype В - response. J Mol Evol 56, 643-644.
314. Snir, S., Warnow, T., and Rao, S. (2008). Short quartet puzzling: a new
quartet-based phylogeny reconstruction algorithm. J Comput Biol 15,
91-103.
315. Sokal, R.R. and Michiner, C.D. (1958). A statistical method for
evaluating systematic relationships. In: University of Kansas Science
Bulletin, pp. 1409-1438.
316. Stenico, M., Lloyd, A.T., and Sharp, P.M. (1994). Codon usage
in Caenorhabditis elegans: delineation of translational selection and
mutational biases. Nucleic Acids Res 22, 2437-2446.
317. Strimmer, K., Goldman, N., and von Haeseler, A. (1997). Bayesian
probabilities and quartet puzzling. Mol Biol Evol 14, 210-211.
318. Strimmer, K. and von Haeseler, A. (1996). Quartet puzzling: a quartet
maximum-likelihood method for reconstructing tree topologies. Mol
Biol Evol 13, 964-969.
319. Studier, J. A. and Keppler, K. J. (1988). A note on the neighbor-joining
algorithm of Saitou and Nei. Mol Biol Evol 5, 729-731.
320. Subramanian, A. R., Kaufmann, M., and Morgenstern, B. (2008).
DIALIGN-TX: greedy and progressive approaches for segment-based
multiple sequence alignment. Algorithms Mol Biol 3, 6.
321. Sueoka, N. (1964). On the evolution of informational macromolecules.
In: Evolving genes and proteins, pp. 479-496. Edited by Bryson, V.
and Vogel, H. J. New York, USA: Academic Press.
322. Sueoka, N., Marmur, J., and Doty 2-nd, P. (1959). Dependence of the
density of deoxyribonucleic acids on guanine-cytosine content. Nature
183, 1429-1431.
250 Литература
323. Sutovsky, Р., Moreno, R.D., Ramalho-Santos, J., Dominko, T.,
Simerly, C., and Schatten, G. (1999). Ubiquitin tag for sperm
mitochondria. Nature 402, 371-372.
324. Sutovsky, P. and Schatten, G. (2000). Paternal contributions to the
mammalian zygote: fertilization after sperm-egg fusion. I nt Rev Cytol
195, 1-65.
325. Swofford, D.L. (2002). Phylogenetic analysis using parsimony (*and
other methods). Version 4. Sunderland, USA: Sinauer Associates.
326. Tajima, F. and Nei, M. (1984). Estimation of evolutionary distance
between nucleotide sequences. Mol Biol Evol 1, 269-285.
327. Tamura, K. (1992). Estimation of the number of nucleotide
substitutions when there are strong transition-transversion and G+C-
content biases. Mol Biol Evol 9, 678-687.
328. Tamura, K., Dudley, J., Nei, M., and Kumar, S. (2007). MEGA4:
Molecular Evolutionary Genetics Analysis (MEGA) software version
4.0. Mol Biol Evol 24, 1596-1599.
329. Tamura, K. and Nei, M. (1993). Estimation of the number of
nucleotide substitutions in the control region of mitochondrial DNA
in humans and chimpanzees. Mol Biol Evol 10, 512-526.
330. Taubenberger, J.K., Reid, A. H., Lourens, R. M., Wang, R., Jin, G.,
and Fanning, T.G. (2005). Characterization of the 1918 influenza
virus polymerase genes. Nature 437, 889-893.
331. Thompson, J.D., Gibson, T.J., Plewniak, F., Jeanmougin, F., and
Higgins, D.G. (1997). The CLUSTAL_X windows interface: flexible
strategies for multiple sequence alignment aided by quality analysis
tools. Nucleic Acids Res 25, 4876-4882.
332. Thompson, J.D., Higgins, D.G., and Gibson, T.J. (1994). CLUSTAL
W: improving the sensitivity of progressive multiple sequence
alignment through sequence weighting, position-specific gap penalties
and weight matrix choice. Nucleic Acids Res 22, 4673-4680.
333. Tillier, E.R. and Collins, R.A. (2000). The contributions of replication
orientation, gene direction, and signal sequences to base-composition
asymmetries in bacterial genomes. J Mol Evol 50, 249-257.
334. Torroni, A., Achilli, A., Macaulay, V., Richards, M., and Bandelt, H. J.
(2006). Harvesting the fruit of the human mtDNA tree. Trends Genet
22, 339-345.
335. Tsai, C.T., Lin, C.H., and Chang, C.Y. (2007). Analysis of codon
usage bias and base compositional constraints in iridovirus genomes.
Virus Res 126, 196-206.
336. Tumpey, T.M., Basler, C.F., Aguilar, P.V., Zeng, H., Solorzano, A.,
Swayne, D.E., Cox, N.J., Katz, J. M., Taubenberger, J. K., Palese, P.,
and Garcia-Sastre, A. (2005). Characterization of the reconstructed
1918 Spanish influenza pandemic virus. Science 310, 77-80.
Литература 251
337. Underhill, Р.А., Passarino, G., Lin, A. A., Marzuki, S., Oefner, P.J.,
Cavalli-Sforza, L.L., and Chambers, G.K. (2001a). Maori origins, Y-
chromosome haplotypes and implications for human history in the
Pacific. Hum Mutat 17, 271-280.
338. Underhill, P.A., Passarino, G., Lin, A. A., Shen, P., Mirazon, L.M.,
Foley, R.A., Oefner, P.J., and Cavalli-Sforza, L.L. (2001b). The
phylogeography of Y chromosome binary haplotypes and the origins
of modern human populations. Ann Hum Genet 65, 43-62.
339. Uzzell, T. and Corbin, K.W. (1971). Fitting discrete probability
distributions to evolutionary events. Science 172, 1089-1096.
340. van Hemert, F. J. and Berkhout, B. (1995). The tendency of lentiviral
open reading frames to become А-rich: constraints imposed by viral
genome organization and cellular tRNA availability. J Mol Evol 41,
132-140.
341. van Hemert, F.J., Berkhout, B., and Lukashov, V.V. (2007). Host-
related nucleotide composition and codon usage as driving forces in
the recent evolution of the Astroviridae. Virol 361, 447-454.
342. Vartanian, J. P., Meyerhans, A., Sala, M., and Wain-Hobson, S.
(1994). G—»A hypermutation of the human immunodeficiency virus
type 1 genome: evidence for dCTP pool imbalance during reverse
transcription. Proc Natl Acad Sci USA 91, 3092-3096.
343. Vartanian, J.-P., Meyerhans, A., Asjo, B., and Wain-Hobson, S.
(1991). Selection, recombination and G—»A hypermutation of human
immunodeficiency virus type 1 genomes. J Virol 65, 1779-1788.
344. Vigilant, L., Stoneking, M., Harpending, H., Hawkes, K„ and
Wilson, A.C. (1991). African populations and the evolution of human
mitochondrial DNA. Science 253, 1503-1507.
345. Villaverde, A., Martinez, M. A., Sobrino, F., Dopazo, J., Moya, A., and
Domingo, E. (1991). Fixation of mutations at the VP1 gene of foot-and-
mouth disease virus. Can quasispecies define a transient molecular
clock? Gene 103, 147-153.
346. von Haeseler, A. and Strimmer, K. (2004). Phylogeny interference
based on maximum-likelihood methods with TREE-PUZZLE. In: The
phylogenetic handbook: a practical approach to DNA and protein
phylogeny, pp. 127-159. Edited by Salemi, M. and Vandamme, A. M.
Cambridge, UK: Cambridge University Press.
347. Wakeley, J. (1993). Substitution rate variation among sites in
hypervariable region 1 of human mitochondrial DNA. J Mol Evol
37, 613-623.
348. Wall, J. D. and Kim, S.K. (2007). Inconsistencies in Neanderthal
genomic DNA sequences. PLoS Genet 3, 1862-1866.
349. Wallace, I.M., O’Sullivan, O., and Higgins, D.G. (2005). Evaluation of
iterative alignment algorithms for multiple alignment. Bioinformatics
21, 1408-1414.
252 Литература
350. Wallace, I.M., O’Sullivan, О., Higgins, D.G., and Notredame, C.
(2006). М-Coffee: combining multiple sequence alignment methods
with Т-Coffee. Nucleic Acids Res 34, 1692-1699.
351. Wang, C. and Lefkowitz, E.J. (2005). Genomic multiple
sequence alignments: refinement using a genetic algorithm. BMC
Bioinformatics 6, 200.
352. Waterman, M.S. (1983). Sequence alignments in the neighborhood of
the optimum with general application to dynamic programming. Proc
Natl Acad Sci USA 80, 3123-3124.
353. Waterman, M.S. (1984). Efficient sequence alignment algorithms.
J Theor Biol 108, 333-337.
354. Wei, X., Ghosh, S.K., Taylor, M.E., Johnson, V.A., Emini, E.A.,
Deutsch, P., Lifson, J. D., Bonhoeffer, S., Nowak, M.A., Hahn, В. H.,
Saag, M.S., and Shaw, G.M. (1995). Viral dynamics in human
immunodeficiency virus type 1 infection. Nature 373, 117-122.
355. Whelan, S. and Goldman, N. (2001). A general empirical model
of protein evolution derived from multiple protein families using a
maximum-likelihood approach. Mol Biol Evol 18, 691-699.
356. Wilgenbusch, J.C. and Swofford, D. (2003). Inferring evolutionary
trees with PAUP*. Curr Protoc Bioinformatics, Chapter 6, Unit 6.4.
357. Wilm, A., Higgins, D.G., and Notredame, C. (2008). R-Coffee: a
method for multiple alignment of non-coding RNA. Nucleic Acids
Res 36, e52.
358. Wilson, A.C. and Cann, R. L. (1992). The recent African genesis of
humans. Sci Am 266, 68-73.
359. Wright, F. (1990). The «effective number of codons» used in a gene.
Gene 87, 23-29.
360. Wright, S. (1931). Evolution in Mendelian populations. Genetics 16,
97-159.
361. Wright, S. (1938). The distribution of gene frequencies under
irreversible mutation. Proc Natl Acad Sci USA 24, 253-259.
362. Wu, C.I. and Li, W. H. (1985). Evidence for higher rates of nucleotide
substitution in rodents than in man. Proc Natl Acad Sci USA 82,
1741-1745.
363. Yang, Z. (1997). PAML: a program package for phylogenetic analysis
by maximum likelihood. Comput Appl Biosci 13, 555 556.
364. Yang, Z. (2007). PAML 4: phylogenetic analysis by maximum
likelihood. Mol Biol Evol 24, 1586-1591.
365. Yokoyama, S., Chung, L., and Gojobori, T. (1988). Molecular evolution
of the human immunodeficiency and related viruses. Mol Biol Evol
5, 237-251.
366. Zhang, J. and Madden, T.L. (1997). PowerBLAST: a new network
BLAST application for interactive or automated sequence analysis
and annotation. Genome Res 7, 649-656.
Литература 253
367. Zhang, X. and Kahveci, T. (2007). QOMA: quasi-optimal multiple
alignment of protein sequences. Bioinformatics 23, 162-168.
368. Zhang, Z., Lin, H., and Li, M. (2007). MANGO: a new approach to
multiple sequence alignment. Comput Syst Bioinformatics Conf 6,
237-247.
369. Zhang, Z., Schwartz, S., Wagner, L., and Miller, W. (2000). A greedy
algorithm for aligning DNA sequences. J Comput Biol 7, 203-214.
370. Zharkikh, A. (1994). Estimation of evolutionary distances between
nucleotide sequences. J Mol Evol 39, 315-329.
371. Zharkikh, A. and Li, W.H. (1992a). Statistical properties of bootstrap
estimation of phylogenetic variability from nucleotide sequences. I.
Four taxa with a molecular clock. Mol Biol Evol 9, 1119-1147.
372. Zharkikh, A. and Li, W.H. (1992b). Statistical properties of bootstrap
estimation of phylogenetic variability from nucleotide sequences: II.
Four taxa without a molecular clock. J Mol Evol 35, 356-366.
373. Zharkikh, A. and Li, W.H. (1995). Estimation of confidence
in phylogeny: the complete-and-partial bootstrap technique. Mol
Phylogenet Evol 4, 44-63.
374. Zhou, H. and Zhou, Y. (2005). SPEM: improving multiple sequence
alignment with sequence profiles and predicted secondary structures.
Bioinformatics 21, 3615-3621.
375. Zhu, T., Korber, B.T., Nahmias, A. J., Hooper, E., Sharp, P.M.,
and Ho, D. D. (1998). An African HIV-1 sequence from 1959 and
implications for the origin of the epidemic. Nature 391, 594-597.
376. Zsiros, J., Jebbink, M.F., Lukashov, V. V., Voute, P.A., and Berkhout,
B. (1999). Biased nucleotide composition of the genome of HERV-K
related endogenous retroviruses and its evolutionary implications. J
Mol Evol 48, 102-111.
377. Zuckerkandl, E. and Pauling, L. (1962). Molecular disease, evolution
and genic heterogeneity. In: Horizons in biochemistry, pp. 189-225.
Edited by Kasha, M. and Pullman, B. New York, USA: Academic
Press.
378. Zuckerkandl, E. and Pauling, L. (1965). Evolutionary divergence and
convergence in proteins. In: Evolving genes and proteins, pp. 97-166.
Edited by Bryson, V. and Vogel, H.J. New York, USA: Academic
Press.
ОГЛАВЛЕНИЕ
Предисловие.................................................. 5
Глава 1. Цели, принципы и понятия
молекулярной эволюции ...................................... 9
1.1. Задачи молекулярной эволюции как науки.............. 9
1.2. Нуклеотидные последовательности..................... 9
1.3. Аминокислотные последовательности.................. 11
1.4. Генетический код................................... 14
1.5. Мутации............................................ 16
1.6. Нуклеотидные замены................................ 17
1.6.1. Транзиции и трансверсии...................... 17
1.6.2. Синонимичные и несинонимичные замены....... 17
1.7. Нуклеотидный и аминокислотный состав,
использование кодонов................................. 19
1.8. Эволюция нуклеотидной последовательности........... 20
1.9. Консенсусные последовательности.................... 26
1.10. Гомологичные и сходные признаки, конвергенция... 29
1.11. Естественный отбор и неодарвинизм................. 30
1.12. Закрепление мутации в популяции................... 31
1.13. Концепция молекулярных часов...................... 35
1.14. Нейтральная теория молекулярной эволюции.......... 36
1.15. Эволюционная систематика ......................... 37
1.16. Проведение эволюционного анализа.................. 39
Глава 2. Выравнивание генетических
последовательностей........................................ 41
2.1. Цели выравнивания последовательностей.............. 41
2.2. Принципы выравнивания последовательностей.......... 41
2.3. Алгоритмы выравнивания двух последовательностей ... 46
2.3.1. Принцип матрицы точек........................ 46
2.3.2. Алгоритмы Нидлмена—Вунша и Смита—Уотерме-
на, глобальное и локальное выравнивание........... 49
2.3.3. Общие принципы динамического программирова-
ния при выравнивании последовательностей.......... 54
2.3.4. Методы слов.................................. 58
2.4. Множественное выравнивание......................... 59
Оглавление 255
Глава 3. Генетические дистанции и эволюционные
модели.................................................... 61
3.1. Наблюдаемые, истинные и расчетные дистанции..... 61
3.2. Эволюционные модели и дистанции между
нуклеотидными последовательностями................... 65
3.2.1. Модель Джукса—Кантора...................... 65
3.2.2. Модель Кимуры.............................. 71
3.2.3. Модель Таджимы—Неи......................... 73
3.2.4. Другие эволюционные модели................. 74
3.2.5. Гамма-дистанции............................ 76
3.2.6. Сравнение различных моделей................ 79
3.3. Синонимичные и несинонимичные дистанции
и их отношение....................................... 81
3.4. Аминокислотные дистанции, матрицы вероятностей ами-
нокислотных замещений................................ 85
3.5. Учет делеций и отсутствующей информации.......... 88
Глава 4. Филогенетический анализ.......................... 90
4.1. Филогенетические деревья......................... 90
4.2. Дистанционные методы построения
филогенетических деревьев ........................... 98
4.2.1. Принципы дистанционных методов............. 98
4.2.2. Метод UPGMA................................ 99
4.2.3. Метод трансформированной дистанции........ 104
4.2.4. Метод минимума эволюции................... 107
4.2.5. Методы связей между соседями.............. 107
4.2.6. Метод присоединения соседей............... 109
4.2.7. Установление длин ветвей.................. 111
4.3. Методы анализа дискретных признаков............. 114
4.3.1. Принципы методов анализа дискретных
признаков........................................ 114
4.3.2. Метод максимальной экономии............... 115
4.3.3. Метод максимального правдоподобия......... 123
4.4. Статистическая оценка дерева, бутстрэп-анализ... 126
4.5. Другие филогенетические методы.................. 131
4.6. Сравнение филогенетических методов ............. 133
4.7. Филогенетический анализ в таксономии, фенетика
и кладистика........................................ 136
Глава 5. Отдельные задачи эволюционного анализа.......... 141
5.1. Рекомбинационный анализ......................... 141
5.2. Анализ нуклеотидного и аминокислотного состава
и использования кодонов............................. 150
5.2.1. Смещения нуклеотидного состава............ 150
5.2.2. Различия в использовании кодонов.......... 156
256 Оглавление
5.2.3. Анализ нуклеотидного состава и использования
кодонов в филогенетических исследованиях.... 160
5.3. Анализ молекулярных часов...................... 168
5.3.1. Вопросы, связанные с концепцией
молекулярных часов............................... 168
5.3.2. Установление и калибровка молекулярных часов —
тест относительных скоростей эволюции............ 169
5.3.3. Другие подходы к установлению молекулярных
часов............................................ 172
5.3.4. Проблемы филогенетического анализа, связанные
с несоблюдением модели молекулярных часов .... 173
5.3.5. Анализ молекулярных часов в эволюции
высших организмов................................ 174
5.3.6. Анализ молекулярных часов в эволюции
вирусов.......................................... 176
5.3.7. Молекулярные часы в эволюции ВИЧ-1 и их ис-
пользование для датирования начала эпидемий
и эволюционных событий........................... 177
5.4. Анализ митохондриальной ДНК.................... 185
5.4.1. Особенности эволюционного анализа митохондри-
альной ДНК....................................... 185
5.4.2. Концепция митохондриальной Евы и использова-
ние анализа мтДНК при изучении происхождения
человека ........................................ 188
5.5. Молекулярная эпидемиология..................... 193
5.5.1. Задачи и принципы молекулярной
эпидемиологии.................................... 193
5.5.2. Установление источника заражения......... 195
5.5.3. Анализ эпидемиологических сетей.......... 195
Глава 6. Компьютерные программы для эволюционного
анализа................................................. 208
6.1. Типы компьютерных программ..................... 208
6.2. Программы для хранения и редактирования
последовательностей................................. 213
6.3. Международные базы генетических данных......... 215
6.4. Программы для выравнивания последовательностей .... 218
6.5. Программы для филогенетического анализа........ 221
6.6. Другие программы............................... 225
Книги для дополнительного чтения........................ 226
Литература.............................................. 228
Владимир Владимирович Лукашое
Окончил отделение биофизики медико-биологи-
ческого факультета 2-го Московского государствен-
ного медицинского института им. Н. И. Пирогова
(ныне - Российский государственный медицинский
университет) и аспирантуру при НИИ вирусологии
им. Д. И. Ивановского, Москва.
Доктор медицинских наук, старший научный сотрудник НИИ вирусоло-
гии им. Д. И. Ивановского, доцент Университета Амстердама (Нидер-
ланды). Автор более ста научных работ, опубликованных в веду-
щих отечественных и зарубежных журналах.